+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nucleocapsid monomer structure from SARS-CoV-2 | ||||||||||||
 マップデータ マップデータ | Nucleocapsid monomer structure from SARS-CoV-2 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | SARS-CoV-2 / N protein / COVID-19 / RNA binding protein / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoplasmic capsid assembly / response to host immune response / viral RNA genome packaging / negative regulation of interferon-beta production / Maturation of nucleoprotein / intracellular membraneless organelle / positive regulation of NLRP3 inflammasome complex assembly / MHC class I protein binding / CD28 dependent PI3K/Akt signaling / SARS-CoV-2 targets host intracellular signalling and regulatory pathways ...cytoplasmic capsid assembly / response to host immune response / viral RNA genome packaging / negative regulation of interferon-beta production / Maturation of nucleoprotein / intracellular membraneless organelle / positive regulation of NLRP3 inflammasome complex assembly / MHC class I protein binding / CD28 dependent PI3K/Akt signaling / SARS-CoV-2 targets host intracellular signalling and regulatory pathways / protein sequestering activity / VEGFR2 mediated vascular permeability / molecular condensate scaffold activity / NOD1/2 Signaling Pathway / TAK1-dependent IKK and NF-kappa-B activation / DDX58/IFIH1-mediated induction of interferon-alpha/beta / MHC class I protein complex / Interleukin-1 signaling / RNA stem-loop binding / Interferon alpha/beta signaling / PIP3 activates AKT signaling / viral capsid / Transcription of SARS-CoV-2 sgRNAs / host cell endoplasmic reticulum-Golgi intermediate compartment / viral nucleocapsid / host cell Golgi apparatus / Translation of Structural Proteins / Virion Assembly and Release / host extracellular space / Induction of Cell-Cell Fusion / Attachment and Entry / host cell perinuclear region of cytoplasm / ribonucleoprotein complex / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / RNA binding / extracellular region / identical protein binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
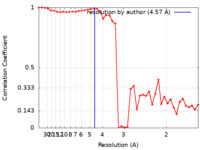
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.57 Å | ||||||||||||
 データ登録者 データ登録者 | Casasanta M / Jonaid GM / Kaylor L / Luqiu W / DiCecco L / Solares M / Berry S / Kelly DF | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 | ジャーナル: Nanoscale / 年: 2021 タイトル: Microchip-based structure determination of low-molecular weight proteins using cryo-electron microscopy. 著者: Michael A Casasanta / G M Jonaid / Liam Kaylor / William Y Luqiu / Maria J Solares / Mariah L Schroen / William J Dearnaley / Jarad Wilson / Madeline J Dukes / Deborah F Kelly /  要旨: Interest in cryo-Electron Microscopy (EM) imaging has skyrocketed in recent years due to its pristine views of macromolecules and materials. As advances in instrumentation and computing algorithms ...Interest in cryo-Electron Microscopy (EM) imaging has skyrocketed in recent years due to its pristine views of macromolecules and materials. As advances in instrumentation and computing algorithms spurred this progress, there is renewed focus to address specimen-related challenges. Here we contribute a microchip-based toolkit to perform complementary structural and biochemical analysis on low-molecular weight proteins. As a model system, we used the SARS-CoV-2 nucleocapsid (N) protein (48 kDa) due to its stability and important role in therapeutic development. Cryo-EM structures of the N protein monomer revealed a flexible N-terminal "top hat" motif and a helical-rich C-terminal domain. To complement our structural findings, we engineered microchip-based immunoprecipitation assays that led to the discovery of the first antibody binding site on the N protein. The data also facilitated molecular modeling of a variety of pandemic and common cold-related coronavirus proteins. Such insights may guide future pandemic-preparedness protocols through immuno-engineering strategies to mitigate viral outbreaks. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29002.map.gz emd_29002.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29002-v30.xml emd-29002-v30.xml emd-29002.xml emd-29002.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29002_fsc.xml emd_29002_fsc.xml | 4.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29002.png emd_29002.png | 187.7 KB | ||
| Filedesc metadata |  emd-29002.cif.gz emd-29002.cif.gz | 6.4 KB | ||
| その他 |  emd_29002_half_map_1.map.gz emd_29002_half_map_1.map.gz emd_29002_half_map_2.map.gz emd_29002_half_map_2.map.gz | 3.5 MB 3.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29002 http://ftp.pdbj.org/pub/emdb/structures/EMD-29002 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29002 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29002 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29002_validation.pdf.gz emd_29002_validation.pdf.gz | 690.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29002_full_validation.pdf.gz emd_29002_full_validation.pdf.gz | 690 KB | 表示 | |
| XML形式データ |  emd_29002_validation.xml.gz emd_29002_validation.xml.gz | 8.7 KB | 表示 | |
| CIF形式データ |  emd_29002_validation.cif.gz emd_29002_validation.cif.gz | 11.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29002 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29002 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29002 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29002 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fd5MC  8fg2C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29002.map.gz / 形式: CCP4 / 大きさ: 4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29002.map.gz / 形式: CCP4 / 大きさ: 4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nucleocapsid monomer structure from SARS-CoV-2 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_29002_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
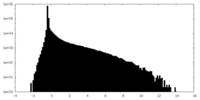
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_29002_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A chain contains the full-length N protein
| 全体 | 名称: A chain contains the full-length N protein |
|---|---|
| 要素 |
|
-超分子 #1: A chain contains the full-length N protein
| 超分子 | 名称: A chain contains the full-length N protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Residues 1-49 were fit into the map separately from residues 50 - 419. The 2 fragments were fit as individual rigid bodies, then refined and rebuilt. There is a missing peptide linkage between residues 49 and 50. |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.689645 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDNGPQNQR NAPRITFGGP SDSTGSNQNG ERSGARSKQR RPQGLPNNTA SWFTALTQHG KEDLKFPRGQ GVPINTNSSP DDQIGYYRR ATRRIRGGDG KMKDLSPRWY FYYLGTGPEA GLPYGANKDG IIWVATEGAL NTPKDHIGTR NPANNAAIVL Q LPQGTTLP ...文字列: MSDNGPQNQR NAPRITFGGP SDSTGSNQNG ERSGARSKQR RPQGLPNNTA SWFTALTQHG KEDLKFPRGQ GVPINTNSSP DDQIGYYRR ATRRIRGGDG KMKDLSPRWY FYYLGTGPEA GLPYGANKDG IIWVATEGAL NTPKDHIGTR NPANNAAIVL Q LPQGTTLP KGFYAEGSRG GSQASSRSSS RSRNSSRNST PGSSRGTSPA RMAGNGGDAA LALLLLDRLN QLESKMSGKG QQ QQGQTVT KKSAAEASKK PRQKRTATKA YNVTQAFGRR GPEQTQGNFG DQELIRQGTD YKHWPQIAQF APSASAFFGM SRI GMEVTP SGTWLTYTGA IKLDDKDPNF KDQVILLNKH IDAYKTFPPT EPKKDKKKKA DETQALPQRQ KKQQTVTLLP AADL DDFSK QLQQSMSSAD STQA UniProtKB: Nucleoprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris (pH 7.5), 150 mM NaCl, 10 mM MgCl2, 10 mM CaCl2 |
| グリッド | モデル: Homemade / 材質: SILICON NITRIDE 詳細: Protein samples were added to the Ni-NTA-coated microchips and incubated for 1 minute at room temperature prior to vitrification. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK III 詳細: A Mark III Vitrobot was used to plunge samples into liquid ethane, operating at room temperature and 100% humidity with 3 - 4 seconds blot time. |
| 詳細 | Sample was enriched using Ni-NTA coated silicon nitride microchips |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS F200C |
|---|---|
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-12 (4k x 3k) 検出モード: INTEGRATING / 撮影したグリッド数: 10 / 実像数: 200 / 平均露光時間: 1.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 100 |
|---|---|
| 得られたモデル |  PDB-8fd5: |
 ムービー
ムービー コントローラー
コントローラー









 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)