+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
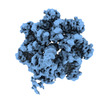
| タイトル | Cryo-EM structure of TnsC-TniQ-DNA complex in type I-B CAST system | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TnsC / TniQ / CRISPR / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 | TniQ / TniQ / : / AAA domain / ATP hydrolysis activity / P-loop containing nucleoside triphosphate hydrolase / ORC1/DEAH AAA+ ATPase domain-containing protein / TniQ family protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Peltigera membranacea (ウスバイヌツメゴケ) / synthetic construct (人工物) / Peltigera membranacea (ウスバイヌツメゴケ) / synthetic construct (人工物) /  Nostoc sp. 'Peltigera membranacea cyanobiont' 210A (バクテリア) Nostoc sp. 'Peltigera membranacea cyanobiont' 210A (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.95 Å | |||||||||
 データ登録者 データ登録者 | Chang L / Wang S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Molecular mechanism for Tn7-like transposon recruitment by a type I-B CRISPR effector. 著者: Shukun Wang / Clinton Gabel / Romana Siddique / Thomas Klose / Leifu Chang /  要旨: Tn7-like transposons have co-opted CRISPR-Cas systems to facilitate the movement of their own DNA. These CRISPR-associated transposons (CASTs) are promising tools for programmable gene knockin. A key ...Tn7-like transposons have co-opted CRISPR-Cas systems to facilitate the movement of their own DNA. These CRISPR-associated transposons (CASTs) are promising tools for programmable gene knockin. A key feature of CASTs is their ability to recruit Tn7-like transposons to nuclease-deficient CRISPR effectors. However, how Tn7-like transposons are recruited by diverse CRISPR effectors remains poorly understood. Here, we present the cryo-EM structure of a recruitment complex comprising the Cascade complex, TniQ, TnsC, and the target DNA in the type I-B CAST from Peltigera membranacea cyanobiont 210A. Target DNA recognition by Cascade induces conformational changes in Cas6 and primes TniQ recruitment through its C-terminal domain. The N-terminal domain of TniQ is bound to the seam region of the TnsC spiral heptamer. Our findings provide insights into the diverse mechanisms for the recruitment of Tn7-like transposons to CRISPR effectors and will aid in the development of CASTs as gene knockin tools. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28994.map.gz emd_28994.map.gz | 30.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28994-v30.xml emd-28994-v30.xml emd-28994.xml emd-28994.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28994.png emd_28994.png | 122.2 KB | ||
| Filedesc metadata |  emd-28994.cif.gz emd-28994.cif.gz | 5.9 KB | ||
| その他 |  emd_28994_half_map_1.map.gz emd_28994_half_map_1.map.gz emd_28994_half_map_2.map.gz emd_28994_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28994 http://ftp.pdbj.org/pub/emdb/structures/EMD-28994 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28994 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28994 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fcvMC  8fcjC  8fcuC  8fcwC  8fcxC  8fd2C  8fd3C  8ff4C  8ff5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28994.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28994.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.054 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28994_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28994_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of TnsC, TniQ and DNA in type I-B CAST system
| 全体 | 名称: Ternary complex of TnsC, TniQ and DNA in type I-B CAST system |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of TnsC, TniQ and DNA in type I-B CAST system
| 超分子 | 名称: Ternary complex of TnsC, TniQ and DNA in type I-B CAST system タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Peltigera membranacea (ウスバイヌツメゴケ) Peltigera membranacea (ウスバイヌツメゴケ) |
-分子 #1: DNA (60-MER)
| 分子 | 名称: DNA (60-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 18.500861 KDa |
| 配列 | 文字列: (DA)(DA)(DG)(DA)(DC)(DG)(DT)(DA)(DG)(DC) (DC)(DC)(DA)(DG)(DC)(DG)(DC)(DG)(DT)(DC) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DA)(DC) (DG)(DT)(DA)(DT)(DC)(DG)(DT)(DA)(DG)(DA) (DT) (DA)(DT)(DA)(DT)(DC) ...文字列: (DA)(DA)(DG)(DA)(DC)(DG)(DT)(DA)(DG)(DC) (DC)(DC)(DA)(DG)(DC)(DG)(DC)(DG)(DT)(DC) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DA)(DC) (DG)(DT)(DA)(DT)(DC)(DG)(DT)(DA)(DG)(DA) (DT) (DA)(DT)(DA)(DT)(DC)(DT)(DA)(DC) (DG)(DC)(DG)(DT)(DA)(DG)(DA)(DT)(DA)(DT) (DA)(DT) |
-分子 #2: DNA (60-MER)
| 分子 | 名称: DNA (60-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 18.482832 KDa |
| 配列 | 文字列: (DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT)(DA)(DC) (DG)(DC)(DG)(DT)(DA)(DG)(DA)(DT)(DA)(DT) (DA)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DT) (DA)(DC)(DG)(DT)(DA)(DG)(DC)(DG)(DG)(DC) (DC) (DG)(DA)(DC)(DG)(DC) ...文字列: (DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT)(DA)(DC) (DG)(DC)(DG)(DT)(DA)(DG)(DA)(DT)(DA)(DT) (DA)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DT) (DA)(DC)(DG)(DT)(DA)(DG)(DC)(DG)(DG)(DC) (DC) (DG)(DA)(DC)(DG)(DC)(DG)(DC)(DT) (DG)(DG)(DG)(DC)(DT)(DA)(DC)(DG)(DT)(DC) (DT)(DT) |
-分子 #3: TniQ
| 分子 | 名称: TniQ / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Nostoc sp. 'Peltigera membranacea cyanobiont' 210A (バクテリア) Nostoc sp. 'Peltigera membranacea cyanobiont' 210A (バクテリア) |
| 分子量 | 理論値: 37.591961 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTIYSEHWSL NPLEIPQRSR LFSLEPVAVG TPYAESLSSY LHRLAQAHCL TSEKLVMGEI APLILKDEDK SELLSKNLSH LLGNSDAKP AINGMREMTE KLVTVLEELT MRQDLRFLTL LSWKGMIYDK GLFRNYRAWC PCCCEEWMQK NKTIYEPLSW S FKDVEFCL ...文字列: MTIYSEHWSL NPLEIPQRSR LFSLEPVAVG TPYAESLSSY LHRLAQAHCL TSEKLVMGEI APLILKDEDK SELLSKNLSH LLGNSDAKP AINGMREMTE KLVTVLEELT MRQDLRFLTL LSWKGMIYDK GLFRNYRAWC PCCCEEWMQK NKTIYEPLSW S FKDVEFCL IHKQRLIEEC SHCGARLPVM ARLSPAGFCS RCYGWLGQEI KGEEEIEKYR VNIQGISELI ALTPQLGYKP IP IELTRKL QLILLVFEQA IGKDVKLLGD LGGIMESLRI ASTTNQSQPY HLVKLIIPVC EKAKISVFQL FGSDFKELGK ILF GNFSLE LKL UniProtKB: TniQ family protein |
-分子 #4: TnsC
| 分子 | 名称: TnsC / タイプ: protein_or_peptide / ID: 4 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Nostoc sp. 'Peltigera membranacea cyanobiont' 210A (バクテリア) Nostoc sp. 'Peltigera membranacea cyanobiont' 210A (バクテリア) |
| 分子量 | 理論値: 43.521934 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTKSTGFPLE LLTRPATERL AYFENYTVAH PRLKEVYEIL MRTIAEPAGA SFIFVYGASG VGKTTLRLRV EQKLTELALP KLESDRARV PVVGIEAIAP ESRYFNWKEY YTRALITLEE PLIDHKFDYG VRGISRDNFG KINVESKVVA PALRRALENA L IHRHPDVF ...文字列: MTKSTGFPLE LLTRPATERL AYFENYTVAH PRLKEVYEIL MRTIAEPAGA SFIFVYGASG VGKTTLRLRV EQKLTELALP KLESDRARV PVVGIEAIAP ESRYFNWKEY YTRALITLEE PLIDHKFDYG VRGISRDNFG KINVESKVVA PALRRALENA L IHRHPDVF FVDEAQHFGK VASGYKLQDQ LDCLKSLANM TGILHCLLGT YELLTFRNLS GQLSRRSVDI HFRRYCADSP ED VQAFKSV LLTFQQHLPL AETPNLVDHW EYFYERTLGC IGTLKDWLKR VLSDALDREA TTITLKDLQK RALSVAQCQK MFK EIQEGE RQLSETEADV QNLRSALGLG AKPIVLPEET PKTTRPPGKV GKRKPKRDPI GVQQDVS UniProtKB: ORC1/DEAH AAA+ ATPase domain-containing protein |
-分子 #5: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 7 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 7 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.95 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 185087 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)