+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of designed modular protein oligomer C4-131 | |||||||||
 マップデータ マップデータ | designed modular protein oligomer C4-131 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Synthetic / Self-assembling / Oligomeric / Helical repeats / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.1 Å | |||||||||
 データ登録者 データ登録者 | Redler RL / Edman NI / Baker D / Ekiert D / Bhabha G | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2023 タイトル: Modulation of FGF pathway signaling and vascular differentiation using designed oligomeric assemblies. 著者: Natasha I Edman / Rachel L Redler / Ashish Phal / Thomas Schlichthaerle / Sanjay R Srivatsan / Ali Etemadi / Seong J An / Andrew Favor / Devon Ehnes / Zhe Li / Florian Praetorius / Max Gordon ...著者: Natasha I Edman / Rachel L Redler / Ashish Phal / Thomas Schlichthaerle / Sanjay R Srivatsan / Ali Etemadi / Seong J An / Andrew Favor / Devon Ehnes / Zhe Li / Florian Praetorius / Max Gordon / Wei Yang / Brian Coventry / Derrick R Hicks / Longxing Cao / Neville Bethel / Piper Heine / Analisa Murray / Stacey Gerben / Lauren Carter / Marcos Miranda / Babak Negahdari / Sangwon Lee / Cole Trapnell / Lance Stewart / Damian C Ekiert / Joseph Schlessinger / Jay Shendure / Gira Bhabha / Hannele Ruohola-Baker / David Baker /   要旨: Growth factors and cytokines signal by binding to the extracellular domains of their receptors and drive association and transphosphorylation of the receptor intracellular tyrosine kinase domains, ...Growth factors and cytokines signal by binding to the extracellular domains of their receptors and drive association and transphosphorylation of the receptor intracellular tyrosine kinase domains, initiating downstream signaling cascades. To enable systematic exploration of how receptor valency and geometry affects signaling outcomes, we designed cyclic homo-oligomers with up to 8 subunits using repeat protein building blocks that can be modularly extended. By incorporating a designed fibroblast growth-factor receptor (FGFR) binding module into these scaffolds, we generated a series of synthetic signaling ligands that exhibit potent valency- and geometry-dependent Ca2+ release and MAPK pathway activation. The high specificity of the designed agonists reveal distinct roles for two FGFR splice variants in driving endothelial and mesenchymal cell fates during early vascular development. The ability to incorporate receptor binding domains and repeat extensions in a modular fashion makes our designed scaffolds broadly useful for probing and manipulating cellular signaling pathways. #2:  ジャーナル: Cell(Cambridge,Mass.) / 年: 2024 ジャーナル: Cell(Cambridge,Mass.) / 年: 2024タイトル: Modulation of FGF pathway signaling and vascular differentiation using designed oligomeric assemblies 著者: Edman NI / Redler RL / Phal A / Schlichthaerle T / Srivatsan SR / Etemadi A / An SJ / Favor A / Ehnes D / Li Z / Praetorius F / Gordon M / Yang W / Coventry B / Hicks DR / Cao L / Bethel N / ...著者: Edman NI / Redler RL / Phal A / Schlichthaerle T / Srivatsan SR / Etemadi A / An SJ / Favor A / Ehnes D / Li Z / Praetorius F / Gordon M / Yang W / Coventry B / Hicks DR / Cao L / Bethel N / Heine P / Murray A / Gerben S / Carter L / Miranda M / Negahdari B / Lee S / Trapnell C / Stewart L / Ekiert DC / Schlessinger J / Shendure J / Bhabha G / Ruohola-Baker H / Baker D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28958.map.gz emd_28958.map.gz | 6.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28958-v30.xml emd-28958-v30.xml emd-28958.xml emd-28958.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28958.png emd_28958.png | 64.1 KB | ||
| Filedesc metadata |  emd-28958.cif.gz emd-28958.cif.gz | 5.9 KB | ||
| その他 |  emd_28958_half_map_1.map.gz emd_28958_half_map_1.map.gz emd_28958_half_map_2.map.gz emd_28958_half_map_2.map.gz | 11.9 MB 11.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28958 http://ftp.pdbj.org/pub/emdb/structures/EMD-28958 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28958 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28958 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28958_validation.pdf.gz emd_28958_validation.pdf.gz | 976.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28958_full_validation.pdf.gz emd_28958_full_validation.pdf.gz | 976 KB | 表示 | |
| XML形式データ |  emd_28958_validation.xml.gz emd_28958_validation.xml.gz | 9.4 KB | 表示 | |
| CIF形式データ |  emd_28958_validation.cif.gz emd_28958_validation.cif.gz | 10.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28958 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28958 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28958 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28958 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28958.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28958.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | designed modular protein oligomer C4-131 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.096 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_28958_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
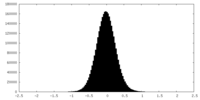
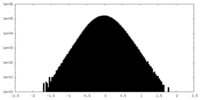
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_28958_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
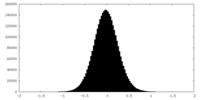
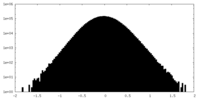
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Self-assembled homo-tetramer of de novo designed protein C4-131
| 全体 | 名称: Self-assembled homo-tetramer of de novo designed protein C4-131 |
|---|---|
| 要素 |
|
-超分子 #1: Self-assembled homo-tetramer of de novo designed protein C4-131
| 超分子 | 名称: Self-assembled homo-tetramer of de novo designed protein C4-131 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: C4-131
| 分子 | 名称: C4-131 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGLKELLKRA EELAKSPDPE DLKEAVRLAE EVVRERPGSE AAKKALEIIQ EAAEKLKKSP DPEAIIAAAR ALLKIAATTG DNEAAKQAIE AASKAAQLAE QRGDDELVCE ALALLIAAQV LLLKQQGVPM LEVAIHVAET ILQILQRLKR KGASEEVRKE CLKRILREIA ...文字列: MGLKELLKRA EELAKSPDPE DLKEAVRLAE EVVRERPGSE AAKKALEIIQ EAAEKLKKSP DPEAIIAAAR ALLKIAATTG DNEAAKQAIE AASKAAQLAE QRGDDELVCE ALALLIAAQV LLLKQQGVPM LEVAIHVAET ILQILQRLKR KGASEEVRKE CLKRILREIA EALQRSGVPE EEIALIMLLI ILLLMMLGSL EHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.75 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: HOLEY ARRAY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: CARBON / 支持フィルム - #1 - トポロジー: CONTINUOUS / 支持フィルム - #1 - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 5 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: blot time = 4s; blot force = 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 26 / 平均露光時間: 2.8 sec. / 平均電子線量: 56.91 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.9 µm / 最小 デフォーカス(公称値): 2.1 µm / 倍率(公称値): 36000 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE / 詳細: Ab initio model generated in Cryosparc |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 9.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3) / 使用した粒子像数: 13986 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3) |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)