+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Asymmetric structure of cleaved HIV-1 AD8 envelope glycoprotein trimer in styrene-maleic acid lipid nanoparticles | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 / envelope glycoprotein / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Dectin-2 family / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell ...Dectin-2 family / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Wang K / Zhang S / Sodroski J / Mao Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Asymmetric conformations of cleaved HIV-1 envelope glycoprotein trimers in styrene-maleic acid lipid nanoparticles. 著者: Kunyu Wang / Shijian Zhang / Eden P Go / Haitao Ding / Wei Li Wang / Hanh T Nguyen / John C Kappes / Heather Desaire / Joseph Sodroski / Youdong Mao /   要旨: During virus entry, the pretriggered human immunodeficiency virus (HIV-1) envelope glycoprotein (Env) trimer initially transits into a default intermediate state (DIS) that remains structurally ...During virus entry, the pretriggered human immunodeficiency virus (HIV-1) envelope glycoprotein (Env) trimer initially transits into a default intermediate state (DIS) that remains structurally uncharacterized. Here, we present cryo-EM structures at near-atomic resolution of two cleaved full-length HIV-1 Env trimers purified from cell membranes in styrene-maleic acid lipid nanoparticles without antibodies or receptors. The cleaved Env trimers exhibited tighter subunit packing than uncleaved trimers. Cleaved and uncleaved Env trimers assumed remarkably consistent yet distinct asymmetric conformations, with one smaller and two larger opening angles. Breaking conformational symmetry is allosterically coupled with dynamic helical transformations of the gp41 N-terminal heptad repeat (HR1) regions in two protomers and with trimer tilting in the membrane. The broken symmetry of the DIS potentially assists Env binding to two CD4 receptors-while resisting antibody binding-and promotes extension of the gp41 HR1 helical coiled-coil, which relocates the fusion peptide closer to the target cell membrane. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28953.map.gz emd_28953.map.gz | 16.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28953-v30.xml emd-28953-v30.xml emd-28953.xml emd-28953.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28953.png emd_28953.png | 124.2 KB | ||
| Filedesc metadata |  emd-28953.cif.gz emd-28953.cif.gz | 5.9 KB | ||
| その他 |  emd_28953_half_map_1.map.gz emd_28953_half_map_1.map.gz emd_28953_half_map_2.map.gz emd_28953_half_map_2.map.gz | 13.8 MB 13.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28953 http://ftp.pdbj.org/pub/emdb/structures/EMD-28953 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28953 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28953 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28953_validation.pdf.gz emd_28953_validation.pdf.gz | 894.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28953_full_validation.pdf.gz emd_28953_full_validation.pdf.gz | 894.4 KB | 表示 | |
| XML形式データ |  emd_28953_validation.xml.gz emd_28953_validation.xml.gz | 9.8 KB | 表示 | |
| CIF形式データ |  emd_28953_validation.cif.gz emd_28953_validation.cif.gz | 11.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28953 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28953 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28953 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28953 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28953.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28953.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.37 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28953_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
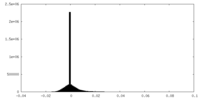
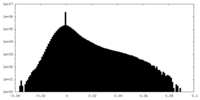
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28953_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
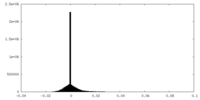
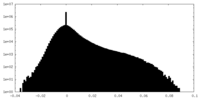
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Env AD8
| 全体 | 名称: HIV-1 Env AD8 |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 Env AD8
| 超分子 | 名称: HIV-1 Env AD8 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: Transmembrane protein gp41
| 分子 | 名称: Transmembrane protein gp41 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 15.674881 KDa |
| 組換発現 | 生物種:  homo sapiens (ヒト) homo sapiens (ヒト) |
| 配列 | 文字列: LGFLGAAGST MGAASITLTV QARLLLSGIV QQQNNLLRAI EAQQHLLQLT VWGIKQLQAR VLAVERYLRD QQLLGIWGCS GKLICTTAV PWNASWSNKT LDMIWNNMTW MEWEREIDNY TGLIYTLIEE SQNQQEKNE UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 52.779613 KDa |
| 組換発現 | 生物種:  homo sapiens (ヒト) homo sapiens (ヒト) |
| 配列 | 文字列: KLWVTVYYGV PVWKEATTTL FCASDAKAYD TEVHNVWATH ACVPTDPNPQ EVVLENVTEN FNMWKNNMVE QMHEDIISLW DQSLKPCVK LTPLCVTLNC TDLRNVTNIN NSSEGMRGEI KNCSFNITTS IRDKVKKDYA LFYRLDVVPI DNDNTSYRLI N CNTSTITQ ...文字列: KLWVTVYYGV PVWKEATTTL FCASDAKAYD TEVHNVWATH ACVPTDPNPQ EVVLENVTEN FNMWKNNMVE QMHEDIISLW DQSLKPCVK LTPLCVTLNC TDLRNVTNIN NSSEGMRGEI KNCSFNITTS IRDKVKKDYA LFYRLDVVPI DNDNTSYRLI N CNTSTITQ ACPKVSFEPI PIHYCTPAGF AILKCKDKKF NGTGPCKNVS TVQCTHGIRP VVSTQLLLNG SLAEEEVVIR SS NFTDNAK NIIVQLKESV EINCTRPNNN TRKSIHIGPG RAFYTTGDII GDIRQAHCNI SRTKWNNTLN QIATKLKEQF GNN KTIVFN QSSGGDPEIV MHSFNCGGEF FYCNSTQLFN STWNFNGTWN LTQSNGTEGN DTITLPCRIK QIINMWQEVG KAMY APPIR GQIRCSSNIT GLILTRDGGN NHNNDTETFR PGGGDMRDNW RSELYKYKVV KIEPLGVAPT KAKR UniProtKB: Envelope glycoprotein |
-分子 #10: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 10 / コピー数: 22 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #11: 1-[(2R)-4-(benzenecarbonyl)-2-methylpiperazin-1-yl]-2-(4-methoxy-...
| 分子 | 名称: 1-[(2R)-4-(benzenecarbonyl)-2-methylpiperazin-1-yl]-2-(4-methoxy-1H-pyrrolo[2,3-b]pyridin-3-yl)ethane-1,2-dione タイプ: ligand / ID: 11 / コピー数: 3 / 式: 83G |
|---|---|
| 分子量 | 理論値: 406.434 Da |
| Chemical component information | 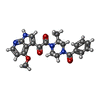 ChemComp-83G: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 365824 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)