+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Arabidopsis thaliana phytochrome A (focused map of platform) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Phytochrome / asymmetric dimer / gene regulation | |||||||||
| 生物種 |  | |||||||||
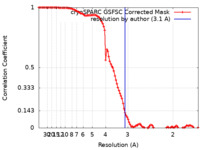
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2023 ジャーナル: Nat Plants / 年: 2023タイトル: The structure of Arabidopsis phytochrome A reveals topological and functional diversification among the plant photoreceptor isoforms. 著者: E Sethe Burgie / Hua Li / Zira T K Gannam / Katrice E McLoughlin / Richard D Vierstra / Huilin Li /  要旨: Plants employ a divergent cohort of phytochrome (Phy) photoreceptors to govern many aspects of morphogenesis through reversible photointerconversion between inactive Pr and active Pfr conformers. The ...Plants employ a divergent cohort of phytochrome (Phy) photoreceptors to govern many aspects of morphogenesis through reversible photointerconversion between inactive Pr and active Pfr conformers. The two most influential are PhyA whose retention of Pfr enables sensation of dim light, while the relative instability of Pfr for PhyB makes it better suited for detecting full sun and temperature. To better understand these contrasts, we solved, by cryo-electron microscopy, the three-dimensional structure of full-length PhyA as Pr. Like PhyB, PhyA dimerizes through head-to-head assembly of its C-terminal histidine kinase-related domains (HKRDs), while the remainder assembles as a head-to-tail light-responsive platform. Whereas the platform and HKRDs associate asymmetrically in PhyB dimers, these lopsided connections are absent in PhyA. Analysis of truncation and site-directed mutants revealed that this decoupling and altered platform assembly have functional consequences for Pfr stability of PhyA and highlights how plant Phy structural diversification has extended light and temperature perception. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28871.map.gz emd_28871.map.gz | 117.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28871-v30.xml emd-28871-v30.xml emd-28871.xml emd-28871.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28871_fsc.xml emd_28871_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28871.png emd_28871.png | 179.4 KB | ||
| その他 |  emd_28871_additional_1.map.gz emd_28871_additional_1.map.gz emd_28871_half_map_1.map.gz emd_28871_half_map_1.map.gz emd_28871_half_map_2.map.gz emd_28871_half_map_2.map.gz | 110.4 MB 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28871 http://ftp.pdbj.org/pub/emdb/structures/EMD-28871 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28871 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28871 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28871_validation.pdf.gz emd_28871_validation.pdf.gz | 846.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28871_full_validation.pdf.gz emd_28871_full_validation.pdf.gz | 846.2 KB | 表示 | |
| XML形式データ |  emd_28871_validation.xml.gz emd_28871_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_28871_validation.cif.gz emd_28871_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28871 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28871 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28871 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28871 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28871.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28871.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_28871_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
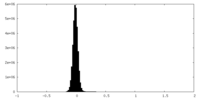
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28871_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28871_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : phytochrome A
| 全体 | 名称: phytochrome A |
|---|---|
| 要素 |
|
-超分子 #1: phytochrome A
| 超分子 | 名称: phytochrome A / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 249 KDa |
-分子 #1: Phytochrome A
| 分子 | 名称: Phytochrome A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: GMSGSRPTQS SEGSRRSRHS ARIIAQTTVD AKLHADFEES GSSFDYSTSV RVTGPVVENQ PPRSDKVTTT YLHHIQKGKL IQPFGCLLAL DEKTFKVIAY SENASELLTM ASHAVPSVGE HPVLGIGTDI RSLFTAPSAS ALQKALGFGD VSLLNPILVH CRTSAKPFYA ...文字列: GMSGSRPTQS SEGSRRSRHS ARIIAQTTVD AKLHADFEES GSSFDYSTSV RVTGPVVENQ PPRSDKVTTT YLHHIQKGKL IQPFGCLLAL DEKTFKVIAY SENASELLTM ASHAVPSVGE HPVLGIGTDI RSLFTAPSAS ALQKALGFGD VSLLNPILVH CRTSAKPFYA IIHRVTGSII IDFEPVKPYE VPMTAAGALQ SYKLAAKAIT RLQSLPSGSM ERLCDTMVQE VFELTGYDRV MAYKFHEDDH GEVVSEVTKP GLEPYLGLHY PATDIPQAAR FLFMKNKVRM IVDCNAKHAR VLQDEKLSFD LTLCGSTLRA PHSCHLQYMA NMDSIASLVM AVVVNEEDGE GDAPDATTQP QKRKRLWGLV VCHNTTPRFV PFPLRYACEF LAQVFAIHVN KEVELDNQMV EKNILRTQTL LCDMLMRDAP LGIVSQSPNI MDLVKCDGAA LLYKDKIWKL GTTPSEFHLQ EIASWLCEYH MDSTGLSTDS LHDAGFPRAL SLGDSVCGMA AVRISSKDMI FWFRSHTAGE VRWGGAKHDP DDRDDARRMH PRSSFKAFLE VVKTRSLPWK DYEMDAIHSL QLILRNAFKD SETTDVNTKV IYSKLNDLKI DGIQELEAVT SEMVRLIETA TVPILAVDSD GLVNGWNTKI AELTGLSVDE AIGKHFLTLV EDSSVEIVKR MLENALEGTE EQNVQFEIKT HLSRADAGPI SLVVNACASR DLHENVVGVC FVAHDLTGQK TVMDKFTRIE GDYKAIIQNP NPLIPPIFGT DEFGWCTEWN PAMSKLTGLK REEVIDKMLL GEVFGTQKSC CRLKNQEAFV NLGIVLNNAV TSQDPEKVSF AFFTRGGKYV ECLLCVSKKL DREGVVTGVF CFLQLASHEL QQALHVQRLA ERTAVKRLKA LAYIKRQIRN PLSGIMFTRK MIEGTELGPE QRRILQTSAL CQKQLSKILD DSDLESIIEG CLDLEMKEFT LNEVLTASTS QVMMKSNGKS VRITNETGEE VMSDTLYGDS IRLQQVLADF MLMAVNFTPS GGQLTVSASL RKDQLGRSVH LANLEIRLTH TGAGIPEFLL NQMFGTEEDV SEEGLSLMVS RKLVKLMNGD VQYLRQAGKS SFIITAELAA ANK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 299 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 193.0 K / 最高: 193.0 K |
| アライメント法 | Coma free - Residual tilt: 0.05 mrad |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 6390 / 平均露光時間: 1.0 sec. / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)