+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acylsulfonamide inhibitor GDC-0310 | |||||||||
 マップデータ マップデータ | Density modified map used for model refinements | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報action potential propagation / detection of mechanical stimulus involved in sensory perception / membrane depolarization during action potential / cardiac muscle cell action potential involved in contraction / node of Ranvier / voltage-gated sodium channel complex / Interaction between L1 and Ankyrins / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity / Phase 0 - rapid depolarisation ...action potential propagation / detection of mechanical stimulus involved in sensory perception / membrane depolarization during action potential / cardiac muscle cell action potential involved in contraction / node of Ranvier / voltage-gated sodium channel complex / Interaction between L1 and Ankyrins / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity / Phase 0 - rapid depolarisation / detection of temperature stimulus involved in sensory perception of pain / behavioral response to pain / neuronal action potential / axon terminus / sensory perception of pain / sodium ion transmembrane transport / post-embryonic development / circadian rhythm / response to toxic substance / Sensory perception of sweet, bitter, and umami (glutamate) taste / inflammatory response / axon / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Kschonsak M / Jao CC / Arthur CP / Rohou AL / Bergeron P / Ortwine D / McKerall SJ / Hackos DH / Deng L / Chen J ...Kschonsak M / Jao CC / Arthur CP / Rohou AL / Bergeron P / Ortwine D / McKerall SJ / Hackos DH / Deng L / Chen J / Sutherlin D / Dragovich PS / Volgraf M / Wright MR / Payandeh J / Ciferri C / Tellis JC | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Cryo-EM reveals an unprecedented binding site for Na1.7 inhibitors enabling rational design of potent hybrid inhibitors. 著者: Marc Kschonsak / Christine C Jao / Christopher P Arthur / Alexis L Rohou / Philippe Bergeron / Daniel F Ortwine / Steven J McKerrall / David H Hackos / Lunbin Deng / Jun Chen / Tianbo Li / ...著者: Marc Kschonsak / Christine C Jao / Christopher P Arthur / Alexis L Rohou / Philippe Bergeron / Daniel F Ortwine / Steven J McKerrall / David H Hackos / Lunbin Deng / Jun Chen / Tianbo Li / Peter S Dragovich / Matthew Volgraf / Matthew R Wright / Jian Payandeh / Claudio Ciferri / John C Tellis /  要旨: The voltage-gated sodium (Na) channel Na1.7 has been identified as a potential novel analgesic target due to its involvement in human pain syndromes. However, clinically available Na channel-blocking ...The voltage-gated sodium (Na) channel Na1.7 has been identified as a potential novel analgesic target due to its involvement in human pain syndromes. However, clinically available Na channel-blocking drugs are not selective among the nine Na channel subtypes, Na1.1-Na1.9. Moreover, the two currently known classes of Na1.7 subtype-selective inhibitors (aryl- and acylsulfonamides) have undesirable characteristics that may limit their development. To this point understanding of the structure-activity relationships of the acylsulfonamide class of Na1.7 inhibitors, exemplified by the clinical development candidate , has been based solely on a single co-crystal structure of an arylsulfonamide inhibitor bound to voltage-sensing domain 4 (VSD4). To advance inhibitor design targeting the Na1.7 channel, we pursued high-resolution ligand-bound Na1.7-VSD4 structures using cryogenic electron microscopy (cryo-EM). Here, we report that engages the Na1.7-VSD4 through an unexpected binding mode orthogonal to the arylsulfonamide inhibitor class binding pose, which identifies a previously unknown ligand binding site in Na channels. This finding enabled the design of a novel hybrid inhibitor series that bridges the aryl- and acylsulfonamide binding pockets and allows for the generation of molecules with substantially differentiated structures and properties. Overall, our study highlights the power of cryo-EM methods to pursue challenging drug targets using iterative and high-resolution structure-guided inhibitor design. This work also underscores an important role of the membrane bilayer in the optimization of selective Na channel modulators targeting VSD4. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28777.map.gz emd_28777.map.gz | 8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28777-v30.xml emd-28777-v30.xml emd-28777.xml emd-28777.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28777.png emd_28777.png | 79.1 KB | ||
| その他 |  emd_28777_additional_1.map.gz emd_28777_additional_1.map.gz emd_28777_half_map_1.map.gz emd_28777_half_map_1.map.gz emd_28777_half_map_2.map.gz emd_28777_half_map_2.map.gz | 226.1 MB 46.6 MB 46.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28777 http://ftp.pdbj.org/pub/emdb/structures/EMD-28777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28777 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28777_validation.pdf.gz emd_28777_validation.pdf.gz | 800.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28777_full_validation.pdf.gz emd_28777_full_validation.pdf.gz | 800.1 KB | 表示 | |
| XML形式データ |  emd_28777_validation.xml.gz emd_28777_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_28777_validation.cif.gz emd_28777_validation.cif.gz | 15.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28777 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28777 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28777 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28777 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28777.map.gz / 形式: CCP4 / 大きさ: 8.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28777.map.gz / 形式: CCP4 / 大きさ: 8.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modified map used for model refinements | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.838 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: non-modified, full map
| ファイル | emd_28777_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | non-modified, full map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
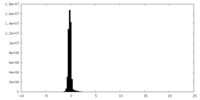
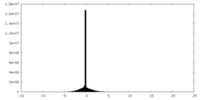
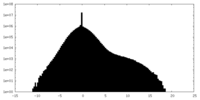
| 密度ヒストグラム |
-ハーフマップ: half-map1
| ファイル | emd_28777_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
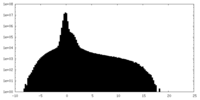
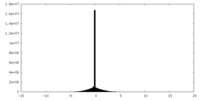
| 密度ヒストグラム |
-ハーフマップ: half-map2
| ファイル | emd_28777_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
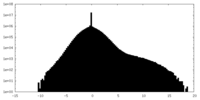
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acyl...
| 全体 | 名称: Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acylsulfonamide inhibitor GDC-0310 |
|---|---|
| 要素 |
|
-超分子 #1: Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acyl...
| 超分子 | 名称: Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acylsulfonamide inhibitor GDC-0310 タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 詳細: Chimeric construct of human Nav1.7 VSD4 and the NavPaS channel from American cockroach Periplaneta americana |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Sodium channel protein PaFPC1,Sodium channel protein type 9 subun...
| 分子 | 名称: Sodium channel protein PaFPC1,Sodium channel protein type 9 subunit alpha chimera タイプ: protein_or_peptide / ID: 1 詳細: Chimeric construct of human Nav1.7 VSD4 and the NavPaS channel from American cockroach Periplaneta americana コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 184.481906 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MWSHPQFEKG GGSGGGSGGS AWSHPQFEKG GSGGDYKDDD DKGGSGGDYK DDDDKMADNS PLIREERQRL FRPYTRAMLT APSAQPAKE NGKTEENKDN SRDKGRGANK DRDGSAHPDQ ALEQGSRLPA RMRNIFPAEL ASTPLEDFDP FYKNKKTFVV V TKAGDIFR ...文字列: MWSHPQFEKG GGSGGGSGGS AWSHPQFEKG GSGGDYKDDD DKGGSGGDYK DDDDKMADNS PLIREERQRL FRPYTRAMLT APSAQPAKE NGKTEENKDN SRDKGRGANK DRDGSAHPDQ ALEQGSRLPA RMRNIFPAEL ASTPLEDFDP FYKNKKTFVV V TKAGDIFR FSGEKSLWML DPFTPIRRVA ISTMVQPIFS YFIMITILIH CIFMIMPATQ TTYILELVFL SIYTIEVVVK VL ARGFILH PFAYLRDPWN WLDFLVTLIG YITLVVDLGH LYALRAFRVL RSWRTVTIVP GWRTIVDALS LSITSLKDLV LLL LFSLSV FALIGLQLFM GNLKHKCVKH FPADGSWGNF TDERWFNYTS NSSHWYIPDD WIEYPLCGNS SGAGMCPPGY TCLQ GYGGN PNYGYTSFDT FGWAFLSVFR LVTLDYWEDL YQLALRSAGP WHILFFIIVV FYGTFCFLNF ILAVVVMSYT HMVKR ADEE KAAERELKKE KKAASVANNT ANGQEQTTIE MNGDEAVVID NNDQAARQQS DPETPAPSVT QRLTDFLCVW DCCVPW QKL QGAIGAVVLS PFFELFIAVI IVLNITFMAL DHHDMNIEFE RILRTGNYIF TSIYIVEAVL KIIALSPKFY FKDSWNV FD FIIVVFAILE LGLEGVQGLS VFRSFRLLRV FRLAKFWPTL NNFMSVMTKS YGAFVNVMYV MFLLLFIFAI IGMQLFGM N YIDNMERFPD GDLPRWNFTD FLHSFMIVFR ALCGEWIESM WDCMLVGDWS CIPFFVAVFF VGNLVILNLL IALLLNNYG SFCTSPTSDE EDSKDEDALA QIVRIFKRFK PNLNAVKLSP MKPDSEDIVE SQEIQGNNIA DAEDVLAGEF PPDCCCNAFY KCFPSRPAR DSSVQRMWSN IRRVCFLLAK NKYFQKFVTA VLVITSVLLA LEDIYLPQRP VLVNITLYVD YVLTAFFVIE M IIMLFAVG FKKYFTSKWY WLDFIVVVAY LLNFVLMCAG IEALQTLRLL RVFRLFRPLS KVNGMQVVTS TLVEAVPHIF NV ILVGIFF WLVFAIMGVQ LFAGKFYKCV DENSTVLSHE ITMDRNDCLH ENYTWENSPM NFDHVGNAYL SLLQVATFKG WLQ IMNDAI DSREVHKQPI RETNIYMYLY FIFFIVFGSF FILKLFVCIL IDIFRQQRRK AEGLSATDSR TQLIYRRAVM RTMS AKPVK RIPKPGNKIQ GCIFDLVTNQ AFDISIMVLI CLNMVTMMVE KEGQSQHMTE VLYWINVVFI ILFTGECVLK LISLR HYYF TVGWNIFDFV VVIISIVGMF LADLIETYFV SPTLFRVIRL ARIGRILRLV KGAKGIRLLL LALRKALRTL FNVSFL LFV IMFVYAVFGM EFFMHIRDAG AIDDVYNFKT FGQSIILLFQ LATSAGWDGV YFAIANEEDC RAPDHELGYP GNCGSRA LG IAYLVSYLII TCLVVINMYA AVILDYVLEV YEDSKEGLTD DDYDMFFEVW QQFDPEATQY IRYDQLSELL EALQPPLQ V QKPNKYKILS MNIPICKDDH IFYKDVLEAL VKDVFSRRGS PVEAGDVQAP NVDEAEYKPV SSTLQRQREE YCVRLIQNA WRKHKQQN |
-分子 #3: beta-D-mannopyranose
| 分子 | 名称: beta-D-mannopyranose / タイプ: ligand / ID: 3 / コピー数: 1 / 式: BMA |
|---|---|
| 分子量 | 理論値: 180.156 Da |
| Chemical component information |  ChemComp-BMA: |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #5: 5-cyclopropyl-4-({1-[(1S)-1-(3,5-dichlorophenyl)ethyl]piperidin-4...
| 分子 | 名称: 5-cyclopropyl-4-({1-[(1S)-1-(3,5-dichlorophenyl)ethyl]piperidin-4-yl}methoxy)-2-fluoro-N-(methanesulfonyl)benzamide タイプ: ligand / ID: 5 / コピー数: 1 / 式: X7R |
|---|---|
| 分子量 | 理論値: 543.478 Da |
| Chemical component information | 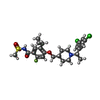 ChemComp-X7R: |
-分子 #6: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine
| 分子 | 名称: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine / タイプ: ligand / ID: 6 / コピー数: 5 / 式: PEE |
|---|---|
| 分子量 | 理論値: 744.034 Da |
| Chemical component information |  ChemComp-PEE: |
-分子 #7: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 78 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil R0./1 / 材質: GOLD / メッシュ: 300 詳細: Grids were incubated with a thiol reactive, self-assembling reaction mixture of 4mM monothiolalkane(C11)PEG6-OH (11-mercaptoundecyl) hexaethyleneglycol (SPT-0011P6, SensoPath Technologies, ...詳細: Grids were incubated with a thiol reactive, self-assembling reaction mixture of 4mM monothiolalkane(C11)PEG6-OH (11-mercaptoundecyl) hexaethyleneglycol (SPT-0011P6, SensoPath Technologies, Inc., Bozeman, MT). Grids were incubated with this self-assembled monolayer (SAM) solution for 24 hours and afterwards rinsed with EtOH. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | The sample was reconstituted into lipid nanodiscs (MSP1E3D1 in 3POPC:1POPE:1POPG) and was monodisperse. The sample was crosslinked with 0.05% glutaraldehyde for 10 minutes at RT, then quenched with 1M Tris pH7.0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 3.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8f0q: |
 ムービー
ムービー コントローラー
コントローラー














 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)