+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
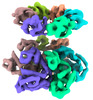
| タイトル | SV40 T-Antigen helicase hexamer | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | SV40 T-Antigen / VIRAL PROTEIN | ||||||||||||
| 生物種 |  Betapolyomavirus macacae (ウイルス) Betapolyomavirus macacae (ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.8 Å | ||||||||||||
 データ登録者 データ登録者 | Yuan Z / Langston L / Georgescu R / Li H / O'Donnell M | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: SV40 T-antigen uses a DNA shearing mechanism to initiate origin unwinding. 著者: Lance D Langston / Zuanning Yuan / Roxana Georgescu / Huilin Li / Michael E O'Donnell /  要旨: Duplication of DNA genomes requires unwinding of the double-strand (ds) DNA so that each single strand (ss) can be copied by a DNA polymerase. The genomes of eukaryotic cells are unwound by two ring- ...Duplication of DNA genomes requires unwinding of the double-strand (ds) DNA so that each single strand (ss) can be copied by a DNA polymerase. The genomes of eukaryotic cells are unwound by two ring-shaped hexameric helicases that initially encircle dsDNA but transition to ssDNA for function as replicative helicases. How the duplex is initially unwound, and the role of the two helicases in this process, is poorly understood. We recently described an initiation mechanism for eukaryotes in which the two helicases are directed inward toward one another and shear the duplex open by pulling on opposite strands of the duplex while encircling dsDNA [L. D. Langston, M. E. O'Donnell, , e46515 (2019)]. Two head-to-head T-Antigen helicases are long known to be loaded at the SV40 origin. We show here that T-Antigen tracks head (N-tier) first on ssDNA, opposite the direction proposed for decades. We also find that SV40 T-Antigen tracks directionally while encircling dsDNA and mainly tracks on one strand of the duplex in the same orientation as during ssDNA translocation. Further, two inward directed T-Antigen helicases on dsDNA are able to melt a 150-bp duplex. These findings explain the "rabbit ear" DNA loops observed at the SV40 origin by electron microscopy and reconfigure how the DNA loops emerge from the double hexamer relative to earlier models. Thus, the mechanism of DNA shearing by two opposing helicases is conserved in a eukaryotic viral helicase and may be widely used to initiate origin unwinding of dsDNA genomes. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28195.map.gz emd_28195.map.gz | 4.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28195-v30.xml emd-28195-v30.xml emd-28195.xml emd-28195.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28195.png emd_28195.png | 137.8 KB | ||
| Filedesc metadata |  emd-28195.cif.gz emd-28195.cif.gz | 4.6 KB | ||
| その他 |  emd_28195_half_map_1.map.gz emd_28195_half_map_1.map.gz emd_28195_half_map_2.map.gz emd_28195_half_map_2.map.gz | 48.8 MB 48.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28195 http://ftp.pdbj.org/pub/emdb/structures/EMD-28195 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28195 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28195 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28195_validation.pdf.gz emd_28195_validation.pdf.gz | 654.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28195_full_validation.pdf.gz emd_28195_full_validation.pdf.gz | 654.4 KB | 表示 | |
| XML形式データ |  emd_28195_validation.xml.gz emd_28195_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_28195_validation.cif.gz emd_28195_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28195 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28195 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28195 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28195 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28195.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28195.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_28195_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
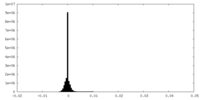
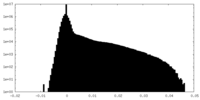
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28195_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
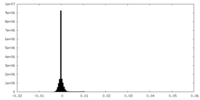
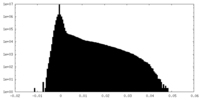
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SV40 T-Antigen
| 全体 | 名称: SV40 T-Antigen |
|---|---|
| 要素 |
|
-超分子 #1: SV40 T-Antigen
| 超分子 | 名称: SV40 T-Antigen / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Betapolyomavirus macacae (ウイルス) Betapolyomavirus macacae (ウイルス) |
-分子 #1: SV40 large antigen
| 分子 | 名称: SV40 large antigen / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:  Betapolyomavirus macacae (ウイルス) Betapolyomavirus macacae (ウイルス) |
| 配列 | 文字列: MDKVLNREES LQLMDLLGLE RSAWGNIPLM RKAYLKKCKE FHPDKGGDEE KMKKMNTLYK KMEDGVKYAH QPDFGGFWDA TEIPTYGTDE WEQWWNAFNE ENLFCSEEMP SSDDEATADS QHSTPPKKKR KVEDPKDFPS ELLSFLSHAV FSNRTLACFA IYTTKEKAAL ...文字列: MDKVLNREES LQLMDLLGLE RSAWGNIPLM RKAYLKKCKE FHPDKGGDEE KMKKMNTLYK KMEDGVKYAH QPDFGGFWDA TEIPTYGTDE WEQWWNAFNE ENLFCSEEMP SSDDEATADS QHSTPPKKKR KVEDPKDFPS ELLSFLSHAV FSNRTLACFA IYTTKEKAAL LYKKIMEKYS VTFISRHNSY NHNILFFLTP HRHRVSAINN YAQKLCTFSF LICKGVNKEY LMYSALTRDP FSVIEESLPG GLKEHDFNPE EAEETKQVSW KLVTEYAMET KCDDVLLLLG MYLEFQYSFE MCLKCIKKEQ PSHYKYHEKH YANAAIFADS KNQKTICQQA VDTVLAKKRV DSLQLTREQM LTNRFNDLLD RMDIMFGSTG SADIEEWMAG VAWLHCLLPK MDSVVYDFLK CMVYNIPKKR YWLFKGPIDS GKTTLAAALL ELCGGKALNV NLPLDRLNFE LGVAIDQFLV VFEDVKGTGG ESRDLPSGQG INNLDNLRDY LDGSVKVNLE KKHLNKRTQI FPPGIVTMNE FSVPKTLQAR FVKQIDFRAK DYLKHCLERS EFLLEKRIIQ SGIALLLMLI WYRPVAEFAQ SIQSRIVEWK ERLDKEFSLS VYQKMKFNVA MGIGVLDWLR NSDDDDEDSQ ENADKNEDGG EKNMEDSGHE TGIDSQSQGS FQAPQSSQSV HDHNQPYHIC RGFTCFKKPP TPPPEPET |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.0025 µm / 最小 デフォーカス(公称値): 0.0015 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 5.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 22375 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)