+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of lineage VII Lassa virus glycoprotein complex (strain Togo/2016/7082) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | glycoprotein complex / Lassa mammarenavirus / LASV / GPC / immune system / viral fusion protein / Lassa virus / lineage VI / Togo/2016/7082 / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / receptor-mediated endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) | |||||||||
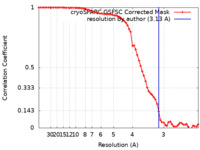
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.13 Å | |||||||||
 データ登録者 データ登録者 | Perrett HR / Ward AB | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2023 ジャーナル: Cell Rep / 年: 2023タイトル: Structural conservation of Lassa virus glycoproteins and recognition by neutralizing antibodies. 著者: Hailee R Perrett / Philip J M Brouwer / Jonathan Hurtado / Maddy L Newby / Lin Liu / Helena Müller-Kräuter / Sarah Müller Aguirre / Judith A Burger / Joey H Bouhuijs / Grace Gibson / ...著者: Hailee R Perrett / Philip J M Brouwer / Jonathan Hurtado / Maddy L Newby / Lin Liu / Helena Müller-Kräuter / Sarah Müller Aguirre / Judith A Burger / Joey H Bouhuijs / Grace Gibson / Terrence Messmer / John S Schieffelin / Aleksandar Antanasijevic / Geert-Jan Boons / Thomas Strecker / Max Crispin / Rogier W Sanders / Bryan Briney / Andrew B Ward /     要旨: Lassa fever is an acute hemorrhagic fever caused by the zoonotic Lassa virus (LASV). The LASV glycoprotein complex (GPC) mediates viral entry and is the sole target for neutralizing antibodies. ...Lassa fever is an acute hemorrhagic fever caused by the zoonotic Lassa virus (LASV). The LASV glycoprotein complex (GPC) mediates viral entry and is the sole target for neutralizing antibodies. Immunogen design is complicated by the metastable nature of recombinant GPCs and the antigenic differences among phylogenetically distinct LASV lineages. Despite the sequence diversity of the GPC, structures of most lineages are lacking. We present the development and characterization of prefusion-stabilized, trimeric GPCs of LASV lineages II, V, and VII, revealing structural conservation despite sequence diversity. High-resolution structures and biophysical characterization of the GPC in complex with GP1-A-specific antibodies suggest their neutralization mechanisms. Finally, we present the isolation and characterization of a trimer-preferring neutralizing antibody belonging to the GPC-B competition group with an epitope that spans adjacent protomers and includes the fusion peptide. Our work provides molecular detail information on LASV antigenic diversity and will guide efforts to design pan-LASV vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28181.map.gz emd_28181.map.gz | 14.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28181-v30.xml emd-28181-v30.xml emd-28181.xml emd-28181.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28181_fsc.xml emd_28181_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28181.png emd_28181.png | 143.6 KB | ||
| マスクデータ |  emd_28181_msk_1.map emd_28181_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28181.cif.gz emd-28181.cif.gz | 6.4 KB | ||
| その他 |  emd_28181_additional_1.map.gz emd_28181_additional_1.map.gz emd_28181_half_map_1.map.gz emd_28181_half_map_1.map.gz emd_28181_half_map_2.map.gz emd_28181_half_map_2.map.gz | 14.3 MB 22.1 MB 22.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28181 http://ftp.pdbj.org/pub/emdb/structures/EMD-28181 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28181 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28181 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ejgMC  8ejdC  8ejeC  8ejfC  8ejhC  8ejiC  8ejjC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28181.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28181.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28181_msk_1.map emd_28181_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
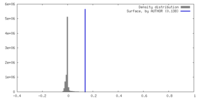
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_28181_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
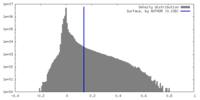
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28181_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28181_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lassa mammarenavirus GPC
| 全体 | 名称: Lassa mammarenavirus GPC |
|---|---|
| 要素 |
|
-超分子 #1: Lassa mammarenavirus GPC
| 超分子 | 名称: Lassa mammarenavirus GPC / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: GPC protein codon-optimized and expressed in HEK 293F cells using covalently-linked I53-50A trimerization scaffold. |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) / 株: Togo/2016/7082 Lassa mammarenavirus (ウイルス) / 株: Togo/2016/7082 |
| 分子量 | 理論値: 275 KDa |
-分子 #1: Glycoprotein GP1
| 分子 | 名称: Glycoprotein GP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 22.180051 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LIYKGSYELQ TLELNMETLN MTMPLSCTKN SSHHYIRVGN ETGLELTLTN TSIINHKFCN LSDAHKRNLY DHALMSILST FHLSIPNFN QYEAMSCDFN GGKISVQYNL SHAYAVDAAE HCGTVANGVL QTFMRMAWGG SYIALDSGCG NWDCIMTSYQ Y LIIQNTTW ...文字列: LIYKGSYELQ TLELNMETLN MTMPLSCTKN SSHHYIRVGN ETGLELTLTN TSIINHKFCN LSDAHKRNLY DHALMSILST FHLSIPNFN QYEAMSCDFN GGKISVQYNL SHAYAVDAAE HCGTVANGVL QTFMRMAWGG SYIALDSGCG NWDCIMTSYQ Y LIIQNTTW EDHCQFSRPS PIGYLGLLSQ RTRDIYIS UniProtKB: Pre-glycoprotein polyprotein GP complex |
-分子 #2: Glycoprotein GP2
| 分子 | 名称: Glycoprotein GP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 19.176748 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GTFTWTLSDS EGNETPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRLKSP AQMSIQLINK AVNALINDQ LIMKNHLRDM MCIPYCNYSK YWYLNHTSSG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE Y IDRQG UniProtKB: Pre-glycoprotein polyprotein GP complex |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: TBS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Wait time 10 s; blotting time varied between 3-7 s; blotting force of 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)