+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DH272.2 Fab in Complex with CH848 TF DS SOSIP Env | |||||||||
 マップデータ マップデータ | DH272.2 Fab bound to CH848 TF DS SOSIP Env | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus (ヒト免疫不全ウイルス) Human immunodeficiency virus (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 14.35 Å | |||||||||
 データ登録者 データ登録者 | Fera D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Front Immunol / 年: 2022 ジャーナル: Front Immunol / 年: 2022タイトル: Analysis of two cooperating antibodies unveils immune pressure imposed on HIV Env to elicit a V3-glycan supersite broadly neutralizing antibody lineage. 著者: Maxwell T Finkelstein / Emma Parker Miller / Molly C Erdman / Daniela Fera /  要旨: Elicitation of broadly neutralizing antibodies (bnAbs) is a goal of vaccine design as a strategy for targeting highly divergent strains of HIV-1. Current HIV-1 vaccine design efforts seek to elicit ...Elicitation of broadly neutralizing antibodies (bnAbs) is a goal of vaccine design as a strategy for targeting highly divergent strains of HIV-1. Current HIV-1 vaccine design efforts seek to elicit bnAbs by first eliciting their precursors through prime-boost regimens. This requires an understanding of the co-evolution between viruses and antibodies. Towards this goal, we have analyzed two cooperating antibodies, DH475 and DH272, which exerted pressure on the HIV population in an infected donor, called CH848, to evolve in such a way that it became sensitive to the V3-glycan supersite DH270 bnAb lineage. We obtained a 2.90Å crystal structure of DH475 in complex with the Man glycan and a negative stain EM model of DH272 in complex with the HIV-1 spike trimer, Env. Coupled with additional modeling studies and biochemical data, our studies reveal that DH475 contacts a V3- and V4-glycan dependent epitope accessible on an open or shed Env and that DH272 makes critical contacts with the V1V2 and V3 loops on HIV-1 Env. Using these data, we suggest a prime-boost regimen that may facilitate the initiation of DH270-like bnAb precursors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28120.map.gz emd_28120.map.gz | 14.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28120-v30.xml emd-28120-v30.xml emd-28120.xml emd-28120.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28120.png emd_28120.png | 13.8 KB | ||
| その他 |  emd_28120_half_map_1.map.gz emd_28120_half_map_1.map.gz emd_28120_half_map_2.map.gz emd_28120_half_map_2.map.gz | 26.6 MB 26.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28120 http://ftp.pdbj.org/pub/emdb/structures/EMD-28120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28120 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28120_validation.pdf.gz emd_28120_validation.pdf.gz | 690.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28120_full_validation.pdf.gz emd_28120_full_validation.pdf.gz | 689.9 KB | 表示 | |
| XML形式データ |  emd_28120_validation.xml.gz emd_28120_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_28120_validation.cif.gz emd_28120_validation.cif.gz | 11.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28120 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28120 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28120 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28120 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28120.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28120.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DH272.2 Fab bound to CH848 TF DS SOSIP Env | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: DH272.2 Fab bound to CH848 TF DS SOSIP Env
| ファイル | emd_28120_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DH272.2 Fab bound to CH848 TF DS SOSIP Env | ||||||||||||
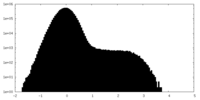
| 投影像・断面図 |
| ||||||||||||
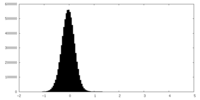
| 密度ヒストグラム |
-ハーフマップ: DH272.2 Fab bound to CH848 TF DS SOSIP Env
| ファイル | emd_28120_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DH272.2 Fab bound to CH848 TF DS SOSIP Env | ||||||||||||
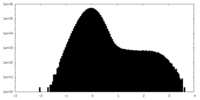
| 投影像・断面図 |
| ||||||||||||
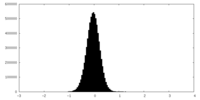
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex between DH272.2 Fab and CH848 TF DS SOSIP Env
| 全体 | 名称: Complex between DH272.2 Fab and CH848 TF DS SOSIP Env |
|---|---|
| 要素 |
|
-超分子 #1: Complex between DH272.2 Fab and CH848 TF DS SOSIP Env
| 超分子 | 名称: Complex between DH272.2 Fab and CH848 TF DS SOSIP Env タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 |
|---|
-超分子 #2: DH272.2 Fab
| 超分子 | 名称: DH272.2 Fab / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK-293T Homo sapiens (ヒト) / 組換細胞: HEK-293T |
-超分子 #3: CH848 TF DS SOSIP Env
| 超分子 | 名称: CH848 TF DS SOSIP Env / タイプ: complex / キメラ: Yes / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus (ヒト免疫不全ウイルス) Human immunodeficiency virus (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293S GnTi- Homo sapiens (ヒト) / 組換細胞: HEK293S GnTi- |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: OTHER / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 14.35 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 使用した粒子像数: 19124 |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X