+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM of P. calidifontis cytochrome filament | |||||||||
 マップデータ マップデータ | cryo-EM of P. calidifontis cytochrome filament | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | helical symmetry / cytochrome filmanet / conductive nanowires / microbial nanowires / ELECTRON TRANSPORT | |||||||||
| 機能・相同性 | Multiheme cytochrome superfamily / membrane / Cytochrome c family protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Pyrobaculum calidifontis (古細菌) Pyrobaculum calidifontis (古細菌) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Wang F / Cvirkaite-Krupovic V / Krupovic M / Egelman EH | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Extracellular cytochrome nanowires appear to be ubiquitous in prokaryotes. 著者: Diana P Baquero / Virginija Cvirkaite-Krupovic / Shengen Shawn Hu / Jessie Lynda Fields / Xing Liu / Christopher Rensing / Edward H Egelman / Mart Krupovic / Fengbin Wang /    要旨: Electrically conductive appendages from the anaerobic bacterium Geobacter sulfurreducens, recently identified as extracellular cytochrome nanowires (ECNs), have received wide attention due to ...Electrically conductive appendages from the anaerobic bacterium Geobacter sulfurreducens, recently identified as extracellular cytochrome nanowires (ECNs), have received wide attention due to numerous potential applications. However, whether other organisms employ similar ECNs for electron transfer remains unknown. Here, using cryoelectron microscopy, we describe the atomic structures of two ECNs from two major orders of hyperthermophilic archaea present in deep-sea hydrothermal vents and terrestrial hot springs. Homologs of Archaeoglobus veneficus ECN are widespread among mesophilic methane-oxidizing Methanoperedenaceae, alkane-degrading Syntrophoarchaeales archaea, and in the recently described megaplasmids called Borgs. The ECN protein subunits lack similarities in their folds; however, they share a common heme arrangement, suggesting an evolutionarily optimized heme packing for efficient electron transfer. The detection of ECNs in archaea suggests that filaments containing closely stacked hemes may be a common and widespread mechanism for long-range electron transfer in both prokaryotic domains of life. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27911.map.gz emd_27911.map.gz | 11.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27911-v30.xml emd-27911-v30.xml emd-27911.xml emd-27911.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27911.png emd_27911.png | 66.1 KB | ||
| Filedesc metadata |  emd-27911.cif.gz emd-27911.cif.gz | 5.9 KB | ||
| その他 |  emd_27911_half_map_1.map.gz emd_27911_half_map_1.map.gz emd_27911_half_map_2.map.gz emd_27911_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27911 http://ftp.pdbj.org/pub/emdb/structures/EMD-27911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27911 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27911_validation.pdf.gz emd_27911_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27911_full_validation.pdf.gz emd_27911_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_27911_validation.xml.gz emd_27911_validation.xml.gz | 14.1 KB | 表示 | |
| CIF形式データ |  emd_27911_validation.cif.gz emd_27911_validation.cif.gz | 16.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27911 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27911 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8e5fMC  8e5gC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27911.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27911.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-EM of P. calidifontis cytochrome filament | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
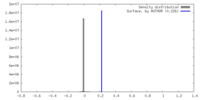
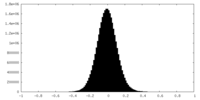
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half A
| ファイル | emd_27911_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
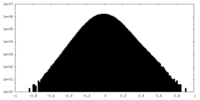
| 密度ヒストグラム |
-ハーフマップ: half B
| ファイル | emd_27911_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
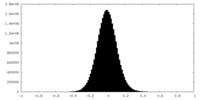
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : extracellular cytochrome filament
| 全体 | 名称: extracellular cytochrome filament |
|---|---|
| 要素 |
|
-超分子 #1: extracellular cytochrome filament
| 超分子 | 名称: extracellular cytochrome filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Pyrobaculum calidifontis (古細菌) Pyrobaculum calidifontis (古細菌) |
-分子 #1: c-type cytochrome
| 分子 | 名称: c-type cytochrome / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pyrobaculum calidifontis (古細菌) Pyrobaculum calidifontis (古細菌) |
| 分子量 | 理論値: 40.756312 KDa |
| 配列 | 文字列: MKKFPALITT LLLLAVFVAA TYGPPYSYNH PTNCISCHSN STGTANSQAL SGLTSGPAAG ACDPSQQECV WSHQVLKGTD VWKKCINCH VAIWNSINSG PGNVHSGLLN SYGCACHAVA HVGYGNPTDG YTACIYFYVP RLSTATPGYF GAKPTLDFRN V YICFKGTP ...文字列: MKKFPALITT LLLLAVFVAA TYGPPYSYNH PTNCISCHSN STGTANSQAL SGLTSGPAAG ACDPSQQECV WSHQVLKGTD VWKKCINCH VAIWNSINSG PGNVHSGLLN SYGCACHAVA HVGYGNPTDG YTACIYFYVP RLSTATPGYF GAKPTLDFRN V YICFKGTP EGTYTFSGNA PTSLMQLLES KGEVTVKALL VGYDKYANGT VKAKSSAADF LETDFFSALE QAGIFRYEWG TA SGAVLKN PSVRTHPLTE EAPNGETIVM GVFDIHTGDF ILVAPYAPYS RAPYYLPVAV NPGVAACFNC HFVYQGQLGT AKV MEVGGV WKIGIPADVL NSLTDPHKIV MPAAQAAGGG VAPNLSLVAL LATATLLGGA FLALRRRAQ UniProtKB: Cytochrome c family protein |
-分子 #2: HEME C
| 分子 | 名称: HEME C / タイプ: ligand / ID: 2 / コピー数: 4 / 式: HEC |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 32.38 Å 想定した対称性 - らせんパラメータ - ΔΦ: 54.72 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 101864 |
|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION |
| 初期モデル | モデルのタイプ: NONE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)