[日本語] English
 万見
万見- EMDB-27875: Full-length APOBEC3G in complex with HIV-1 Vif, CBF-beta, and fork RNA -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full-length APOBEC3G in complex with HIV-1 Vif, CBF-beta, and fork RNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Viral protein - human protein complex / ribonucleoprotein complex / VIRAL PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RUNX3 regulates RUNX1-mediated transcription / RUNX1 regulates transcription of genes involved in BCR signaling / RUNX1 regulates transcription of genes involved in interleukin signaling / RUNX2 regulates bone development / core-binding factor complex / RUNX1 regulates expression of components of tight junctions / positive regulation of CD8-positive, alpha-beta T cell differentiation / RUNX2 regulates chondrocyte maturation / single-stranded DNA cytosine deaminase / negative regulation of CD4-positive, alpha-beta T cell differentiation ...RUNX3 regulates RUNX1-mediated transcription / RUNX1 regulates transcription of genes involved in BCR signaling / RUNX1 regulates transcription of genes involved in interleukin signaling / RUNX2 regulates bone development / core-binding factor complex / RUNX1 regulates expression of components of tight junctions / positive regulation of CD8-positive, alpha-beta T cell differentiation / RUNX2 regulates chondrocyte maturation / single-stranded DNA cytosine deaminase / negative regulation of CD4-positive, alpha-beta T cell differentiation / negative regulation of single stranded viral RNA replication via double stranded DNA intermediate / : / DNA cytosine deamination / cytidine to uridine editing / lymphocyte differentiation / RUNX1 and FOXP3 control the development of regulatory T lymphocytes (Tregs) / cytidine deaminase activity / RUNX2 regulates genes involved in cell migration / Transcriptional regulation by RUNX2 / RUNX2 regulates genes involved in differentiation of myeloid cells / RUNX1 regulates transcription of genes involved in differentiation of keratinocytes / transposable element silencing / RUNX3 Regulates Immune Response and Cell Migration / myeloid cell differentiation / definitive hemopoiesis / RUNX1 regulates transcription of genes involved in differentiation of myeloid cells / Regulation of RUNX1 Expression and Activity / RUNX1 regulates transcription of genes involved in WNT signaling / RUNX1 regulates estrogen receptor mediated transcription / RUNX2 regulates osteoblast differentiation / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / RUNX3 regulates p14-ARF / cell maturation / viral life cycle / Regulation of RUNX3 expression and activity / P-body / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / virion component / Regulation of RUNX2 expression and activity / Transcriptional regulation of granulopoiesis / protein polyubiquitination / osteoblast differentiation / RUNX1 regulates transcription of genes involved in differentiation of HSCs / defense response to virus / Estrogen-dependent gene expression / sequence-specific DNA binding / transcription by RNA polymerase II / host cell cytoplasm / transcription coactivator activity / ribonucleoprotein complex / innate immune response / regulation of transcription by RNA polymerase II / host cell plasma membrane / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / RNA binding / zinc ion binding / nucleoplasm / nucleus / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
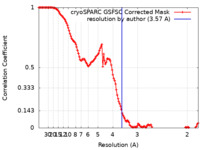
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.57 Å | |||||||||
 データ登録者 データ登録者 | Ito F / Alvarez-Cabrera AL / Liu S / Yang H / Shiriaeva A / Zhou ZH / Chen XS | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Structural basis for HIV-1 antagonism of host APOBEC3G via Cullin E3 ligase. 著者: Fumiaki Ito / Ana L Alvarez-Cabrera / Shiheng Liu / Hanjing Yang / Anna Shiriaeva / Z Hong Zhou / Xiaojiang S Chen /  要旨: Human APOBEC3G (A3G) is a virus restriction factor that inhibits HIV-1 replication and triggers lethal hypermutation on viral reverse transcripts. HIV-1 viral infectivity factor (Vif) breaches this ...Human APOBEC3G (A3G) is a virus restriction factor that inhibits HIV-1 replication and triggers lethal hypermutation on viral reverse transcripts. HIV-1 viral infectivity factor (Vif) breaches this host A3G immunity by hijacking a cellular E3 ubiquitin ligase complex to target A3G for ubiquitination and degradation. The molecular mechanism of A3G targeting by Vif-E3 ligase is unknown, limiting the antiviral efforts targeting this host-pathogen interaction crucial for HIV-1 infection. Here, we report the cryo-electron microscopy structures of A3G bound to HIV-1 Vif in complex with T cell transcription cofactor CBF-β and multiple components of the Cullin-5 RING E3 ubiquitin ligase. The structures reveal unexpected RNA-mediated interactions of Vif with A3G primarily through A3G's noncatalytic domain, while A3G's catalytic domain is poised for ubiquitin transfer. These structures elucidate the molecular mechanism by which HIV-1 Vif hijacks the host ubiquitin ligase to specifically target A3G to establish infection and offer structural information for the rational development of antiretroviral therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27875.map.gz emd_27875.map.gz | 108.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27875-v30.xml emd-27875-v30.xml emd-27875.xml emd-27875.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27875_fsc.xml emd_27875_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27875.png emd_27875.png | 69.1 KB | ||
| Filedesc metadata |  emd-27875.cif.gz emd-27875.cif.gz | 6.9 KB | ||
| その他 |  emd_27875_half_map_1.map.gz emd_27875_half_map_1.map.gz emd_27875_half_map_2.map.gz emd_27875_half_map_2.map.gz | 200.4 MB 200.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27875 http://ftp.pdbj.org/pub/emdb/structures/EMD-27875 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27875 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27875 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27875_validation.pdf.gz emd_27875_validation.pdf.gz | 716.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27875_full_validation.pdf.gz emd_27875_full_validation.pdf.gz | 716.2 KB | 表示 | |
| XML形式データ |  emd_27875_validation.xml.gz emd_27875_validation.xml.gz | 21.4 KB | 表示 | |
| CIF形式データ |  emd_27875_validation.cif.gz emd_27875_validation.cif.gz | 27.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27875 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27875 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27875 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27875 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8e40MC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27875.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27875.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.92 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_27875_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27875_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of APOBEC3G with HIV-1 Vif and CBF-beta bound to fork RNA
| 全体 | 名称: Complex of APOBEC3G with HIV-1 Vif and CBF-beta bound to fork RNA |
|---|---|
| 要素 |
|
-超分子 #1: Complex of APOBEC3G with HIV-1 Vif and CBF-beta bound to fork RNA
| 超分子 | 名称: Complex of APOBEC3G with HIV-1 Vif and CBF-beta bound to fork RNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 99 KDa |
-分子 #1: DNA dC->dU-editing enzyme APOBEC-3G
| 分子 | 名称: DNA dC->dU-editing enzyme APOBEC-3G / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: single-stranded DNA cytosine deaminase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.18743 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGGSGGMKP QIRNMVEPMD PRTFVSNFNN RPILSGLDTV WLCCEVKTKD PSGPPLDAKI FQGKVYPKAK YHPEMRFLRW FHKWRQLHH DQEYKVTWYV SWSPCTRCAN SVATFLAKDP KVTLTIFVAR LYYFWDPDYQ QALRILAEAG ATMKIMNYNE F QDCWNKFV ...文字列: GPGGSGGMKP QIRNMVEPMD PRTFVSNFNN RPILSGLDTV WLCCEVKTKD PSGPPLDAKI FQGKVYPKAK YHPEMRFLRW FHKWRQLHH DQEYKVTWYV SWSPCTRCAN SVATFLAKDP KVTLTIFVAR LYYFWDPDYQ QALRILAEAG ATMKIMNYNE F QDCWNKFV DGRGKPFKPW NNLPKHYTLL QATLGELLRH LMDPGTFTSN FNNKPWVSGQ HETYLCYKVE RLHNDTWVPL NQ HRGFLRN QAPNIHGFPK GRHAQLCFLD LIPFWKLDGQ QYRVTCFTSW SPCFSCAQEM AKFISNNEHV SLCIFAARIY DDQ GRYQEG LRTLHRDGAK IAMMNYSEFE YCWDTFVDRQ GRPFQPWDGL DEHSQALSGR LRAILQNQGN UniProtKB: DNA dC->dU-editing enzyme APOBEC-3G |
-分子 #2: Virion infectivity factor
| 分子 | 名称: Virion infectivity factor / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 21.136408 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPMENRWQVM IVWQVDRMRI NTWKRLVKHH MYISRKAKDW FYRHHYESTN PKISSEVHIP LGDAKLVITT YWGLHTGERD WHLGQGVSI EWRKKRYSTQ VDPDLADQLI HLHYFDCFSE SAIRNTILGR IVSPRCEYQA GHNKVGSLQY LALAALIKPK Q IKPPLPSV RKLTEDRWNK UniProtKB: Virion infectivity factor |
-分子 #3: Core-binding factor subunit beta
| 分子 | 名称: Core-binding factor subunit beta / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.643814 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPRVVPDQRS KFENEEFFRK LSRECEIKYT GFRDRPHEER QARFQNACRD GRSEIAFVAT GTNLSLQFFP ASWQGEQRQT PSREYVDLE REAGKVYLKA PMILNGVCVI WKGWIDLQRL DGMGCLEFDE ERAQQEDALA QQAFEEARRR TREFEDRD UniProtKB: Core-binding factor subunit beta |
-分子 #4: RNA
| 分子 | 名称: RNA / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 6.875011 KDa |
| 配列 | 文字列: UUUUUUUUUU UUUUAAAAAA AA |
-分子 #5: RNA
| 分子 | 名称: RNA / タイプ: rna / ID: 5 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.00087 KDa |
| 配列 | 文字列: UUUUUUUUAA AAAAAAAAAA AAAAA |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 11803 / 平均露光時間: 8.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 150000 |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)