+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | RNA-mediated APOBEC3G dimer | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
| Biological species |  | |||||||||
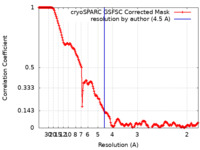
| Method | single particle reconstruction / cryo EM / Resolution: 4.5 Å | |||||||||
 Authors Authors | Ito F / Alvarez-Cabrera AL / Liu S / Yang H / Shiriaeva A / Zhou ZH / Chen XS | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: Sci Adv / Year: 2023 Journal: Sci Adv / Year: 2023Title: Structural basis for HIV-1 antagonism of host APOBEC3G via Cullin E3 ligase. Authors: Fumiaki Ito / Ana L Alvarez-Cabrera / Shiheng Liu / Hanjing Yang / Anna Shiriaeva / Z Hong Zhou / Xiaojiang S Chen /  Abstract: Human APOBEC3G (A3G) is a virus restriction factor that inhibits HIV-1 replication and triggers lethal hypermutation on viral reverse transcripts. HIV-1 viral infectivity factor (Vif) breaches this ...Human APOBEC3G (A3G) is a virus restriction factor that inhibits HIV-1 replication and triggers lethal hypermutation on viral reverse transcripts. HIV-1 viral infectivity factor (Vif) breaches this host A3G immunity by hijacking a cellular E3 ubiquitin ligase complex to target A3G for ubiquitination and degradation. The molecular mechanism of A3G targeting by Vif-E3 ligase is unknown, limiting the antiviral efforts targeting this host-pathogen interaction crucial for HIV-1 infection. Here, we report the cryo-electron microscopy structures of A3G bound to HIV-1 Vif in complex with T cell transcription cofactor CBF-β and multiple components of the Cullin-5 RING E3 ubiquitin ligase. The structures reveal unexpected RNA-mediated interactions of Vif with A3G primarily through A3G's noncatalytic domain, while A3G's catalytic domain is poised for ubiquitin transfer. These structures elucidate the molecular mechanism by which HIV-1 Vif hijacks the host ubiquitin ligase to specifically target A3G to establish infection and offer structural information for the rational development of antiretroviral therapeutics. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_27887.map.gz emd_27887.map.gz | 254.6 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-27887-v30.xml emd-27887-v30.xml emd-27887.xml emd-27887.xml | 15.2 KB 15.2 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_27887_fsc.xml emd_27887_fsc.xml | 16.9 KB | Display |  FSC data file FSC data file |
| Images |  emd_27887.png emd_27887.png | 22.8 KB | ||
| Others |  emd_27887_half_map_1.map.gz emd_27887_half_map_1.map.gz emd_27887_half_map_2.map.gz emd_27887_half_map_2.map.gz | 475 MB 475 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27887 http://ftp.pdbj.org/pub/emdb/structures/EMD-27887 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27887 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27887 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_27887.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_27887.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.92 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #2
| File | emd_27887_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_27887_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : APOBEC3G dimer mediated by RNA
| Entire | Name: APOBEC3G dimer mediated by RNA |
|---|---|
| Components |
|
-Supramolecule #1: APOBEC3G dimer mediated by RNA
| Supramolecule | Name: APOBEC3G dimer mediated by RNA / type: complex / ID: 1 / Chimera: Yes / Parent: 0 / Macromolecule list: all |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 108 KDa |
-Macromolecule #1: APOBEC3G
| Macromolecule | Name: APOBEC3G / type: protein_or_peptide / ID: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Recombinant expression | Organism:  |
| Sequence | String: GPGGSGGMKP QIRNMVEPMD PRTFVSNFNN RPILSGLDTV WLCCEVKTKD PSGPPLDAKI FQGKVYPKAK YHPEMRFLRW FHKWRQLHHD QEYKVTWYVS WSPCTRCANS VATFLAKDPK VTLTIFVARL YYFWDPDYQQ ALRILAEAGA TMKIMNYNEF QDCWNKFVDG ...String: GPGGSGGMKP QIRNMVEPMD PRTFVSNFNN RPILSGLDTV WLCCEVKTKD PSGPPLDAKI FQGKVYPKAK YHPEMRFLRW FHKWRQLHHD QEYKVTWYVS WSPCTRCANS VATFLAKDPK VTLTIFVARL YYFWDPDYQQ ALRILAEAGA TMKIMNYNEF QDCWNKFVDG RGKPFKPWNN LPKHYTLLQA TLGELLRHLM DPGTFTSNFN NKPWVSGQHE TYLCYKVERL HNDTWVPLNQ HRGFLRNQAP NIHGFPKGRH AQLCFLDLIP FWKLDGQQYR VTCFTSWSPC FSCAQEMAKF ISNNEHVSLC IFAARIYDDQ GRYQEGLRTL HRDGAKIAMM NYSEFEYCWD TFVDRQGRPF QPWDGLDEHS QALSGRLRAI LQNQGN |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.15 mg/mL |
|---|---|
| Buffer | pH: 7.5 |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | TFS GLACIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Number grids imaged: 1 / Number real images: 11803 / Average exposure time: 8.0 sec. / Average electron dose: 40.0 e/Å2 |
| Electron beam | Acceleration voltage: 200 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.0 mm / Nominal defocus max: 3.0 µm / Nominal defocus min: 1.2 µm / Nominal magnification: 150000 |
 Movie
Movie Controller
Controller







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)