+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Influenza A virus A/Melbourne/1/1946 (H1N1) hemagglutinin bound to CR6261 Fab | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NIAID hemagglutinin stalk binding antibody / Structural Genomics / Seattle Structural Genomics Center for Infectious Disease / SSGCID / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
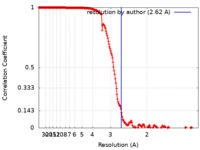
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.62 Å | |||||||||
 データ登録者 データ登録者 | Seattle Structural Genomics Center for Infectious Disease (SSGCID) | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To be published ジャーナル: To be publishedタイトル: CryoEM structure of Influenza A virus A/Melbourne/1/1946 (H1N1) hemagglutinin bound to CR6261 Fab 著者: Yang M / Edwards TE / Horanyi PS / Lorimer DD | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27441.map.gz emd_27441.map.gz | 624.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27441-v30.xml emd-27441-v30.xml emd-27441.xml emd-27441.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27441_fsc.xml emd_27441_fsc.xml | 19.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27441.png emd_27441.png | 56.6 KB | ||
| Filedesc metadata |  emd-27441.cif.gz emd-27441.cif.gz | 6.8 KB | ||
| その他 |  emd_27441_half_map_1.map.gz emd_27441_half_map_1.map.gz emd_27441_half_map_2.map.gz emd_27441_half_map_2.map.gz | 614.8 MB 614.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27441 http://ftp.pdbj.org/pub/emdb/structures/EMD-27441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27441 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27441_validation.pdf.gz emd_27441_validation.pdf.gz | 929.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27441_full_validation.pdf.gz emd_27441_full_validation.pdf.gz | 928.8 KB | 表示 | |
| XML形式データ |  emd_27441_validation.xml.gz emd_27441_validation.xml.gz | 28.2 KB | 表示 | |
| CIF形式データ |  emd_27441_validation.cif.gz emd_27441_validation.cif.gz | 37.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27441 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27441 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27441.map.gz / 形式: CCP4 / 大きさ: 662.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27441.map.gz / 形式: CCP4 / 大きさ: 662.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.675 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_27441_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
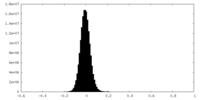
| 投影像・断面図 |
| ||||||||||||
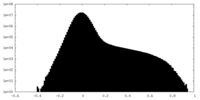
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27441_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
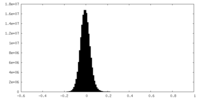
| 投影像・断面図 |
| ||||||||||||
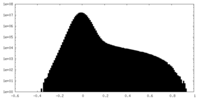
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CryoEM structure of Influenza A virus A/Melbourne/1/1946 (H1N1) h...
| 全体 | 名称: CryoEM structure of Influenza A virus A/Melbourne/1/1946 (H1N1) hemagglutinin bound to CR6261 Fab |
|---|---|
| 要素 |
|
-超分子 #1: CryoEM structure of Influenza A virus A/Melbourne/1/1946 (H1N1) h...
| 超分子 | 名称: CryoEM structure of Influenza A virus A/Melbourne/1/1946 (H1N1) hemagglutinin bound to CR6261 Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 分子量 | 理論値: 450 KDa |
-分子 #1: Hemagglutinin HA1 chain
| 分子 | 名称: Hemagglutinin HA1 chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス)株: A/Melbourne/1/1946(H1N1) |
| 分子量 | 理論値: 40.34752 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MVLVNQSHQG FNKEHTSKMV SAIVLYVLLA AAAHSAFAGS DTICIGYHAN NSTDTVDTVL EKNVTVTHSV NLLEDSHNGK LCRLKGIAP LQLGKCNIAG WILGNPECDS LLPASSWSYI VETPNSKNGI CYPGDFIDYE ELREQLSSVS SFERFEIFPK E SSWPKHST ...文字列: MVLVNQSHQG FNKEHTSKMV SAIVLYVLLA AAAHSAFAGS DTICIGYHAN NSTDTVDTVL EKNVTVTHSV NLLEDSHNGK LCRLKGIAP LQLGKCNIAG WILGNPECDS LLPASSWSYI VETPNSKNGI CYPGDFIDYE ELREQLSSVS SFERFEIFPK E SSWPKHST TKGVTAACSH AGKSSFYRNL LWLTKKEDSY PKLSNSYVNK KGKEVLVLWG VHHPSSSKEQ QTLYQNENAY VS VVSSNYN RRFIPEIAER PEVKDQAGRI NYYWTLLEPG DTIIFEANGN LVAPWYAFAL SRGFGSGIIT SNASMHECNT KCQ TPQGAI NSSLPFQNIH PVTIGECPKY VKSAKLRMVT GLRNIPSI |
-分子 #2: Hemagglutinin HA2 chain
| 分子 | 名称: Hemagglutinin HA2 chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス)株: A/Melbourne/1/1946(H1N1) |
| 分子量 | 理論値: 23.845508 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GLFGAIAGFI EGGWTGMIDG WYGYHHQNEQ GSGYAADQKS TQNAINGITN KVNSVIEKMN TQFTAVGKEF NNLEKRMENL NKKVDDGFL DIWTYNAELL VLLENERTLD FHDSNVKNLY EKVKIQLKNN AKEIGNGCFE FYHKCDNECM ESVRNGTYDY P KYSKEFLV ...文字列: GLFGAIAGFI EGGWTGMIDG WYGYHHQNEQ GSGYAADQKS TQNAINGITN KVNSVIEKMN TQFTAVGKEF NNLEKRMENL NKKVDDGFL DIWTYNAELL VLLENERTLD FHDSNVKNLY EKVKIQLKNN AKEIGNGCFE FYHKCDNECM ESVRNGTYDY P KYSKEFLV PRGSPGSGYI PEAPRDGQAY VRKDGEWVLL STFLGHHHHH H |
-分子 #3: CR6261 Fab heavy chain
| 分子 | 名称: CR6261 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.066621 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEWSWVFLFF LSVTTGVHSE VQLVESGAEV KKPGSSVKVS CKASGGPFRS YAISWVRQAP GQGPEWMGGI IPIFGTTKYA PKFQGRVTI TADDFAGTVY MELSSLRSED TAMYYCAKHM GYQVRETMDV WGKGTTVTVS SASTKGPSVF PLAPSSKSTS G GTAALGCL ...文字列: MEWSWVFLFF LSVTTGVHSE VQLVESGAEV KKPGSSVKVS CKASGGPFRS YAISWVRQAP GQGPEWMGGI IPIFGTTKYA PKFQGRVTI TADDFAGTVY MELSSLRSED TAMYYCAKHM GYQVRETMDV WGKGTTVTVS SASTKGPSVF PLAPSSKSTS G GTAALGCL VKDYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKRV EP KSCDKHH HHHH |
-分子 #4: CR6261 Fab light chain
| 分子 | 名称: CR6261 Fab light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.514395 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEWSWVFLFF LSVTTGVHSQ SVLTQPPSVS AAPGQKVTIS CSGSSSNIGN DYVSWYQQLP GTAPKLLIYD NNKRPSGIPD RFSGSKSGT SATLGITGLQ TGDEANYYCA TWDRRPTAYV VFGGGTKLTV LGAAAGQPKA APSVTLFPPS SEELQANKAT L VCLISDFY ...文字列: MEWSWVFLFF LSVTTGVHSQ SVLTQPPSVS AAPGQKVTIS CSGSSSNIGN DYVSWYQQLP GTAPKLLIYD NNKRPSGIPD RFSGSKSGT SATLGITGLQ TGDEANYYCA TWDRRPTAYV VFGGGTKLTV LGAAAGQPKA APSVTLFPPS SEELQANKAT L VCLISDFY PGAVTVAWKA DSSPVKAGVE TTTPSKQSNN KYAASSYLSL TPEQWKSHRS YSCQVTHEGS TVEKTVAPTE CS |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 61 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: LACEY | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 9179 / 平均露光時間: 2.4 sec. / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)