+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Gokushovirus EC6098 | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of the gokushovirus EC6098 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Capsid / VIRUS | |||||||||
| 機能・相同性 | Microviridae F protein / Microviridae F protein superfamily / Capsid protein (F protein) / Capsid/spike protein, ssDNA virus / T=1 icosahedral viral capsid / structural molecule activity / Major capsid protein / Putative DNA binding protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Escherichia phage EC6098 (ファージ) Escherichia phage EC6098 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Lee H / Fane BA / Hafenstein SL | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2022 ジャーナル: J Virol / 年: 2022タイトル: Cryo-EM Structure of Gokushovirus ΦEC6098 Reveals a Novel Capsid Architecture for a Single-Scaffolding Protein, Microvirus Assembly System. 著者: Hyunwook Lee / Alexis J Baxter / Carol M Bator / Bentley A Fane / Susan L Hafenstein /  要旨: Ubiquitous and abundant in ecosystems and microbiomes, gokushoviruses constitute a subfamily, distantly related to bacteriophages ΦX174, α3, and G4. A high-resolution cryo-EM structure of ...Ubiquitous and abundant in ecosystems and microbiomes, gokushoviruses constitute a subfamily, distantly related to bacteriophages ΦX174, α3, and G4. A high-resolution cryo-EM structure of gokushovirus ΦEC6098 was determined, and the atomic model was built . Although gokushoviruses lack external scaffolding and spike proteins, which extensively interact with the ΦX174 capsid protein, the core of the ΦEC6098 coat protein (VP1) displayed a similar structure. There are, however, key differences. At each ΦEC6098 icosahedral 3-fold axis, a long insertion loop formed mushroom-like protrusions, which have been noted in lower-resolution gokushovirus structures. Hydrophobic interfaces at the bottom of these protrusions may confer stability to the capsid shell. In ΦX174, the N-terminus of the capsid protein resides directly atop the 3-fold axes of symmetry; however, the ΦEC6098 N-terminus stretched across the inner surface of the capsid shell, reaching nearly to the 5-fold axis of the neighboring pentamer. Thus, this extended N-terminus interconnected pentamers on the inside of the capsid shell, presumably promoting capsid assembly, a function performed by the ΦX174 external scaffolding protein. There were also key differences between the ΦX174-like DNA-binding J proteins and its ΦEC6098 homologue VP8. As seen with the J proteins, C-terminal VP8 residues were bound into a pocket within the major capsid protein; however, its N-terminal residues were disordered, likely due to flexibility. We show that the combined location and interaction of VP8's C-terminus and a portion of VP1's N-terminus are reminiscent of those seen with the ΦX174 and α3 J proteins. There is a dramatic structural and morphogenetic divide within the . The well-studied ΦX174-like viruses have prominent spikes at their icosahedral vertices, which are absent in gokushoviruses. Instead, gokushovirus major coat proteins form extensive mushroom-like protrusions at the 3-fold axes of symmetry. In addition, gokushoviruses lack an external scaffolding protein, the more critical of the two ΦX174 assembly proteins, but retain an internal scaffolding protein. The ΦEC6098 virion suggests that key external scaffolding functions are likely performed by coat protein domains unique to gokushoviruses. Thus, within one family, different assembly paths have been taken, demonstrating how a two-scaffolding protein system can evolve into a one-scaffolding protein system, or vice versa. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27397.map.gz emd_27397.map.gz | 371.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27397-v30.xml emd-27397-v30.xml emd-27397.xml emd-27397.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27397.png emd_27397.png | 126 KB | ||
| Filedesc metadata |  emd-27397.cif.gz emd-27397.cif.gz | 5.4 KB | ||
| その他 |  emd_27397_additional_1.map.gz emd_27397_additional_1.map.gz emd_27397_half_map_1.map.gz emd_27397_half_map_1.map.gz emd_27397_half_map_2.map.gz emd_27397_half_map_2.map.gz | 429.8 MB 373 MB 373.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27397 http://ftp.pdbj.org/pub/emdb/structures/EMD-27397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27397 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27397_validation.pdf.gz emd_27397_validation.pdf.gz | 667.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27397_full_validation.pdf.gz emd_27397_full_validation.pdf.gz | 667.1 KB | 表示 | |
| XML形式データ |  emd_27397_validation.xml.gz emd_27397_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_27397_validation.cif.gz emd_27397_validation.cif.gz | 21.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27397 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27397 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27397 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27397 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8desMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27397.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27397.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the gokushovirus EC6098 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
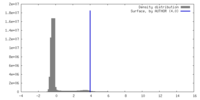
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: The sharpened map of the gokushovirus EC6098
| ファイル | emd_27397_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The sharpened map of the gokushovirus EC6098 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
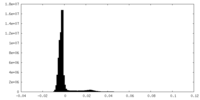
| 密度ヒストグラム |
-ハーフマップ: The first half map of the gokushovirus EC6098
| ファイル | emd_27397_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The first half map of the gokushovirus EC6098 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: The second half map of the gokushovirus EC6098
| ファイル | emd_27397_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The second half map of the gokushovirus EC6098 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia phage EC6098
| 全体 | 名称:  Escherichia phage EC6098 (ファージ) Escherichia phage EC6098 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia phage EC6098
| 超分子 | 名称: Escherichia phage EC6098 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2720215 / 生物種: Escherichia phage EC6098 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage EC6098 (ファージ) Escherichia phage EC6098 (ファージ) |
| 分子量 | 理論値: 63.367758 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKFGRKVPS NAKSQHNFSV IPSANIQRSV FNRSSGYKTT FDAGYLIPVF LDEALPGDTF HLKTSVLARL STPVVPFMDN LRLDIQYFS VPYRLVWDNW QKFNGEQKNP GDSTDYLIPQ IKAPAGGFPV GSLADYFGVP TGVENISVSA LPFRAYNLIY N EWYRDENL ...文字列: MSKFGRKVPS NAKSQHNFSV IPSANIQRSV FNRSSGYKTT FDAGYLIPVF LDEALPGDTF HLKTSVLARL STPVVPFMDN LRLDIQYFS VPYRLVWDNW QKFNGEQKNP GDSTDYLIPQ IKAPAGGFPV GSLADYFGVP TGVENISVSA LPFRAYNLIY N EWYRDENL INSAPLPLGD EEETGLANFP LRKRAKRHDY FTSALPWPQK GEGVEIGLGV PPSYTLEYPY YKEGMGFISS NY GASGNIG RTFPTYIARS SVGDDSSSNI GNAAYFSEGL ENGINFPNAP ARGRYDVLGG FDPNTPPVTL KKEGGEVVDN LTI NSLRQA FQLQRLLERD ARGGTRYIEI IRSHFGVISP DARVQRPEYL GSGSFDININ PVLQNSATTD ASPQGNLAAY GVSG GVNRG FSHSFVEHCF VIGLVSVRAD LTYQQGIPRM FSRQTRFDFY WPALAHLGEQ AILNKEIYAQ GNAKDDEVFG YQERY AEYR YRPSQITGKL RSTDPQSLDV WHLAQRFDSL PALNQEFIEE NPPMKRVLAV QDEPQFIMDA FFDLKCVRPM PVYSVP GLI DHF UniProtKB: Major capsid protein |
-分子 #2: Putative DNA binding protein
| 分子 | 名称: Putative DNA binding protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage EC6098 (ファージ) Escherichia phage EC6098 (ファージ) |
| 分子量 | 理論値: 4.83082 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARSRRRMSK RSSRRSFRKY AKTHKRNFKA RSMRGGIRL UniProtKB: Putative DNA binding protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 32308 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)