+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast mitochondrial small subunit assembly intermediate (State 2) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ribonucleoprotein complex / Mitochondria Biogenesis / RIBOSOME | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial small ribosomal subunit assembly / mitochondrial translational initiation / Mitochondrial protein degradation / mitochondrial ribosome / mitochondrial small ribosomal subunit / mitochondrial translation / sporulation resulting in formation of a cellular spore / superoxide dismutase activity / RNA splicing / methyltransferase activity ...mitochondrial small ribosomal subunit assembly / mitochondrial translational initiation / Mitochondrial protein degradation / mitochondrial ribosome / mitochondrial small ribosomal subunit / mitochondrial translation / sporulation resulting in formation of a cellular spore / superoxide dismutase activity / RNA splicing / methyltransferase activity / mRNA processing / peroxisome / 4 iron, 4 sulfur cluster binding / ribosomal small subunit biogenesis / ribosomal small subunit assembly / small ribosomal subunit / small ribosomal subunit rRNA binding / mitochondrial inner membrane / rRNA binding / structural constituent of ribosome / ribosome / translation / mRNA binding / GTP binding / mitochondrion / RNA binding / ATP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
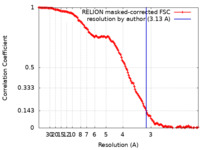
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.13 Å | |||||||||
 データ登録者 データ登録者 | Burnside C / Harper NJ / Klinge S | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Principles of mitoribosomal small subunit assembly in eukaryotes. 著者: Nathan J Harper / Chloe Burnside / Sebastian Klinge /  要旨: Mitochondrial ribosomes (mitoribosomes) synthesize proteins encoded within the mitochondrial genome that are assembled into oxidative phosphorylation complexes. Thus, mitoribosome biogenesis is ...Mitochondrial ribosomes (mitoribosomes) synthesize proteins encoded within the mitochondrial genome that are assembled into oxidative phosphorylation complexes. Thus, mitoribosome biogenesis is essential for ATP production and cellular metabolism. Here we used cryo-electron microscopy to determine nine structures of native yeast and human mitoribosomal small subunit assembly intermediates, illuminating the mechanistic basis for how GTPases are used to control early steps of decoding centre formation, how initial rRNA folding and processing events are mediated, and how mitoribosomal proteins have active roles during assembly. Furthermore, this series of intermediates from two species with divergent mitoribosomal architecture uncovers both conserved principles and species-specific adaptations that govern the maturation of mitoribosomal small subunits in eukaryotes. By revealing the dynamic interplay between assembly factors, mitoribosomal proteins and rRNA that are required to generate functional subunits, our structural analysis provides a vignette for how molecular complexity and diversity can evolve in large ribonucleoprotein assemblies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27250.map.gz emd_27250.map.gz | 227.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27250-v30.xml emd-27250-v30.xml emd-27250.xml emd-27250.xml | 56.1 KB 56.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27250_fsc.xml emd_27250_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27250.png emd_27250.png | 50.6 KB | ||
| マスクデータ |  emd_27250_msk_1.map emd_27250_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27250.cif.gz emd-27250.cif.gz | 13.7 KB | ||
| その他 |  emd_27250_half_map_1.map.gz emd_27250_half_map_1.map.gz emd_27250_half_map_2.map.gz emd_27250_half_map_2.map.gz | 191.9 MB 192.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27250 http://ftp.pdbj.org/pub/emdb/structures/EMD-27250 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27250 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27250 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27250_validation.pdf.gz emd_27250_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27250_full_validation.pdf.gz emd_27250_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_27250_validation.xml.gz emd_27250_validation.xml.gz | 22.4 KB | 表示 | |
| CIF形式データ |  emd_27250_validation.cif.gz emd_27250_validation.cif.gz | 29.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27250 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27250 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27250 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27250 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8d8kMC  8cspC  8csqC  8csrC  8cssC  8cstC  8csuC  8d8jC  8d8lC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27250.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27250.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
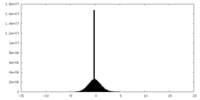
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.057 Å | ||||||||||||||||||||||||||||||||||||
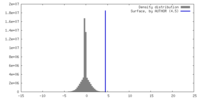
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
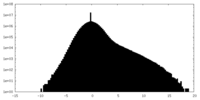
-マスク #1
| ファイル |  emd_27250_msk_1.map emd_27250_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
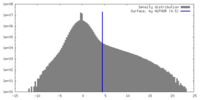
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27250_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
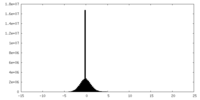
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27250_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
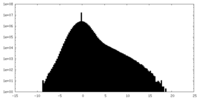
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Yeast mitochondrial small subunit assembly intermediate (State 2)
+超分子 #1: Yeast mitochondrial small subunit assembly intermediate (State 2)
+分子 #1: Probable S-adenosyl-L-methionine-dependent RNA methyltransferase ...
+分子 #2: 37S ribosomal protein MRP13, mitochondrial
+分子 #3: Ribosomal protein VAR1, mitochondrial
+分子 #4: Mitochondrial group I intron splicing factor CCM1
+分子 #5: 37S ribosomal protein SWS2, mitochondrial
+分子 #6: 37S ribosomal protein MRP2, mitochondrial
+分子 #7: 37S ribosomal protein S28, mitochondrial
+分子 #8: 37S ribosomal protein S16, mitochondrial
+分子 #9: 37S ribosomal protein S17, mitochondrial
+分子 #10: 37S ribosomal protein RSM18, mitochondrial
+分子 #11: 37S ribosomal protein S19, mitochondrial
+分子 #12: 37S ribosomal protein MRP21, mitochondrial
+分子 #13: 37S ribosomal protein S25, mitochondrial
+分子 #14: 37S ribosomal protein PET123, mitochondrial
+分子 #15: 37S ribosomal protein S23, mitochondrial
+分子 #16: Mitochondrial 37S ribosomal protein S27
+分子 #17: 37S ribosomal protein S24, mitochondrial
+分子 #18: 37S ribosomal protein MRP10, mitochondrial
+分子 #19: 37S ribosomal protein MRP51, mitochondrial
+分子 #20: 37S ribosomal protein MRP4, mitochondrial
+分子 #21: Protein FYV4, mitochondrial
+分子 #22: 37S ribosomal protein S26, mitochondrial
+分子 #23: 37S ribosomal protein NAM9, mitochondrial
+分子 #24: 37S ribosomal protein MRP1, mitochondrial
+分子 #25: 37S ribosomal protein S5, mitochondrial
+分子 #26: 37S ribosomal protein MRP17, mitochondrial
+分子 #27: 37S ribosomal protein S35, mitochondrial
+分子 #28: 37S ribosomal protein S7, mitochondrial
+分子 #29: 37S ribosomal protein S8, mitochondrial
+分子 #30: 37S ribosomal protein S9, mitochondrial
+分子 #32: 37S ribosomal protein S10, mitochondrial
+分子 #33: 37S ribosomal protein S18, mitochondrial
+分子 #34: unknown protein sequence
+分子 #35: MRPS12 isoform 1
+分子 #31: 15S ribosomal RNA
+分子 #36: IRON/SULFUR CLUSTER
+分子 #37: MAGNESIUM ION
+分子 #38: ADENOSINE-5'-TRIPHOSPHATE
+分子 #39: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 61.73 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 64000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)