+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure of Inactive MOR Bound to Alvimopan and Mb6 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Antagonist / Complex / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Opioid Signalling / Peptide ligand-binding receptors / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / G-protein activation / G protein-coupled opioid receptor activity / G protein-coupled opioid receptor signaling pathway / G alpha (i) signalling events / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway ...Opioid Signalling / Peptide ligand-binding receptors / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / G-protein activation / G protein-coupled opioid receptor activity / G protein-coupled opioid receptor signaling pathway / G alpha (i) signalling events / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / negative regulation of nitric oxide biosynthetic process / regulation of NMDA receptor activity / positive regulation of neurogenesis / negative regulation of cytosolic calcium ion concentration / transmission of nerve impulse / G-protein alpha-subunit binding / sensory perception of pain / presynaptic modulation of chemical synaptic transmission / locomotory behavior / G protein-coupled receptor activity / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / GABA-ergic synapse / adenylate cyclase-activating dopamine receptor signaling pathway / presynapse / perikaryon / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of ERK1 and ERK2 cascade / endosome / axon / dendrite / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Robertson MJ / Skiniotis G | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Structure determination of inactive-state GPCRs with a universal nanobody. 著者: Michael J Robertson / Makaía M Papasergi-Scott / Feng He / Alpay B Seven / Justin G Meyerowitz / Ouliana Panova / Maria Claudia Peroto / Tao Che / Georgios Skiniotis /  要旨: Cryogenic electron microscopy (cryo-EM) has widened the field of structure-based drug discovery by allowing for routine determination of membrane protein structures previously intractable. Despite ...Cryogenic electron microscopy (cryo-EM) has widened the field of structure-based drug discovery by allowing for routine determination of membrane protein structures previously intractable. Despite representing one of the largest classes of therapeutic targets, most inactive-state G protein-coupled receptors (GPCRs) have remained inaccessible for cryo-EM because their small size and membrane-embedded nature impedes projection alignment for high-resolution map reconstructions. Here we demonstrate that the same single-chain camelid antibody (nanobody) recognizing a grafted intracellular loop can be used to obtain cryo-EM structures of inactive-state GPCRs at resolutions comparable or better than those obtained by X-ray crystallography. Using this approach, we obtained structures of neurotensin 1 receptor bound to antagonist SR48692, μ-opioid receptor bound to alvimopan, apo somatostatin receptor 2 and histamine receptor 2 bound to famotidine. We expect this rapid, straightforward approach to facilitate the broad exploration of GPCR inactive states without the need for extensive engineering and crystallization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26591.map.gz emd_26591.map.gz | 107.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26591-v30.xml emd-26591-v30.xml emd-26591.xml emd-26591.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26591.png emd_26591.png | 92 KB | ||
| Filedesc metadata |  emd-26591.cif.gz emd-26591.cif.gz | 6.1 KB | ||
| その他 |  emd_26591_additional_1.map.gz emd_26591_additional_1.map.gz emd_26591_half_map_1.map.gz emd_26591_half_map_1.map.gz emd_26591_half_map_2.map.gz emd_26591_half_map_2.map.gz | 1.5 MB 116.2 MB 116.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26591 http://ftp.pdbj.org/pub/emdb/structures/EMD-26591 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26591 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26591 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26591_validation.pdf.gz emd_26591_validation.pdf.gz | 935.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26591_full_validation.pdf.gz emd_26591_full_validation.pdf.gz | 934.9 KB | 表示 | |
| XML形式データ |  emd_26591_validation.xml.gz emd_26591_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_26591_validation.cif.gz emd_26591_validation.cif.gz | 16.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26591 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26591 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26591 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26591 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26591.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26591.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8677 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Phenix model-free density modified map
| ファイル | emd_26591_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Phenix model-free density modified map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26591_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26591_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of MOR and Nb6
| 全体 | 名称: Complex of MOR and Nb6 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of MOR and Nb6
| 超分子 | 名称: Complex of MOR and Nb6 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: MOR
| 超分子 | 名称: MOR / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Mb6
| 超分子 | 名称: Mb6 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Mu-type opioid receptor
| 分子 | 名称: Mu-type opioid receptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 47.920734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTIIALSYI FCLVFADYKD DDDAMGPGNI SDCSDPLAPA SCSPAPGSWL NLSHVDGNQS DPCGPNRTGL GGSHSLCPQT GSPSMVTAI TIMALYSIVC VVGLFGNFLV MYVIVRYTKM KTATNIYIFN LALADALATS TLPFQSVNYL MGTWPFGNIL C KIVISIDY ...文字列: MKTIIALSYI FCLVFADYKD DDDAMGPGNI SDCSDPLAPA SCSPAPGSWL NLSHVDGNQS DPCGPNRTGL GGSHSLCPQT GSPSMVTAI TIMALYSIVC VVGLFGNFLV MYVIVRYTKM KTATNIYIFN LALADALATS TLPFQSVNYL MGTWPFGNIL C KIVISIDY YNMFTSIFTL CTMSVDRYIA VCHPVKALDF RTPRNAKIVN VCNWILSSAI GLPVMFMATT KYRQGSIDCT LT FSHPTWY WENLLKICVF IFAFIMPVLI ITVCYGLMIL RLKSVRLLSG SREKDRNLRR ITRMVLVVVA VFIVCWTPIH IYV IIKALI TIPETTFQTV SWHFCIALGY TNSCLNPVLY AFLDENFKRC FREFCIPTSS TIEQQNSARI RQNTREHPST ANTV DRTNH QLENLEAETA PLPDIHHHHH H UniProtKB: Mu-type opioid receptor |
-分子 #2: Megabody 6
| 分子 | 名称: Megabody 6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 55.662078 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQESGGG LVRKTTTSVI DTTNDAQNLL TQAQTIVNTL KDYCPILIAK SSSSNGGTNN ANTPSWQTAG GGKNSCATFG AEFSAASDM INNAQKIVQE TQQLSANQPK NITQPHNLNL NSPSSLTALA QKMLKNAQSQ AEILKLANQV ESDFNKLSSG H LKDYIGKC ...文字列: QVQLQESGGG LVRKTTTSVI DTTNDAQNLL TQAQTIVNTL KDYCPILIAK SSSSNGGTNN ANTPSWQTAG GGKNSCATFG AEFSAASDM INNAQKIVQE TQQLSANQPK NITQPHNLNL NSPSSLTALA QKMLKNAQSQ AEILKLANQV ESDFNKLSSG H LKDYIGKC DASAISSANM TMQNQKNNWG NGCAGVEETQ SLLKTSAADF NNQTPQINQA QNLANTLIQE LGNNTYEQLS RL LTNDNGT NSKTSAQAIN QAVNNLNERA KTLAGGTTNS PAYQATLLAL RSVLGLWNSM GYAVICGGYT KSPGENNQKD FHY TDENGN GTTINCGGST NSNGTHSYNG TNTLKADKNV SLSIEQYEKI HEAYQILSKA LKQAGLAPLN SKGEKLEAHV TTSK PSLRL SCAASGTIFR LYDMGWYRRV SGNQRELVAS ITSGGSTKYG DSVKGRFTIS RDNAKNTVYL QMSSLKPEDT AVYYC NAEY RTGIWEELLD GWGQGTQVTV SSHHHHHHEP EA |
-分子 #3: N-[(2S)-2-{[(3R,4R)-4-(3-hydroxyphenyl)-3,4-dimethylpiperidin-1-y...
| 分子 | 名称: N-[(2S)-2-{[(3R,4R)-4-(3-hydroxyphenyl)-3,4-dimethylpiperidin-1-yl]methyl}-3-phenylpropanoyl]glycine タイプ: ligand / ID: 3 / コピー数: 1 / 式: NG0 |
|---|---|
| 分子量 | 理論値: 424.533 Da |
| Chemical component information | 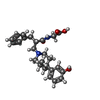 ChemComp-NG0: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 61.38 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 301332 |
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)












































