+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of G6PD-WT dimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | apo protein / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pentose biosynthetic process / ribose phosphate biosynthetic process / response to iron(III) ion / positive regulation of calcium ion transmembrane transport via high voltage-gated calcium channel / glucose-6-phosphate dehydrogenase (NADP+) / glucose-6-phosphate dehydrogenase activity / Pentose phosphate pathway / pentose-phosphate shunt, oxidative branch / negative regulation of cell growth involved in cardiac muscle cell development / NADPH regeneration ...pentose biosynthetic process / ribose phosphate biosynthetic process / response to iron(III) ion / positive regulation of calcium ion transmembrane transport via high voltage-gated calcium channel / glucose-6-phosphate dehydrogenase (NADP+) / glucose-6-phosphate dehydrogenase activity / Pentose phosphate pathway / pentose-phosphate shunt, oxidative branch / negative regulation of cell growth involved in cardiac muscle cell development / NADPH regeneration / glucose 6-phosphate metabolic process / NADP+ metabolic process / pentose-phosphate shunt / D-glucose binding / NFE2L2 regulates pentose phosphate pathway genes / response to food / erythrocyte maturation / cholesterol biosynthetic process / negative regulation of reactive oxygen species metabolic process / regulation of neuron apoptotic process / glutathione metabolic process / substantia nigra development / TP53 Regulates Metabolic Genes / lipid metabolic process / centriolar satellite / cytoplasmic side of plasma membrane / glucose metabolic process / NADP binding / cellular response to oxidative stress / response to ethanol / intracellular membrane-bounded organelle / protein homodimerization activity / extracellular exosome / identical protein binding / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Wei X / Marmorstein R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of G6PD-WT dimer 著者: Wei X / Marmorstein R | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26425.map.gz emd_26425.map.gz | 20.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26425-v30.xml emd-26425-v30.xml emd-26425.xml emd-26425.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26425.png emd_26425.png | 59.2 KB | ||
| Filedesc metadata |  emd-26425.cif.gz emd-26425.cif.gz | 5.6 KB | ||
| その他 |  emd_26425_half_map_1.map.gz emd_26425_half_map_1.map.gz emd_26425_half_map_2.map.gz emd_26425_half_map_2.map.gz | 20.6 MB 20.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26425 http://ftp.pdbj.org/pub/emdb/structures/EMD-26425 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26425 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26425 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26425_validation.pdf.gz emd_26425_validation.pdf.gz | 815.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26425_full_validation.pdf.gz emd_26425_full_validation.pdf.gz | 815.2 KB | 表示 | |
| XML形式データ |  emd_26425_validation.xml.gz emd_26425_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_26425_validation.cif.gz emd_26425_validation.cif.gz | 11.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26425 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26425 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26425 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26425 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uagMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26425.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26425.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_26425_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
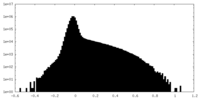
| 投影像・断面図 |
| ||||||||||||
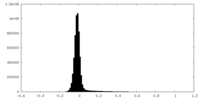
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26425_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
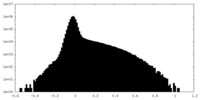
| 投影像・断面図 |
| ||||||||||||
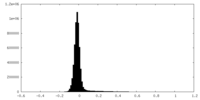
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : G6PD protein
| 全体 | 名称: G6PD protein |
|---|---|
| 要素 |
|
-超分子 #1: G6PD protein
| 超分子 | 名称: G6PD protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 110 KDa |
-分子 #1: Glucose-6-phosphate 1-dehydrogenase
| 分子 | 名称: Glucose-6-phosphate 1-dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: glucose-6-phosphate dehydrogenase (NADP+) |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 60.403754 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAEQVALSRT QVCGILREEL FQGDAFHQSD THIFIIMGAS GDLAKKKIYP TIWWLFRDGL LPENTFIVGY ARSRLTVADI RKQSEPFFK ATPEEKLKLE DFFARNSYVA GQYDDAASYQ RLNSHMNALH LGSQANRLFY LALPPTVYEA VTKNIHESCM S QIGWNRII ...文字列: MAEQVALSRT QVCGILREEL FQGDAFHQSD THIFIIMGAS GDLAKKKIYP TIWWLFRDGL LPENTFIVGY ARSRLTVADI RKQSEPFFK ATPEEKLKLE DFFARNSYVA GQYDDAASYQ RLNSHMNALH LGSQANRLFY LALPPTVYEA VTKNIHESCM S QIGWNRII VEKPFGRDLQ SSDRLSNHIS SLFREDQIYR IDHYLGKEMV QNLMVLRFAN RIFGPIWNRD NIACVILTFK EP FGTEGRG GYFDEFGIIR DVMQNHLLQM LCLVAMEKPA STNSDDVRDE KVKVLKCISE VQANNVVLGQ YVGNPDGEGE ATK GYLDDP TVPRGSTTAT FAAVVLYVEN ERWDGVPFIL RCGKALNERK AEVRLQFHDV AGDIFHQQCK RNELVIRVQP NEAV YTKMM TKKPGMFFNP EESELDLTYG NRYKNVKLPD AYERLILDVF CGSQMHFVRS DELREAWRIF TPLLHQIELE KPKPI PYIY GSRGPTEADE LMKRVGFQYE GTYKWVNPHK LLEHHHHHH UniProtKB: Glucose-6-phosphate 1-dehydrogenase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)