+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
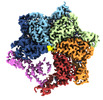
| タイトル | Skd3_ATPyS_FITC-casein Hexamer, AAA+ only | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cryoEM / AAA+ / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RIG-I signaling pathway / granulocyte differentiation / ATP-dependent protein disaggregase activity / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / antiviral innate immune response / mitochondrial intermembrane space / cellular response to heat / ATP hydrolysis activity / mitochondrion / extracellular region ...RIG-I signaling pathway / granulocyte differentiation / ATP-dependent protein disaggregase activity / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / antiviral innate immune response / mitochondrial intermembrane space / cellular response to heat / ATP hydrolysis activity / mitochondrion / extracellular region / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.96 Å | |||||||||
 データ登録者 データ登録者 | Rizo AN | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2022 ジャーナル: Cell Rep / 年: 2022タイトル: Unique structural features govern the activity of a human mitochondrial AAA+ disaggregase, Skd3. 著者: Ryan R Cupo / Alexandrea N Rizo / Gabriel A Braun / Eric Tse / Edward Chuang / Kushol Gupta / Daniel R Southworth / James Shorter /  要旨: The AAA+ protein, Skd3 (human CLPB), solubilizes proteins in the mitochondrial intermembrane space, which is critical for human health. Skd3 variants with defective protein-disaggregase activity ...The AAA+ protein, Skd3 (human CLPB), solubilizes proteins in the mitochondrial intermembrane space, which is critical for human health. Skd3 variants with defective protein-disaggregase activity cause severe congenital neutropenia (SCN) and 3-methylglutaconic aciduria type 7 (MGCA7). How Skd3 disaggregates proteins remains poorly understood. Here, we report a high-resolution structure of a Skd3-substrate complex. Skd3 adopts a spiral hexameric arrangement that engages substrate via pore-loop interactions in the nucleotide-binding domain (NBD). Substrate-bound Skd3 hexamers stack head-to-head via unique, adaptable ankyrin-repeat domain (ANK)-mediated interactions to form dodecamers. Deleting the ANK linker region reduces dodecamerization and disaggregase activity. We elucidate apomorphic features of the Skd3 NBD and C-terminal domain that regulate disaggregase activity. We also define how Skd3 subunits collaborate to disaggregate proteins. Importantly, SCN-linked subunits sharply inhibit disaggregase activity, whereas MGCA7-linked subunits do not. These advances illuminate Skd3 structure and mechanism, explain SCN and MGCA7 inheritance patterns, and suggest therapeutic strategies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26121.map.gz emd_26121.map.gz | 156.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26121-v30.xml emd-26121-v30.xml emd-26121.xml emd-26121.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26121.png emd_26121.png | 168.8 KB | ||
| Filedesc metadata |  emd-26121.cif.gz emd-26121.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26121 http://ftp.pdbj.org/pub/emdb/structures/EMD-26121 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26121 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26121 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26121_validation.pdf.gz emd_26121_validation.pdf.gz | 501.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26121_full_validation.pdf.gz emd_26121_full_validation.pdf.gz | 501 KB | 表示 | |
| XML形式データ |  emd_26121_validation.xml.gz emd_26121_validation.xml.gz | 6.9 KB | 表示 | |
| CIF形式データ |  emd_26121_validation.cif.gz emd_26121_validation.cif.gz | 7.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26121 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26121 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26121 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26121 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ttrMC  7ttsC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26121.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26121.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Skd3 complex bound to FITC-casein
| 全体 | 名称: Skd3 complex bound to FITC-casein |
|---|---|
| 要素 |
|
-超分子 #1: Skd3 complex bound to FITC-casein
| 超分子 | 名称: Skd3 complex bound to FITC-casein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-分子 #1: Caseinolytic peptidase B protein homolog
| 分子 | 名称: Caseinolytic peptidase B protein homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 66.115047 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GGSYSKSPSN KDAALLEAAR ANNMQEVSRL LSEGADVNAK HRLGWTALMV AAINRNNSVV QVLLAAGADP NLGDDFSSVY KTAKEQGIH SLEDGGQDGA SRHITNQWTS ALEFRRWLGL PAGVLITRED DFNNRLNNRA SFKGCTALHY AVLADDYRTV K ELLDGGAN ...文字列: GGSYSKSPSN KDAALLEAAR ANNMQEVSRL LSEGADVNAK HRLGWTALMV AAINRNNSVV QVLLAAGADP NLGDDFSSVY KTAKEQGIH SLEDGGQDGA SRHITNQWTS ALEFRRWLGL PAGVLITRED DFNNRLNNRA SFKGCTALHY AVLADDYRTV K ELLDGGAN PLQRNEMGHT PLDYAREGEV MKLLRTSEAK YQEKQRKREA EERRRFPLEQ RLKEHIIGQE SAIATVGAAI RR KENGWYD EEHPLVFLFL GSSGIGKTEL AKQTAKYMHK DAKKGFIRLD MSEFQERHEV AKFIGSPPGY VGHEEGGQLT KKL KQCPNA VVLFDEVDKA HPDVLTIMLQ LFDEGRLTDG KGKTIDCKDA IFIMTSNVAS DEIAQHALQL RQEALEMSRN RIAE NLGDV QISDKITISK NFKENVIRPI LKAHFRRDEF LGRINEIVYF LPFCHSELIQ LVNKELNFWA KRAKQRHNIT LLWDR EVAD VLVDGYNVHY GARSIKHEVE RRVVNQLAAA YEQDLLPGGC TLRITVEDSD KQLLKSPELP SPQAEKRLPK LRLEII DKD SKTRRLDIRA PLHPEKVCNT I UniProtKB: Mitochondrial disaggregase |
-分子 #2: Beta-casein
| 分子 | 名称: Beta-casein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.759648 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)ARELEE LNVP GEIVE SLSSSEESIT RINKKIEKFQ SEEQQQTEDE LQDKIHPFAQ TQSLVYPFPG PIPNSLPQNI PPLTQTPVVV PPFLQ PEVM GVSKVKGAMA ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)ARELEE LNVP GEIVE SLSSSEESIT RINKKIEKFQ SEEQQQTEDE LQDKIHPFAQ TQSLVYPFPG PIPNSLPQNI PPLTQTPVVV PPFLQ PEVM GVSKVKGAMA PKHKEMPFPK YPVEPLTESQ SLTLTDVENL HLPLPLLQSW MHQPHQPLPP TVMFPPQSVL SLSQSK VLP VPQKAVPYPQ RDMPIQAFLL YQEPVLGPVR GPFPIIV UniProtKB: Beta-casein |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 4 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 68.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















