登録情報 データベース : EMDB / ID : EMD-25647タイトル Structure of electron bifurcating Ni-Fe hydrogenase complex HydABCSL in FMN/NAD(H) bound state Electron bifurcating Ni-Fe hydrogenase complex HydABCSL in FMN/NAD(H) bound state 複合体 : NiFe hydrogenase complex ABCSLリガンド : x 6種 / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
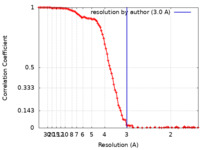
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Acetomicrobium mobile (バクテリア)手法 / / 解像度 : 3.0 Å Feng X / Li H 資金援助 Organization Grant number 国 Department of Energy (DOE, United States) DE-SC0020085 Department of Energy (DOE, United States) DE-FG02-95ER20175
ジャーナル : Sci Adv / 年 : 2022タイトル : Structure and electron transfer pathways of an electron-bifurcating NiFe-hydrogenase.著者 : Xiang Feng / Gerrit J Schut / Dominik K Haja / Michael W W Adams / Huilin Li / 要旨 : Electron bifurcation enables thermodynamically unfavorable biochemical reactions. Four groups of bifurcating flavoenzyme are known and three use FAD to bifurcate. FeFe-HydABC hydrogenase represents ... Electron bifurcation enables thermodynamically unfavorable biochemical reactions. Four groups of bifurcating flavoenzyme are known and three use FAD to bifurcate. FeFe-HydABC hydrogenase represents the fourth group, but its bifurcation site is unknown. We report cryo-EM structures of the related NiFe-HydABCSL hydrogenase that reversibly oxidizes H and couples endergonic reduction of ferredoxin with exergonic reduction of NAD. FMN surrounded by a unique arrangement of iron sulfur clusters forms the bifurcating center. NAD binds to FMN in HydB, and electrons from H via HydA to a HydB [4Fe-4S] cluster enable the FMN to reduce NAD. Low-potential electron transfer from FMN to the HydC [2Fe-2S] cluster and subsequent reduction of a uniquely penta-coordinated HydB [2Fe-2S] cluster require conformational changes, leading to ferredoxin binding and reduction by a [4Fe-4S] cluster in HydB. This work clarifies the electron transfer pathways for a large group of hydrogenases underlying many essential functions in anaerobic microorganisms. 履歴 登録 2021年12月6日 - ヘッダ(付随情報) 公開 2022年3月16日 - マップ公開 2022年3月16日 - 更新 2024年12月25日 - 現状 2024年12月25日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Acetomicrobium mobile (バクテリア)
Acetomicrobium mobile (バクテリア) データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Sci Adv / 年: 2022
ジャーナル: Sci Adv / 年: 2022
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_25647.map.gz
emd_25647.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-25647-v30.xml
emd-25647-v30.xml emd-25647.xml
emd-25647.xml EMDBヘッダ
EMDBヘッダ emd_25647_fsc.xml
emd_25647_fsc.xml FSCデータファイル
FSCデータファイル emd_25647.png
emd_25647.png emd-25647.cif.gz
emd-25647.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-25647
http://ftp.pdbj.org/pub/emdb/structures/EMD-25647 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25647
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25647 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_25647.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_25647.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)