+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Kinetically trapped Pseudomonas-phage PaP3 portal protein - delta barrel mutant class-2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Portal protein / Dodecamer / VIRAL PROTEIN | |||||||||
| 機能・相同性 | : / Phage SU10 portal protein / ORF.04 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Phage PaP3 (ファージ) / Phage PaP3 (ファージ) /  Pseudomonas virus PaP3 (ウイルス) Pseudomonas virus PaP3 (ウイルス) | |||||||||
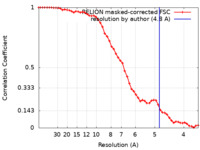
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Hou C-FD / Swanson NA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2022 ジャーナル: J Mol Biol / 年: 2022タイトル: Cryo-EM Structure of a Kinetically Trapped Dodecameric Portal Protein from the Pseudomonas-phage PaP3. 著者: Chun-Feng David Hou / Nicholas A Swanson / Fenglin Li / Ruoyu Yang / Ravi K Lokareddy / Gino Cingolani /  要旨: Portal proteins are dodecameric assemblies that occupy a unique 5-fold vertex of the icosahedral capsid of tailed bacteriophages and herpesviruses. The portal vertex interrupts the icosahedral ...Portal proteins are dodecameric assemblies that occupy a unique 5-fold vertex of the icosahedral capsid of tailed bacteriophages and herpesviruses. The portal vertex interrupts the icosahedral symmetry, and in vivo, its assembly and incorporation in procapsid are controlled by the scaffolding protein. Ectopically expressed portal oligomers are polymorphic in solution, and portal rings built by a different number of subunits have been documented in the literature. In this paper, we describe the cryo-EM structure of the portal protein from the Pseudomonas-phage PaP3, which we determined at 3.4 Å resolution. Structural analysis revealed a dodecamer with helical rather than rotational symmetry, which we hypothesize is kinetically trapped. The helical assembly was stabilized by local mispairing of portal subunits caused by the slippage of crown and barrel helices that move like a lever with respect to the portal body. Removing the C-terminal barrel promoted assembly of undecameric and dodecameric rings with quasi-rotational symmetry, suggesting that the barrel contributes to subunits mispairing. However, ΔC-portal rings were intrinsically asymmetric, with most particles having one open portal subunit interface. Together, these data expand the structural repertoire of viral portal proteins to Pseudomonas-phages and shed light on the unexpected plasticity of the portal protein quaternary structure. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25560.map.gz emd_25560.map.gz | 8.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25560-v30.xml emd-25560-v30.xml emd-25560.xml emd-25560.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25560_fsc.xml emd_25560_fsc.xml | 5.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25560.png emd_25560.png | 54.3 KB | ||
| マスクデータ |  emd_25560_msk_1.map emd_25560_msk_1.map | 11.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25560.cif.gz emd-25560.cif.gz | 6.3 KB | ||
| その他 |  emd_25560_half_map_1.map.gz emd_25560_half_map_1.map.gz emd_25560_half_map_2.map.gz emd_25560_half_map_2.map.gz | 8.6 MB 8.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25560 http://ftp.pdbj.org/pub/emdb/structures/EMD-25560 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25560 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25560 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25560_validation.pdf.gz emd_25560_validation.pdf.gz | 814.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25560_full_validation.pdf.gz emd_25560_full_validation.pdf.gz | 814.1 KB | 表示 | |
| XML形式データ |  emd_25560_validation.xml.gz emd_25560_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_25560_validation.cif.gz emd_25560_validation.cif.gz | 14.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25560 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25560 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25560 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25560 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25560.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25560.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
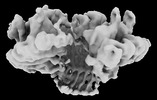
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.82 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25560_msk_1.map emd_25560_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_25560_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_25560_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dodecameric complex of phage PaP3 portal, C-terminal truncated mutant
| 全体 | 名称: Dodecameric complex of phage PaP3 portal, C-terminal truncated mutant |
|---|---|
| 要素 |
|
-超分子 #1: Dodecameric complex of phage PaP3 portal, C-terminal truncated mutant
| 超分子 | 名称: Dodecameric complex of phage PaP3 portal, C-terminal truncated mutant タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Phage PaP3 (ファージ) Phage PaP3 (ファージ) |
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas virus PaP3 (ウイルス) Pseudomonas virus PaP3 (ウイルス) |
| 分子量 | 理論値: 80.981344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKRRKIKPM DDEQVLRHLD QLVNDALDFN SSELSKQRSE ALKYYFGEPF GNERPGKSGI VSRDVQETVD WIMPSLMKVF TSGGQVVKY EPDTAEDVEQ AEQETEYVNY LFMRKNEGFK VMFDWFQDTL MMKTGVVKVY VEEVLKPTFE RFSGLSEDMV A DILSDPDT ...文字列: MAKRRKIKPM DDEQVLRHLD QLVNDALDFN SSELSKQRSE ALKYYFGEPF GNERPGKSGI VSRDVQETVD WIMPSLMKVF TSGGQVVKY EPDTAEDVEQ AEQETEYVNY LFMRKNEGFK VMFDWFQDTL MMKTGVVKVY VEEVLKPTFE RFSGLSEDMV A DILSDPDT SILAQSVDDD GTYTIKIRKD KKKREIKVLC VKPENFLVDR LATCIDDARF LCHREKYTVS DLRLLGVPED VI EELPYDE YEFSDSQPER LVRDNFDMTG QLQYNSGDDA EANREVWASE CYTLLDVDGD GISELRRILY VGDYIISNEP WDC RPFADL NAYRIAHKFH GMSVYDKIRD IQEIRSVLMR NIMDNIYRTN QGRSVVLDGQ VNLEDLLTNE AAGIVRVKSM NSIT PLETP QLSGEVYGML DRLEADRGKR TGITDRTRGL DQNTLHSNQA AMSVNQLMTA AEQQIDLIAR MFAETGVKRL FQLLH DHAI KYQNQEEVFQ LRGKWVAVNP ANWRERSDLT VTVGIGNMNK DQQMLHLMRI WEMAQAVVGG GGLGVLVSEQ NLYNIL KEV TENAGYKDPD RFWTNPNSPE ALQAKAIREQ KEAQPKPEDI KAQADAQRAQ SDALAKQAEA QMKQVEAQIR LAEIELK KQ EAVLQQREMA LKEAELQLER DRFTWERARN EAEYHLEATQ ARAAYIGDGK VPETKKPTKA VRR UniProtKB: ORF.04 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 150000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL / 温度因子: 129 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7sz4: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)