+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of 7SK core RNP with circular RNA | |||||||||||||||
 Map data Map data | 7SK core RNP with circular RNA | |||||||||||||||
 Sample Sample |
| |||||||||||||||
 Keywords Keywords | non-coding RNA / La-related protein / methylphosphate capping enzyme / transcription regulation / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||||||||
| Function / homology |  Function and homology information Function and homology informationRNA 5'-gamma-phosphate methyltransferase activity / U6 2'-O-snRNA methylation / snRNA metabolic process / snRNA modification / 7SK snRNP / positive regulation of snRNA transcription by RNA polymerase II / 7SK snRNA binding / positive regulation of protein localization to Cajal body / RNA methylation / RNA methyltransferase activity ...RNA 5'-gamma-phosphate methyltransferase activity / U6 2'-O-snRNA methylation / snRNA metabolic process / snRNA modification / 7SK snRNP / positive regulation of snRNA transcription by RNA polymerase II / 7SK snRNA binding / positive regulation of protein localization to Cajal body / RNA methylation / RNA methyltransferase activity / snRNA binding / box C/D sno(s)RNA 3'-end processing / negative regulation of viral transcription / S-adenosylmethionine-dependent methyltransferase activity / regulation of mRNA splicing, via spliceosome / O-methyltransferase activity / negative regulation of transcription elongation by RNA polymerase II / positive regulation of G1/S transition of mitotic cell cycle / U6 snRNA binding / RNA splicing / Transferases; Transferring one-carbon groups; Methyltransferases / mRNA processing / spermatogenesis / cell differentiation / ribonucleoprotein complex / negative regulation of transcription by RNA polymerase II / RNA binding / nucleoplasm / nucleus / cytosol Similarity search - Function | |||||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.7 Å | |||||||||||||||
 Authors Authors | Yang Y / Liu S | |||||||||||||||
| Funding support |  United States, 4 items United States, 4 items
| |||||||||||||||
 Citation Citation |  Journal: Mol Cell / Year: 2022 Journal: Mol Cell / Year: 2022Title: Structural basis of RNA conformational switching in the transcriptional regulator 7SK RNP. Authors: Yuan Yang / Shiheng Liu / Sylvain Egloff / Catherine D Eichhorn / Tanya Hadjian / James Zhen / Tamás Kiss / Z Hong Zhou / Juli Feigon /    Abstract: 7SK non-coding RNA (7SK) negatively regulates RNA polymerase II (RNA Pol II) elongation by inhibiting positive transcription elongation factor b (P-TEFb), and its ribonucleoprotein complex (RNP) is ...7SK non-coding RNA (7SK) negatively regulates RNA polymerase II (RNA Pol II) elongation by inhibiting positive transcription elongation factor b (P-TEFb), and its ribonucleoprotein complex (RNP) is hijacked by HIV-1 for viral transcription and replication. Methylphosphate capping enzyme (MePCE) and La-related protein 7 (Larp7) constitutively associate with 7SK to form a core RNP, while P-TEFb and other proteins dynamically assemble to form different complexes. Here, we present the cryo-EM structures of 7SK core RNP formed with two 7SK conformations, circular and linear, and uncover a common RNA-dependent MePCE-Larp7 complex. Together with NMR, biochemical, and cellular data, these structures reveal the mechanism of MePCE catalytic inactivation in the core RNP, unexpected interactions between Larp7 and RNA that facilitate a role as an RNP chaperone, and that MePCE-7SK-Larp7 core RNP serves as a scaffold for switching between different 7SK conformations essential for RNP assembly and regulation of P-TEFb sequestration and release. | |||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_25198.map.gz emd_25198.map.gz | 28.4 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-25198-v30.xml emd-25198-v30.xml emd-25198.xml emd-25198.xml | 18.9 KB 18.9 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_25198.png emd_25198.png | 98.8 KB | ||
| Filedesc metadata |  emd-25198.cif.gz emd-25198.cif.gz | 7 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25198 http://ftp.pdbj.org/pub/emdb/structures/EMD-25198 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25198 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25198 | HTTPS FTP |
-Related structure data
| Related structure data |  7slqMC  7slpC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_25198.map.gz / Format: CCP4 / Size: 30.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_25198.map.gz / Format: CCP4 / Size: 30.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | 7SK core RNP with circular RNA | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
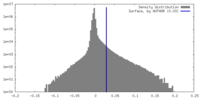
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
- Sample components
Sample components
-Entire : 7SK circular core RNP with MePCE, Larp7 and circular 7SK RNA
| Entire | Name: 7SK circular core RNP with MePCE, Larp7 and circular 7SK RNA |
|---|---|
| Components |
|
-Supramolecule #1: 7SK circular core RNP with MePCE, Larp7 and circular 7SK RNA
| Supramolecule | Name: 7SK circular core RNP with MePCE, Larp7 and circular 7SK RNA type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#3 |
|---|
-Supramolecule #2: 7SK snRNA methylphosphate capping enzyme, La-related protein 7
| Supramolecule | Name: 7SK snRNA methylphosphate capping enzyme, La-related protein 7 type: complex / ID: 2 / Parent: 1 / Macromolecule list: #1-#2 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
-Supramolecule #3: RNA
| Supramolecule | Name: RNA / type: complex / ID: 3 / Parent: 1 / Macromolecule list: #3 |
|---|
-Macromolecule #1: 7SK snRNA methylphosphate capping enzyme
| Macromolecule | Name: 7SK snRNA methylphosphate capping enzyme / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO EC number: Transferases; Transferring one-carbon groups; Methyltransferases |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 35.122797 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MGSSHHHHHH SSGLVPRGSP LPAAGFKKQQ RKFQYGNYCK YYGYRNPSCE DGRLRVLKPE WFRGRDVLDL GCNVGHLTLS IACKWGPSR MVGLDIDSRL IHSARQNIRH YLSEELRLPP QTLEGDPGAE GEEGTTTVRK RSCFPASLTA SRGPIAAPQV P LDGADTSV ...String: MGSSHHHHHH SSGLVPRGSP LPAAGFKKQQ RKFQYGNYCK YYGYRNPSCE DGRLRVLKPE WFRGRDVLDL GCNVGHLTLS IACKWGPSR MVGLDIDSRL IHSARQNIRH YLSEELRLPP QTLEGDPGAE GEEGTTTVRK RSCFPASLTA SRGPIAAPQV P LDGADTSV FPNNVVFVTG NYVLDRDDLV EAQTPEYDVV LCLSLTKWVH LNWGDEGLKR MFRRIYRHLR PGGILVLEPQ PW SSYGKRK TLTETIYKNY YRIQLKPEQF SSYLTSPDVG FSSYELVATP HNTSKGFQRP VYLFHKARSP SH UniProtKB: 7SK snRNA methylphosphate capping enzyme |
-Macromolecule #2: La-related protein 7
| Macromolecule | Name: La-related protein 7 / type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 55.109129 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GMETESGNQE KVMEEESTEK KKEVEKKKRS RVKQVLADIA KQVDFWFGDA NLHKDRFLRE QIEKSRDGYV DISLLVSFNK MKKLTTDGK LIARALRSSA VVELDLEGTR IRRKKPLGER PKDEDERTVY VELLPKNVNH SWIERVFGKC GNVVYISIPH Y KSTGDPKG ...String: GMETESGNQE KVMEEESTEK KKEVEKKKRS RVKQVLADIA KQVDFWFGDA NLHKDRFLRE QIEKSRDGYV DISLLVSFNK MKKLTTDGK LIARALRSSA VVELDLEGTR IRRKKPLGER PKDEDERTVY VELLPKNVNH SWIERVFGKC GNVVYISIPH Y KSTGDPKG FAFVEFETKE QAAKAIEFLN NPPEEAPRKP GIFPKTVKNK PIPALRVVEE KKKKKKKKGR MKKEDNIQAK EE NMDTSNT SISKMKRSRP TSEGSDIEST GEEVIPLRVL SKSEWMDLKK EYLALQKASM ASLKKTISQI KSESEMETDS GVP QNTGMK NEKTANREEC RTQEKVNATG PQFVSGVIVK IISTEPLPGR KQVRDTLAAI SEVLYVDLLE GDTECHARFK TPED AQAVI NAYTEINKKH CWKLEILSGD HEQRYWQKIL VDRQAKLNQP REKKRGTEKL ITKAEKIRLA KTQQASKHIR FSEYD UniProtKB: La-related protein 7 |
-Macromolecule #3: Minimal circular 7SK RNA
| Macromolecule | Name: Minimal circular 7SK RNA / type: rna / ID: 3 / Number of copies: 1 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 21.032305 KDa |
| Sequence | String: (G5J)GAUGUGAGG CUUCGGCCAG ACACAUCCAA AUGAGGCGCU GCAUGUGGCA GUCUGCCUUU CUUUU |
-Macromolecule #4: S-ADENOSYL-L-HOMOCYSTEINE
| Macromolecule | Name: S-ADENOSYL-L-HOMOCYSTEINE / type: ligand / ID: 4 / Number of copies: 1 / Formula: SAH |
|---|---|
| Molecular weight | Theoretical: 384.411 Da |
| Chemical component information |  ChemComp-SAH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Grid | Model: Quantifoil R2/1 / Material: COPPER / Mesh: 300 / Pretreatment - Type: PLASMA CLEANING / Pretreatment - Time: 20 sec. / Pretreatment - Atmosphere: OTHER / Pretreatment - Pressure: 0.00933 kPa |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 283.15 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Detector mode: SUPER-RESOLUTION / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal magnification: 130000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)