[日本語] English
 万見
万見- EMDB-24785: Cryo-EM structure of human GlcNAc-1-phosphotransferase A2B2 subcomplex -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human GlcNAc-1-phosphotransferase A2B2 subcomplex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報UDP-N-acetylglucosamine-lysosomal-enzyme N-acetylglucosaminephosphotransferase / N-glycan processing to lysosome / secretion of lysosomal enzymes / UDP-N-acetylglucosamine-lysosomal-enzyme N-acetylglucosaminephosphotransferase activity / carbohydrate phosphorylation / lysosome organization / Golgi membrane / calcium ion binding / Golgi apparatus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
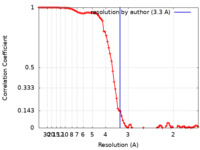
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 | ジャーナル: Nat Struct Mol Biol / 年: 2022 タイトル: Bound nucleotide can control the dynamic architecture of monomeric actin. 著者: Rustam Ali / Jacob A Zahm / Michael K Rosen /  要旨: Polymerization of actin into cytoskeletal filaments is coupled to its bound adenine nucleotides. The mechanism by which nucleotide modulates actin functions has not been evident from analyses of ATP- ...Polymerization of actin into cytoskeletal filaments is coupled to its bound adenine nucleotides. The mechanism by which nucleotide modulates actin functions has not been evident from analyses of ATP- and ADP-bound crystal structures of the actin monomer. We report that NMR chemical shift differences between the two forms are globally distributed. Furthermore, microsecond-millisecond motions are spread throughout the molecule in the ATP form, but largely confined to subdomains 1 and 2, and the nucleotide binding site in the ADP form. Through these motions, the ATP- and ADP-bound forms sample different high-energy conformations. A deafness-causing, fast-nucleating actin mutant populates the high-energy conformer of ATP-actin more than the wild-type protein, suggesting that this conformer may be on the pathway to nucleation. Together, the data suggest a model in which differential sampling of a nucleation-compatible form of the actin monomer may contribute to control of actin filament dynamics by nucleotide. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24785.map.gz emd_24785.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24785-v30.xml emd-24785-v30.xml emd-24785.xml emd-24785.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24785_fsc.xml emd_24785_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24785.png emd_24785.png | 110.8 KB | ||
| その他 |  emd_24785_half_map_1.map.gz emd_24785_half_map_1.map.gz emd_24785_half_map_2.map.gz emd_24785_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24785 http://ftp.pdbj.org/pub/emdb/structures/EMD-24785 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24785 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24785 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24785_validation.pdf.gz emd_24785_validation.pdf.gz | 756.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24785_full_validation.pdf.gz emd_24785_full_validation.pdf.gz | 756.5 KB | 表示 | |
| XML形式データ |  emd_24785_validation.xml.gz emd_24785_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_24785_validation.cif.gz emd_24785_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24785 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24785 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24785 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24785 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7s06MC  7s05C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24785.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24785.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_24785_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
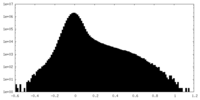
| 投影像・断面図 |
| ||||||||||||
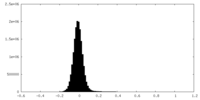
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_24785_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
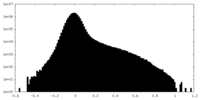
| 投影像・断面図 |
| ||||||||||||
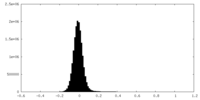
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GlcNAc-1-phosphotransferase
| 全体 | 名称: GlcNAc-1-phosphotransferase |
|---|---|
| 要素 |
|
-超分子 #1: GlcNAc-1-phosphotransferase
| 超分子 | 名称: GlcNAc-1-phosphotransferase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
-分子 #1: N-acetylglucosamine-1-phosphotransferase subunits alpha/beta
| 分子 | 名称: N-acetylglucosamine-1-phosphotransferase subunits alpha/beta タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: UDP-N-acetylglucosamine-lysosomal-enzyme N-acetylglucosaminephosphotransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 134.7875 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DEDQVDPRLI DGKWSRDQYH VLFDSYRDNI AGKSFQNRLC LPMPIDVVYT WVNGTDLELL KELQQVREQM EEEQKAMREI LGKNTTEPT KKSEKQLECL LTHCIKVPML VLDPALPANI TLKDLPSLYP SFHSASDIFN VAKPKNPSTN VSVVVFDSTK D VEDAHSGL ...文字列: DEDQVDPRLI DGKWSRDQYH VLFDSYRDNI AGKSFQNRLC LPMPIDVVYT WVNGTDLELL KELQQVREQM EEEQKAMREI LGKNTTEPT KKSEKQLECL LTHCIKVPML VLDPALPANI TLKDLPSLYP SFHSASDIFN VAKPKNPSTN VSVVVFDSTK D VEDAHSGL LKGNSRQTVW RGYLTTDKEV PGLVLMQDLA FLSGFPPTFK ETNQLKTKLP ENLSSKVKLL QLYSEASVAL LK LNNPKDF QELNKQTKKN MTIDGKELTI SPAYLLWDLS AISQSKQDED ISASRFEDNE ELRYSLRSIE RHAPWVRNIF IVT NGQIPS WLNLDNPRVT IVTHQDVFRN LSHLPTFSSP AIESHIHRIE GLSQKFIYLN DDVMFGKDVW PDDFYSHSKG QKVY LTWPV PNCAEGCPGS WIKDGYCDKA CNNSACDWDG GDCSGNSGGS RYIAGGGGTG SIGVGQPWQF GGGINSVSYC NQGCA NSWL ADKFCDQACN VLSCGFDAGD CGQDHFHELY KVILLPNQTH YIIPKGECLP YFSFAEVAKR GVEGAYSDNP IIRHAS IAN KWKTIHLIMH SGMNATTIHF NLTFQNTNDE EFKMQITVEV DTREGPKLNS TAQKGYENLV SPITLLPEAE ILFEDIP KE KRFPKFKRHD VNSTRRAQEE VKIPLVNISL LPKDAQLSLN TLDLQLEHGD ITLKGYNLSK SALLRSFLMN SQHAKIKN Q AIITDETNDS LVAPQEKQVH KSILPNSLGV SERLQRLTFP AVSVKVNGHD QGQNPPLDLE TTARFRVETH TQKTIGGNV TKEKPPSLIV PLESQMTKEK KITGKEKENS RMEENAENHI GVTEVLLGRK LQHYTDSYLG FLPWEKKKYF QDLLDEEESL KTQLAYFTD SKNRARYKRD TFADSLRYVN KILNSKFGFT SRKVPAHMPH MIDRIVMQEL QDMFPEEFDK TSFHKVRHSE D MQFAFSYF YYLMSAVQPL NISQVFDEVD TDQSGVLSDR EIRTLATRIH ELPLSLQDLT GLEHMLINCS KMLPADITQL NN IPPTQES YYDPNLPPVT KSLVTNCKPV TDKIHKAYKD KNKYRFEIMG EEEIAFKMIR TNVSHVVGQL DDIRKNPRKF VCL NDNIDH NHKDAQTVKA VLRDFYESMF PIPSQFELPR EYRNRFLHMH ELQEWRAYRD KLK |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 299 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 193.0 K / 最高: 193.0 K |
| アライメント法 | Coma free - Residual tilt: 0.05 mrad |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 13320 / 平均露光時間: 1.5 sec. / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7s06: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X