+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24428 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - apo class | |||||||||
 マップデータ マップデータ | local resolution filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA-dependent RNA polymerase / viral replication-transcription complex / transcription / viral proteins / REPLICATION-TRANSCRIPTION complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein guanylyltransferase activity / RNA endonuclease activity producing 3'-phosphomonoesters, hydrolytic mechanism / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / Maturation of replicase proteins / TRAF3-dependent IRF activation pathway / ISG15-specific peptidase activity ...protein guanylyltransferase activity / RNA endonuclease activity producing 3'-phosphomonoesters, hydrolytic mechanism / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / Maturation of replicase proteins / TRAF3-dependent IRF activation pathway / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / snRNP Assembly / Translation of Replicase and Assembly of the Replication Transcription Complex / Replication of the SARS-CoV-2 genome / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ / double membrane vesicle viral factory outer membrane / host cell endoplasmic reticulum-Golgi intermediate compartment / SARS coronavirus main proteinase / 5'-3' DNA helicase activity / 3'-5'-RNA exonuclease activity / host cell endosome / symbiont-mediated degradation of host mRNA / mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / omega peptidase activity / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / SARS-CoV-2 modulates host translation machinery / host cell Golgi apparatus / symbiont-mediated suppression of host NF-kappaB cascade / symbiont-mediated perturbation of host ubiquitin-like protein modification / DNA helicase / methyltransferase cap1 activity / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ / single-stranded RNA binding / regulation of autophagy / viral protein processing / lyase activity / host cell perinuclear region of cytoplasm / host cell endoplasmic reticulum membrane / RNA helicase / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / symbiont-mediated suppression of host gene expression / copper ion binding / viral translational frameshifting / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / lipid binding / host cell nucleus / SARS-CoV-2 activates/modulates innate and adaptive immune responses / ATP hydrolysis activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
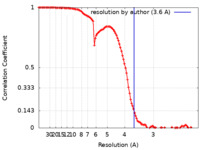
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Chen J / Malone B | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Ensemble cryo-EM reveals conformational states of the nsp13 helicase in the SARS-CoV-2 helicase replication-transcription complex. 著者: James Chen / Qi Wang / Brandon Malone / Eliza Llewellyn / Yakov Pechersky / Kashyap Maruthi / Ed T Eng / Jason K Perry / Elizabeth A Campbell / David E Shaw / Seth A Darst /  要旨: The SARS-CoV-2 nonstructural proteins coordinate genome replication and gene expression. Structural analyses revealed the basis for coupling of the essential nsp13 helicase with the RNA-dependent RNA ...The SARS-CoV-2 nonstructural proteins coordinate genome replication and gene expression. Structural analyses revealed the basis for coupling of the essential nsp13 helicase with the RNA-dependent RNA polymerase (RdRp) where the holo-RdRp and RNA substrate (the replication-transcription complex or RTC) associated with two copies of nsp13 (nsp13-RTC). One copy of nsp13 interacts with the template-RNA in an opposing polarity to the RdRp and is envisaged to drive the RdRp backward on the RNA template (backtracking), prompting questions as to how the RdRp can efficiently synthesize RNA in the presence of nsp13. Here we use cryogenic-electron microscopy and molecular dynamics simulations to analyze the nsp13-RTC, revealing four distinct conformational states of the helicases. The results indicate a mechanism for the nsp13-RTC to turn backtracking on and off, using an allosteric mechanism to switch between RNA synthesis or backtracking in response to stimuli at the RdRp active site. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: Ensemble cryo-electron microscopy reveals conformational states of the nsp13 helicase in the SARS-CoV-2 helicase replication-transcription complex 著者: Chen J / Wang Q / Malone B / Llewellyn E / Pechersky Y / Maruthi K / Eng ET / Perry JK / Campbell EA / Shaw DE / Darst SA | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24428.map.gz emd_24428.map.gz | 14.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24428-v30.xml emd-24428-v30.xml emd-24428.xml emd-24428.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24428_fsc.xml emd_24428_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24428.png emd_24428.png | 100.4 KB | ||
| Filedesc metadata |  emd-24428.cif.gz emd-24428.cif.gz | 7.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24428 http://ftp.pdbj.org/pub/emdb/structures/EMD-24428 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24428 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24428 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24428_validation.pdf.gz emd_24428_validation.pdf.gz | 386 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24428_full_validation.pdf.gz emd_24428_full_validation.pdf.gz | 385.5 KB | 表示 | |
| XML形式データ |  emd_24428_validation.xml.gz emd_24428_validation.xml.gz | 12.6 KB | 表示 | |
| CIF形式データ |  emd_24428_validation.cif.gz emd_24428_validation.cif.gz | 16.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24428 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24428 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24428 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24428 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24428.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24428.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution filtered map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : SARS-CoV-2 replication/transcription complex bound to nsp13 helic...
+超分子 #1: SARS-CoV-2 replication/transcription complex bound to nsp13 helic...
+分子 #1: RNA-directed RNA polymerase
+分子 #2: Non-structural protein 8
+分子 #3: Non-structural protein 7
+分子 #4: Helicase
+分子 #5: Product RNA
+分子 #6: Template RNA
+分子 #7: ZINC ION
+分子 #8: MAGNESIUM ION
+分子 #9: ADENOSINE-5'-DIPHOSPHATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


































 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)