+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human ternary NatC complex with a Bisubstrate inhibitor | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NatC / NAA30 / NAA35 / NAA38 / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報N-terminal methionine Nalpha-acetyltransferase NatC / NatC complex / smooth muscle cell proliferation / protein N-terminal-methionine acetyltransferase activity / Retrograde transport at the Trans-Golgi-Network / protein-N-terminal amino-acid acetyltransferase activity / protein stabilization / negative regulation of apoptotic process / RNA binding / nucleoplasm ...N-terminal methionine Nalpha-acetyltransferase NatC / NatC complex / smooth muscle cell proliferation / protein N-terminal-methionine acetyltransferase activity / Retrograde transport at the Trans-Golgi-Network / protein-N-terminal amino-acid acetyltransferase activity / protein stabilization / negative regulation of apoptotic process / RNA binding / nucleoplasm / nucleus / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.64 Å | |||||||||
 データ登録者 データ登録者 | Deng S / Marmorstein R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Molecular role of NAA38 in thermostability and catalytic activity of the human NatC N-terminal acetyltransferase. 著者: Sunbin Deng / Sarah M Gardner / Leah Gottlieb / Buyan Pan / E James Petersson / Ronen Marmorstein /  要旨: N-terminal acetylation occurs on over 80% of human proteins and is catalyzed by a family of N-terminal acetyltransferases (NATs). All NATs contain a small catalytic subunit, while some also contain a ...N-terminal acetylation occurs on over 80% of human proteins and is catalyzed by a family of N-terminal acetyltransferases (NATs). All NATs contain a small catalytic subunit, while some also contain a large auxiliary subunit that facilitates catalysis and ribosome targeting for co-translational acetylation. NatC is one of the major NATs containing an NAA30 catalytic subunit, but uniquely contains two auxiliary subunits, large NAA35 and small NAA38. Here, we report the cryo-EM structures of human NatC (hNatC) complexes with and without NAA38, together with biochemical studies, to reveal that NAA38 increases the thermostability and broadens the substrate-specificity profile of NatC by ordering an N-terminal segment of NAA35 and reorienting an NAA30 N-terminal peptide binding loop for optimal catalysis, respectively. We also note important differences in engagement with a stabilizing inositol hexaphosphate molecule between human and yeast NatC. These studies provide new insights for the function and evolution of the NatC complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24070.map.gz emd_24070.map.gz | 28.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24070-v30.xml emd-24070-v30.xml emd-24070.xml emd-24070.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24070.png emd_24070.png | 140.2 KB | ||
| Filedesc metadata |  emd-24070.cif.gz emd-24070.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24070 http://ftp.pdbj.org/pub/emdb/structures/EMD-24070 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24070 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24070 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24070_validation.pdf.gz emd_24070_validation.pdf.gz | 521.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24070_full_validation.pdf.gz emd_24070_full_validation.pdf.gz | 521.3 KB | 表示 | |
| XML形式データ |  emd_24070_validation.xml.gz emd_24070_validation.xml.gz | 5.9 KB | 表示 | |
| CIF形式データ |  emd_24070_validation.cif.gz emd_24070_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24070 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24070 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24070 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24070 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7mx2MC  7rb3C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24070.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24070.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : heterotimeric human NatC complex with a bisubstrate inhibitor
| 全体 | 名称: heterotimeric human NatC complex with a bisubstrate inhibitor |
|---|---|
| 要素 |
|
-超分子 #1: heterotimeric human NatC complex with a bisubstrate inhibitor
| 超分子 | 名称: heterotimeric human NatC complex with a bisubstrate inhibitor タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 114 KDa |
-超分子 #2: N-alpha-acetyltransferase NatC complex
| 超分子 | 名称: N-alpha-acetyltransferase NatC complex / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: bisubstrate inhibitor
| 超分子 | 名称: bisubstrate inhibitor / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: N-alpha-acetyltransferase 35, NatC auxiliary subunit
| 分子 | 名称: N-alpha-acetyltransferase 35, NatC auxiliary subunit タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 83.754031 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVMKASVDDD DSGWELSMPE KMEKSNTNWV DITQDFEEAC RELKLGELLH DKLFGLFEAM SAIEMMDPKM DAGMIGNQVN RKVLNFEQA IKDGTIKIKD LTLPELIGIM DTCFCCLITW LEGHSLAQTV FTCLYIHNPD FIEDPAMKAF ALGILKICDI A REKVNKAA ...文字列: MVMKASVDDD DSGWELSMPE KMEKSNTNWV DITQDFEEAC RELKLGELLH DKLFGLFEAM SAIEMMDPKM DAGMIGNQVN RKVLNFEQA IKDGTIKIKD LTLPELIGIM DTCFCCLITW LEGHSLAQTV FTCLYIHNPD FIEDPAMKAF ALGILKICDI A REKVNKAA VFEEEDFQSM TYGFKMANSV TDLRVTGMLK DVEDDMQRRV KSTRSRQGEE RDPEVELEHQ QCLAVFSRVK FT RVLLTVL IAFTKKETSA VAEAQKLMVQ AADLLSAIHN SLHHGIQAQN DTTKGDHPIM MGFEPLVNQR LLPPTFPRYA KII KREEMV NYFARLIDRI KTVCEVVNLT NLHCILDFFC EFSEQSPCVL SRSLLQTTFL VDNKKVFGTH LMQDMVKDAL RSFV SPPVL SPKCYLYNNH QAKDCIDSFV THCVRPFCSL IQIHGHNRAR QRDKLGHILE EFATLQDEAE KVDAALHTML LKQEP QRQH LACLGTWVLY HNLRIMIQYL LSGFELELYS MHEYYYIYWY LSEFLYAWLM STLSRADGSQ MAEERIMEEQ QKGRSS KKT KKKKKVRPLS REITMSQAYQ NMCAGMFKTM VAFDMDGKVR KPKFELDSEQ VRYEHRFAPF NSVMTPPPVH YLQFKEM SD LNKYSPPPQS PELYVAASKH FQQAKMILEN IPNPDHEVNR ILKVAKPNFV VMKLLAGGHK KESKVPPEFD FSAHKYFP V VKLV UniProtKB: N-alpha-acetyltransferase 35, NatC auxiliary subunit |
-分子 #2: N-alpha-acetyltransferase 38, NatC auxiliary subunit
| 分子 | 名称: N-alpha-acetyltransferase 38, NatC auxiliary subunit タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.530276 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGAGPTMLL REENGCCSRR QSSSSAGDSD GEREDSAAER ARQQLEALLN KTMRIRMTDG RTLVGCFLCT DRDCNVILGS AQEFLKPSD SFSAGEPRVL GLAMVPGHHI VSIEVQRESL TGPPYL UniProtKB: N-alpha-acetyltransferase 38, NatC auxiliary subunit |
-分子 #3: N-alpha-acetyltransferase 30
| 分子 | 名称: N-alpha-acetyltransferase 30 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: N-terminal methionine Nalpha-acetyltransferase NatC |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.083293 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RTIRYVRYES ELQMPDIMRL ITKDLSEPYS IYTYRYFIHN WPQLCFLAMV GEECVGAIVC KLDMHKKMFR RGYIAMLAVD SKYRRNGIG TNLVKKAIYA MVEGDCDEVV LETEITNKSA LKLYENLGFV RDKRLFRYYL NGVDALRLKL WLR UniProtKB: N-alpha-acetyltransferase 30 |
-分子 #4: peptide portion of bisubstrate inhibitor
| 分子 | 名称: peptide portion of bisubstrate inhibitor / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 416.535 Da |
| 配列 | 文字列: MLGP |
-分子 #5: CARBOXYMETHYL COENZYME *A
| 分子 | 名称: CARBOXYMETHYL COENZYME *A / タイプ: ligand / ID: 5 / コピー数: 1 / 式: CMC |
|---|---|
| 分子量 | 理論値: 825.57 Da |
| Chemical component information | 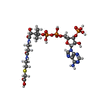 ChemComp-CMC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 平均電子線量: 1.6 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 平均電子線量: 1.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7mx2: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)