+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2340 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The electron microscopy reconstruction of the baseplate of lactococcal phage Tuc2009. | |||||||||
 マップデータ マップデータ | Reconstruction of the baseplate of lactococcal phage Tuc2009 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | lactococcal phage / tuc2009 / baseplate / electron microscopy / single-particle | |||||||||
| 生物種 |  Lactococcus phage Tuc2009 (ファージ) Lactococcus phage Tuc2009 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 36.0 Å | |||||||||
 データ登録者 データ登録者 | Collins B / Bebeacua C / Mahony J / Douillard F / Veesler D / Blangy B / Cambillau C / van Sinderen D | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2013 ジャーナル: J Virol / 年: 2013タイトル: Structure and functional analysis of the host recognition device of lactococcal phage tuc2009. 著者: Barry Collins / Cecilia Bebeacua / Jennifer Mahony / Stéphanie Blangy / François P Douillard / David Veesler / Christian Cambillau / Douwe van Sinderen /  要旨: Many phages employ a large heteropolymeric organelle located at the tip of the tail, termed the baseplate, for host recognition. Contrast electron microscopy (EM) of the lactococcal phage Tuc2009 ...Many phages employ a large heteropolymeric organelle located at the tip of the tail, termed the baseplate, for host recognition. Contrast electron microscopy (EM) of the lactococcal phage Tuc2009 baseplate and its host-binding subunits, the so-called tripods, allowed us to obtain a low-resolution structural image of this organelle. Structural comparisons between the baseplate of the related phage TP901-1 and that of Tuc2009 demonstrated that they are highly similar, except for the presence of an additional protein in the Tuc2009 baseplate (BppATuc2009), which is attached to the top of the Tuc2009 tripod structure. Recombinantly produced Tuc2009 or TP901-1 tripods were shown to bind specifically to their particular host cell surfaces and are capable of almost fully and specifically eliminating Tuc2009 or TP901-1 phage adsorption, respectively. In the case of Tuc2009, such adsorption-blocking ability was reduced in tripods that lacked BppATuc2009, indicating that this protein increases the binding specificity and/or affinity of the Tuc2009 tripod to its host receptor. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2340.map.gz emd_2340.map.gz | 8.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2340-v30.xml emd-2340-v30.xml emd-2340.xml emd-2340.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2340.png emd_2340.png | 103.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2340 http://ftp.pdbj.org/pub/emdb/structures/EMD-2340 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2340 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2340 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2340_validation.pdf.gz emd_2340_validation.pdf.gz | 198.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2340_full_validation.pdf.gz emd_2340_full_validation.pdf.gz | 197.3 KB | 表示 | |
| XML形式データ |  emd_2340_validation.xml.gz emd_2340_validation.xml.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2340 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2340 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2340 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2340 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2340.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2340.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the baseplate of lactococcal phage Tuc2009 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.95 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
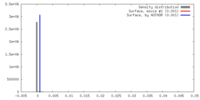
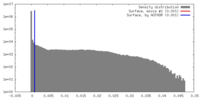
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Baseplate of lactoccocal phage Tuc2009
| 全体 | 名称: Baseplate of lactoccocal phage Tuc2009 |
|---|---|
| 要素 |
|
-超分子 #1000: Baseplate of lactoccocal phage Tuc2009
| 超分子 | 名称: Baseplate of lactoccocal phage Tuc2009 / タイプ: sample / ID: 1000 詳細: The baseplate was selected from images collected from a sample of the entire phages. 集合状態: 6 / Number unique components: 1 |
|---|
-超分子 #1: Lactococcus phage Tuc2009
| 超分子 | 名称: Lactococcus phage Tuc2009 / タイプ: virus / ID: 1 / Name.synonym: Tuc2009 / NCBI-ID: 35241 / 生物種: Lactococcus phage Tuc2009 / ウイルスタイプ: OTHER / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: Tuc2009 |
|---|---|
| 宿主 | 生物種:  Lactococcus lactis (乳酸菌) / 株: UC509.9 / 別称: BACTERIA(EUBACTERIA) Lactococcus lactis (乳酸菌) / 株: UC509.9 / 別称: BACTERIA(EUBACTERIA) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris-HCL, 10 mM MgSO4, 100 mM NaCl |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Grids with adsorbed protein floated on 2% w/v uranyl acetate for 20 seconds |
| グリッド | 詳細: 300 mesh copper grid with thin carbon layer, glow discharged for 30 seconds |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100,000 times magnification |
| 日付 | 2012年6月1日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / 実像数: 2000 / 平均電子線量: 10 e/Å2 / 詳細: Images were collected using a 2kx2k CCD camera |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 48500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 48500 |
| 試料ステージ | 試料ホルダー: Room temperature holder / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 36.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, Spider, Xmipp / 使用した粒子像数: 2000 |
|---|
-原子モデル構築 1
| 初期モデル | PDB ID:  4div |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The baseplate was manually fitted and then automatically refined |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)