[日本語] English
 万見
万見- EMDB-23293: Trimeric human Arginase 1 in complex with mAb1 - 2 hArg:3 mAb1 complex -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23293 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Trimeric human Arginase 1 in complex with mAb1 - 2 hArg:3 mAb1 complex | |||||||||
 マップデータ マップデータ | Full map of 2 hARG trimers bound to 3Mab1, with better definition for one of the trimer/Fab half. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Arginase / Metalloenzyme / IMMUNE SYSTEM / HYDROLASE-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of neutrophil mediated killing of fungus / negative regulation of T-helper 2 cell cytokine production / Urea cycle / arginase / arginase activity / : / urea cycle / response to nematode / negative regulation of type II interferon-mediated signaling pathway / defense response to protozoan ...positive regulation of neutrophil mediated killing of fungus / negative regulation of T-helper 2 cell cytokine production / Urea cycle / arginase / arginase activity / : / urea cycle / response to nematode / negative regulation of type II interferon-mediated signaling pathway / defense response to protozoan / negative regulation of activated T cell proliferation / L-arginine catabolic process / negative regulation of T cell proliferation / specific granule lumen / azurophil granule lumen / manganese ion binding / adaptive immune response / innate immune response / Neutrophil degranulation / extracellular space / extracellular region / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
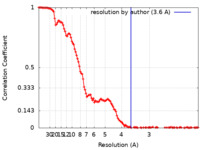
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Gomez-Llorente Y / Scapin G | |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2021 ジャーナル: Commun Biol / 年: 2021タイトル: Cryo-EM structures of inhibitory antibodies complexed with arginase 1 provide insight into mechanism of action. 著者: Rachel L Palte / Veronica Juan / Yacob Gomez-Llorente / Marc Andre Bailly / Kalyan Chakravarthy / Xun Chen / Daniel Cipriano / Ghassan N Fayad / Laurence Fayadat-Dilman / Symon Gathiaka / ...著者: Rachel L Palte / Veronica Juan / Yacob Gomez-Llorente / Marc Andre Bailly / Kalyan Chakravarthy / Xun Chen / Daniel Cipriano / Ghassan N Fayad / Laurence Fayadat-Dilman / Symon Gathiaka / Heiko Greb / Brian Hall / Mas Handa / Mark Hsieh / Esther Kofman / Heping Lin / J Richard Miller / Nhung Nguyen / Jennifer O'Neil / Hussam Shaheen / Eric Sterner / Corey Strickland / Angie Sun / Shane Taremi / Giovanna Scapin /  要旨: Human Arginase 1 (hArg1) is a metalloenzyme that catalyzes the hydrolysis of L-arginine to L-ornithine and urea, and modulates T-cell-mediated immune response. Arginase-targeted therapies have been ...Human Arginase 1 (hArg1) is a metalloenzyme that catalyzes the hydrolysis of L-arginine to L-ornithine and urea, and modulates T-cell-mediated immune response. Arginase-targeted therapies have been pursued across several disease areas including immunology, oncology, nervous system dysfunction, and cardiovascular dysfunction and diseases. Currently, all published hArg1 inhibitors are small molecules usually less than 350 Da in size. Here we report the cryo-electron microscopy structures of potent and inhibitory anti-hArg antibodies bound to hArg1 which form distinct macromolecular complexes that are greater than 650 kDa. With local resolutions of 3.5 Å or better we unambiguously mapped epitopes and paratopes for all five antibodies and determined that the antibodies act through orthosteric and allosteric mechanisms. These hArg1:antibody complexes present an alternative mechanism to inhibit hArg1 activity and highlight the ability to utilize antibodies as probes in the discovery and development of peptide and small molecule inhibitors for enzymes in general. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23293.map.gz emd_23293.map.gz | 139.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23293-v30.xml emd-23293-v30.xml emd-23293.xml emd-23293.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23293_fsc.xml emd_23293_fsc.xml | 15.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23293.png emd_23293.png | 34.6 KB | ||
| マスクデータ |  emd_23293_msk_1.map emd_23293_msk_1.map | 147.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23293.cif.gz emd-23293.cif.gz | 6.6 KB | ||
| その他 |  emd_23293_half_map_1.map.gz emd_23293_half_map_1.map.gz emd_23293_half_map_2.map.gz emd_23293_half_map_2.map.gz | 136.8 MB 136.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23293 http://ftp.pdbj.org/pub/emdb/structures/EMD-23293 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23293 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23293 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23293_validation.pdf.gz emd_23293_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23293_full_validation.pdf.gz emd_23293_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_23293_validation.xml.gz emd_23293_validation.xml.gz | 19.3 KB | 表示 | |
| CIF形式データ |  emd_23293_validation.cif.gz emd_23293_validation.cif.gz | 25.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23293 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23293 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23293 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23293 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23293.map.gz / 形式: CCP4 / 大きさ: 147.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23293.map.gz / 形式: CCP4 / 大きさ: 147.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full map of 2 hARG trimers bound to 3Mab1, with better definition for one of the trimer/Fab half. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
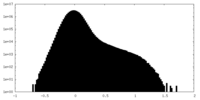
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23293_msk_1.map emd_23293_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
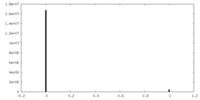
| 密度ヒストグラム |
-ハーフマップ: Half map B.
| ファイル | emd_23293_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
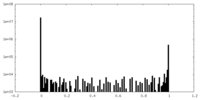
| 密度ヒストグラム |
-ハーフマップ: Half map A.
| ファイル | emd_23293_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
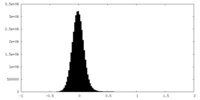
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Trimeric human arginase in complex with mAb1
| 全体 | 名称: Trimeric human arginase in complex with mAb1 |
|---|---|
| 要素 |
|
-超分子 #1: Trimeric human arginase in complex with mAb1
| 超分子 | 名称: Trimeric human arginase in complex with mAb1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: 2 trimers of human arginase bound to the Fab regions of 3 mAb1 molecules. |
|---|---|
| 分子量 | 理論値: 427 KDa |
-超分子 #2: Trimeric human arginase
| 超分子 | 名称: Trimeric human arginase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: mAb1
| 超分子 | 名称: mAb1 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Arginase-1
| 分子 | 名称: Arginase-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: arginase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 34.779879 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAKSRTIGI IGAPFSKGQP RGGVEEGPTV LRKAGLLEKL KEQECDVKDY GDLPFADIPN DSPFQIVKNP RSVGKASEQL AGKVAEVKK NGRISLVLGG DHSLAIGSIS GHARVHPDLG VIWVDAHTDI NTPLTTTSGN LHGQPVSFLL KELKGKIPDV P GFSWVTPC ...文字列: MSAKSRTIGI IGAPFSKGQP RGGVEEGPTV LRKAGLLEKL KEQECDVKDY GDLPFADIPN DSPFQIVKNP RSVGKASEQL AGKVAEVKK NGRISLVLGG DHSLAIGSIS GHARVHPDLG VIWVDAHTDI NTPLTTTSGN LHGQPVSFLL KELKGKIPDV P GFSWVTPC ISAKDIVYIG LRDVDPGEHY ILKTLGIKYF SMTEVDRLGI GKVMEETLSY LLGRKKRPIH LSFDVDGLDP SF TPATGTP VVGGLTYREG LYITEEIYKT GLLSGLDIME VNPSLGKTPE EVTRTVNTAV AITLACFGLA REGNHKPIDY LNP PK UniProtKB: Arginase-1 |
-分子 #2: mAb1 heavy chain
| 分子 | 名称: mAb1 heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50.269605 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVQSGAE VKKPGASVKV SCKASGYTFT NYGISWVRQA PGQGLEWMGW ISAYNGNTNY AQKLQGRVTM TTDTSTSTAY MELRSLRSD DTAVYYCARE GAYGYRSPYH NWFDPWGQGT LVTVSSAKTT APSVYPLAPV CGDTTGSSVT LGCLVKGYFP E PVTLTWNS ...文字列: QVQLVQSGAE VKKPGASVKV SCKASGYTFT NYGISWVRQA PGQGLEWMGW ISAYNGNTNY AQKLQGRVTM TTDTSTSTAY MELRSLRSD DTAVYYCARE GAYGYRSPYH NWFDPWGQGT LVTVSSAKTT APSVYPLAPV CGDTTGSSVT LGCLVKGYFP E PVTLTWNS GSLSSGVHTF PAVLQSDLYT LSSSVTVTSS TWPSQSITCN VAHPASSTKV DKKIEPRGPT IKPCPPCKCP AP NLLGGPS VFIFPPKIKD VLMISLSPIV TCVVVDVSED DPDVQISWFV NNVEVHTAQT QTHREDYNST LRVVSALPIQ HQD WMSGKE FKCKVNNKDL PAPIERTISK PKGSVRAPQV YVLPPPEEEM TKKQVTLTCM VTDFMPEDIY VEWTNNGKTE LNYK NTEPV LDSDGSYFMY SKLRVEKKNW VERNSYSCSV VHEGLHNHHT TKSFSRTPGK |
-分子 #3: mAb1 light chain
| 分子 | 名称: mAb1 light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.488965 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EIVMTQSPAT LSLSPGERAT LSCRASQSVS SYLAWYQQKP GQAPRLLIYD ASNRATGIPA RFSGSGSGTD FTLTISSLEP EDFAVYYCQ QHSLLPRTFG GGTKVEIKRA DAAPTVSIFP PSSEQLTSGG ASVVCFLNNF YPKDINVKWK IDGSERQNGV L NSWTDQDS ...文字列: EIVMTQSPAT LSLSPGERAT LSCRASQSVS SYLAWYQQKP GQAPRLLIYD ASNRATGIPA RFSGSGSGTD FTLTISSLEP EDFAVYYCQ QHSLLPRTFG GGTKVEIKRA DAAPTVSIFP PSSEQLTSGG ASVVCFLNNF YPKDINVKWK IDGSERQNGV L NSWTDQDS KDSTYSMSST LTLTKDEYER HNSYTCEATH KTSTSPIVKS FNRNEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 1 / 実像数: 1506 / 平均露光時間: 6.0 sec. / 平均電子線量: 45.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7lex: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)