+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23250 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of PCV2 Replicase bound to ssDNA | |||||||||||||||
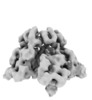
 マップデータ マップデータ | Cryo-EM map of PCV2 Replicase in complex with dsDNA at 4.4 Angstrom resolution | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Rolling circle replication / CRESS-DNA virus / SF3 helicase / Porcine Circovirus / PCV2 / REPLICATION / HYDROLASE-DNA complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nucleotidyltransferase activity / endonuclease activity / DNA replication / RNA helicase activity / nucleotide binding / host cell nucleus / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Porcine circovirus 2 (ウイルス) / Porcine circovirus 2 (ウイルス) /  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Khayat R | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2021 ジャーナル: mBio / 年: 2021タイトル: Mechanism of DNA Interaction and Translocation by the Replicase of a Circular Rep-Encoding Single-Stranded DNA Virus. 著者: Elvira Tarasova / Sonali Dhindwal / Matthew Popp / Sakeenah Hussain / Reza Khayat /  要旨: Circular Rep-encoding single-stranded DNA (CRESS-DNA) viruses infect members from all three domains of life (, , and ). The replicase (Rep) from these viruses is responsible for initiating rolling ...Circular Rep-encoding single-stranded DNA (CRESS-DNA) viruses infect members from all three domains of life (, , and ). The replicase (Rep) from these viruses is responsible for initiating rolling circle replication (RCR) of their genomes. Rep is a multifunctional enzyme responsible for nicking and ligating ssDNA and unwinding double-stranded DNA (dsDNA). We report the structure of porcine circovirus 2 (PCV2) Rep bound to ADP and single-stranded DNA (ssDNA), and Rep bound to ADP and double-stranded DNA (dsDNA). The structures demonstrate Rep to be a member of the superfamily 3 (SF3) of ATPases Associated with diverse cellular Activities (AAA) superfamily clade 4. At the Rep N terminus is an endonuclease domain () that is responsible for ssDNA nicking and ligation, in the center of Rep is an oligomerization domain () responsible for hexamerization, and at the C terminus is an ATPase domain () responsible for ssDNA/dsDNA interaction and translocation. The Rep binds to DNA such that the faces the replication fork. The six spiral around the DNA to interact with the backbone phosphates from four consecutive nucleotides. Three of the six are able to sense the backbone phosphates from the second strand of dsDNA. Heterogeneous classification of the data demonstrates the and to be mobile. Furthermore, we demonstrate that Rep exhibits basal nucleoside triphosphatase (NTPase) activity. CRESS-DNA viruses encompass a significant portion of the biosphere's virome. However, little is known about the structure of Rep responsible for initiating the RCR of CRESS-DNA viruses. We use cryo-electron microscopy (cryo-EM) to determine the structure of PCV2 Rep in complex with ADP and ss/dsDNA. Our structures demonstrate CRESS-DNA Reps to be SF3 members (clade 4) of the AAA+ superfamily. The structures further provide the mechanism by which CRESS-DNA virus Reps recognize DNA and translocate DNA for genome replication. Our structures also demonstrate the and of PCV2 Rep to be highly mobile. We propose the mobile nature of these domains to be necessary for proper functioning of Reps. We further demonstrate that Reps exhibit basal NTPase activity. Our studies also provide initial insight into the mechanism of RCR. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23250.map.gz emd_23250.map.gz | 32.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23250-v30.xml emd-23250-v30.xml emd-23250.xml emd-23250.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23250.png emd_23250.png | 45.6 KB | ||
| Filedesc metadata |  emd-23250.cif.gz emd-23250.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23250 http://ftp.pdbj.org/pub/emdb/structures/EMD-23250 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23250 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23250 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23250_validation.pdf.gz emd_23250_validation.pdf.gz | 568.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23250_full_validation.pdf.gz emd_23250_full_validation.pdf.gz | 568.4 KB | 表示 | |
| XML形式データ |  emd_23250_validation.xml.gz emd_23250_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_23250_validation.cif.gz emd_23250_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23250 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23250 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23250 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23250 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23250.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23250.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of PCV2 Replicase in complex with dsDNA at 4.4 Angstrom resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PCV2 Replicase in complex with ssDNA
| 全体 | 名称: PCV2 Replicase in complex with ssDNA |
|---|---|
| 要素 |
|
-超分子 #1: PCV2 Replicase in complex with ssDNA
| 超分子 | 名称: PCV2 Replicase in complex with ssDNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Porcine circovirus 2 (ウイルス) Porcine circovirus 2 (ウイルス) |
| 分子量 | 理論値: 216 KDa |
-分子 #1: ATP-dependent helicase Rep
| 分子 | 名称: ATP-dependent helicase Rep / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:   Porcine circovirus 2 (ウイルス) Porcine circovirus 2 (ウイルス) |
| 分子量 | 理論値: 35.8765 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPSKKNGRSG PQPHKRWVFT LNNPSEDERK KIRELPISLF DYFIVGEEGN EEGRTPHLQG FANFVKKQTF NKVKWYFGAR CHIEKAKGT DQQNKEYCSK EGNLLMECGA PRSQGQRSDL STAVSTLLES GSLVTVAEQH PVTFVRNFRG LAELLKVSGK M QKRDWKTN ...文字列: MPSKKNGRSG PQPHKRWVFT LNNPSEDERK KIRELPISLF DYFIVGEEGN EEGRTPHLQG FANFVKKQTF NKVKWYFGAR CHIEKAKGT DQQNKEYCSK EGNLLMECGA PRSQGQRSDL STAVSTLLES GSLVTVAEQH PVTFVRNFRG LAELLKVSGK M QKRDWKTN VHVIVGPPGC GKSKWAANFA DPETTYWKPP RNKWWDGYHG EEVVVIDDFY GWLPWDDLLR LCDRYPLTVE TK GGTVPFL ARSILITSNQ TPLEWYSSTA VPAVEALYRR ITSLVFWKNA TEQSTEEGGQ FVTLSPPCPE FPYEINY UniProtKB: Replication-associated protein |
-分子 #2: DNA (5'-D(P*TP*TP*TP*TP*TP*CP*GP*AP*TP*CP*GP*AP*TP*C)-3')
| 分子 | 名称: DNA (5'-D(P*TP*TP*TP*TP*TP*CP*GP*AP*TP*CP*GP*AP*TP*C)-3') タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 4.236762 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DC)(DG)(DA)(DT)(DC) (DG)(DA)(DT)(DC) |
-分子 #3: DNA (5'-D(P*GP*AP*TP*CP*GP*AP*TP*CP*GP*A)-3')
| 分子 | 名称: DNA (5'-D(P*GP*AP*TP*CP*GP*AP*TP*CP*GP*A)-3') / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Porcine circovirus 2 (ウイルス) Porcine circovirus 2 (ウイルス) |
| 分子量 | 理論値: 3.06903 KDa |
| 配列 | 文字列: (DG)(DA)(DT)(DC)(DG)(DA)(DT)(DC)(DG)(DA) |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 4 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.2 詳細: 20 mM HEPES, pH 8.2, 500 mM NaCl, 0.2 mM TCEP, 10 mM MgCl2 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 4 uL samples were applied to the grids for 3 seconds and blotted using a blot force of 2, a blot time of 4 seconds, and a drain time of 0 seconds.. |
| 詳細 | Sample was monodisperse according to size exclusion chromatography |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 6.0 sec. / 平均電子線量: 71.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 65.3 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7las: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















