+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2310 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of active, full-length human telomerase dimer, determined by single-particle electron microscopy in negative stain | |||||||||
 マップデータ マップデータ | 3D map of human telomerase dimer bound to G-overhang oligonucleotide (TTAGGG)2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Telomerase reverse transcriptase / human / telomere length extension | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of hair cycle / template-free RNA nucleotidyltransferase / positive regulation of transdifferentiation / TERT-RMRP complex / DNA strand elongation / RNA-directed RNA polymerase complex / siRNA transcription / positive regulation of protein localization to nucleolus / telomerase catalytic core complex / RNA-templated DNA biosynthetic process ...positive regulation of hair cycle / template-free RNA nucleotidyltransferase / positive regulation of transdifferentiation / TERT-RMRP complex / DNA strand elongation / RNA-directed RNA polymerase complex / siRNA transcription / positive regulation of protein localization to nucleolus / telomerase catalytic core complex / RNA-templated DNA biosynthetic process / establishment of protein localization to telomere / telomerase activity / Regulation of MITF-M-dependent genes involved in DNA replication, damage repair and senescence / nuclear telomere cap complex / siRNA processing / telomere maintenance via recombination / positive regulation of vascular associated smooth muscle cell migration / telomerase holoenzyme complex / telomerase RNA binding / DNA biosynthetic process / RNA-templated transcription / telomeric DNA binding / positive regulation of stem cell proliferation / mitochondrial nucleoid / negative regulation of cellular senescence / replicative senescence / Telomere Extension By Telomerase / positive regulation of Wnt signaling pathway / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / positive regulation of G1/S transition of mitotic cell cycle / response to cadmium ion / positive regulation of protein binding / telomere maintenance via telomerase / negative regulation of endothelial cell apoptotic process / positive regulation of vascular associated smooth muscle cell proliferation / telomere maintenance / positive regulation of D-glucose import / mitochondrion organization / Formation of the beta-catenin:TCF transactivating complex / PML body / regulation of protein stability / positive regulation of miRNA transcription / RNA-directed DNA polymerase / transcription coactivator binding / positive regulation of angiogenesis / RNA-directed DNA polymerase activity / protein import into nucleus / protein-folding chaperone binding / heart development / cellular response to hypoxia / negative regulation of neuron apoptotic process / chromosome, telomeric region / tRNA binding / nuclear speck / RNA-directed RNA polymerase activity / nucleolus / protein homodimerization activity / DNA binding / RNA binding / nucleoplasm / metal ion binding / identical protein binding / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 30.0 Å | |||||||||
 データ登録者 データ登録者 | Sauerwald A / Sandin S / Cristofari G / Scheres SHW / Lingner J / Rhodes D | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2013 ジャーナル: Nat Struct Mol Biol / 年: 2013タイトル: Structure of active dimeric human telomerase. 著者: Anselm Sauerwald / Sara Sandin / Gaël Cristofari / Sjors H W Scheres / Joachim Lingner / Daniela Rhodes /  要旨: Telomerase contains a large RNA subunit, TER, and a protein catalytic subunit, TERT. Whether telomerase functions as a monomer or dimer has been a matter of debate. Here we report biochemical and ...Telomerase contains a large RNA subunit, TER, and a protein catalytic subunit, TERT. Whether telomerase functions as a monomer or dimer has been a matter of debate. Here we report biochemical and labeling data that show that in vivo-assembled human telomerase contains two TERT subunits and binds two telomeric DNA substrates. Notably, catalytic activity requires both TERT active sites to be functional, which demonstrates that human telomerase functions as a dimer. We also present the three-dimensional structure of the active full-length human telomerase dimer, determined by single-particle EM in negative stain. Telomerase has a bilobal architecture with the two monomers linked by a flexible interface. The monomer reconstruction at 23-Å resolution and fitting of the atomic structure of the TERT subunit from beetle Tribolium castaneum into the EM density reveals the spatial relationship between RNA and protein subunits, providing insights into telomerase architecture. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2310.map.gz emd_2310.map.gz | 133.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2310-v30.xml emd-2310-v30.xml emd-2310.xml emd-2310.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2310.png EMD-2310.png | 39.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2310 http://ftp.pdbj.org/pub/emdb/structures/EMD-2310 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2310 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2310 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2310_validation.pdf.gz emd_2310_validation.pdf.gz | 188.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2310_full_validation.pdf.gz emd_2310_full_validation.pdf.gz | 187.9 KB | 表示 | |
| XML形式データ |  emd_2310_validation.xml.gz emd_2310_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2310 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2310 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2310 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2310 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2310.map.gz / 形式: CCP4 / 大きさ: 144.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2310.map.gz / 形式: CCP4 / 大きさ: 144.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D map of human telomerase dimer bound to G-overhang oligonucleotide (TTAGGG)2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Three-dimensional structure of active, full-length human telomera...
| 全体 | 名称: Three-dimensional structure of active, full-length human telomerase dimer, determined by single-particle electron microscopy in negative stain |
|---|---|
| 要素 |
|
-超分子 #1000: Three-dimensional structure of active, full-length human telomera...
| 超分子 | 名称: Three-dimensional structure of active, full-length human telomerase dimer, determined by single-particle electron microscopy in negative stain タイプ: sample / ID: 1000 詳細: The composition analysed by mass spectroscopy.Purified telomerase contains the hTERT subunits and two accessory proteins, Nop10 and dyskerin. Telomerase complexes have a molecular weight ...詳細: The composition analysed by mass spectroscopy.Purified telomerase contains the hTERT subunits and two accessory proteins, Nop10 and dyskerin. Telomerase complexes have a molecular weight consistent with that of a dimer consisting of two hTERT (127 kDa) and two hTER (153 kDa) subunits, as well as the two accessory proteins Nop10 (7.7 kDa) and dyskerin (58 kDA). 集合状態: Dimer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 670 KDa / 理論値: 626 KDa / 手法: Sedimentation |
-分子 #1: Telomerase reverse transcriptase
| 分子 | 名称: Telomerase reverse transcriptase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: TERT / コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞: Human Embryonic Kidney cells Homo sapiens (ヒト) / 別称: Human / 細胞: Human Embryonic Kidney cells |
| 分子量 | 実験値: 127 KDa / 理論値: 670 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK239T / 組換プラスミド: pCDNA6 Homo sapiens (ヒト) / 組換細胞: HEK239T / 組換プラスミド: pCDNA6 |
| 配列 | UniProtKB: Telomerase reverse transcriptase / GO: telomere maintenance / InterPro: Telomerase reverse transcriptase |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 20 mM Tris, 150 mM KCl, 1 mM MgCl2 |
| 染色 | タイプ: NEGATIVE 詳細: Continuous carbon-coated grids were freshly prepared and glow-discharged before use. 13 microl of telomerase sample (8-10 nM) were deposited on the grid for 15-30 minutes, blotted with filter ...詳細: Continuous carbon-coated grids were freshly prepared and glow-discharged before use. 13 microl of telomerase sample (8-10 nM) were deposited on the grid for 15-30 minutes, blotted with filter paper and negatively stained with 2 drops of 1-2% (w/v) uranyl acetate solution. |
| グリッド | 詳細: 200 mesh carbon coated with thin carbon, glow discharged |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| アライメント法 | Legacy - 非点収差: Corrected at 100,000 times magnification |
| 詳細 | The films were developed in Kodak developer at full strength for 12 min. |
| 日付 | 2011年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 482 / 平均電子線量: 10 e/Å2 / 詳細: The micrographs were compressed x4 / Od range: 1.4 |
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 倍率(補正後): 42550 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.0015 µm / 最小 デフォーカス(公称値): 0.001 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 詳細 | Electron tomography was used to calculate a low-resolution reference model. Distinct conformations were further classified by three-dimensional maximum-likelihood analysis in Fourier space (MLF3D).Density map calculated from a sub-population of 3,659 particles. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 30.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, XMIPP 詳細: 3D structure was refined using the low-resolution subtomogram average as a reference model for 3D single particle refinement with EMAN2. Distinct conformations were further classified by ...詳細: 3D structure was refined using the low-resolution subtomogram average as a reference model for 3D single particle refinement with EMAN2. Distinct conformations were further classified by three-dimensional maximum-likelihood analysis in Fourier space (MLF3D). Density map calculated from a sub-population of 3,659 particles. The absolute hand and the overall correctness of the dimer structure were assessed using a modified version of tilt-pair validation method. 使用した粒子像数: 20127 |
| 最終 2次元分類 | クラス数: 100 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera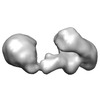










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















