+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23081 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
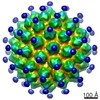
| タイトル | High resolution cryo EM analysis of HPV16 identifies minor structural protein L2 and describes capsid flexibility | |||||||||
 マップデータ マップデータ | HPV16 quasivirus capsid. Recombined map from pentavalent and hexavalent subvolume refinements. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HPV16 / quasivirus / L1 / capsomer / subparticle / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=7 icosahedral viral capsid / endocytosis involved in viral entry into host cell / host cell nucleus / virion attachment to host cell / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human papillomavirus type 16 (パピローマウイルス) Human papillomavirus type 16 (パピローマウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Hartmann SR / Goetschius DJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2021 ジャーナル: Sci Rep / 年: 2021タイトル: High resolution cryo EM analysis of HPV16 identifies minor structural protein L2 and describes capsid flexibility. 著者: Daniel J Goetschius / Samantha R Hartmann / Suriyasri Subramanian / Carol M Bator / Neil D Christensen / Susan L Hafenstein /  要旨: Human papillomavirus (HPV) is a significant health burden and leading cause of virus-induced cancers. HPV is epitheliotropic and its replication is tightly associated with terminal keratinocyte ...Human papillomavirus (HPV) is a significant health burden and leading cause of virus-induced cancers. HPV is epitheliotropic and its replication is tightly associated with terminal keratinocyte differentiation making production and purification of high titer virus preparations for research problematic, therefore alternative HPV production methods have been developed for virological and structural studies. In this study we use HPV16 quasivirus, composed of HPV16 L1/L2 capsid proteins with a packaged cottontail rabbit papillomavirus genome. We have achieved the first high resolution, 3.1 Å, structure of HPV16 by using a local subvolume refinement approach. The high resolution enabled us to build L1 unambiguously and identify L2 protein strands. The L2 density is incorporated adjacent to conserved L1 residues on the interior of the capsid. Further interpretation with our own software for Icosahedral Subvolume Extraction and Correlated Classification revealed flexibility, on the whole-particle level through diameter analysis and local movement with inter-capsomer analysis. Inter-capsomer expansion or contraction, governed by the connecting arms, showed no bias in the magnitude or direction of capsomer movement. We propose that papillomavirus capsids are dynamic and capsomers move as rigid bodies connected by flexible linkers. The resulting virus structure will provide a framework for continuing biochemical, genetic and biophysical research for papillomaviruses. Furthermore, our approach has allowed insight into the resolution barrier that has previously been a limitation in papillomavirus structural studies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23081.map.gz emd_23081.map.gz | 696.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23081-v30.xml emd-23081-v30.xml emd-23081.xml emd-23081.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23081.png emd_23081.png | 225.5 KB | ||
| Filedesc metadata |  emd-23081.cif.gz emd-23081.cif.gz | 5.7 KB | ||
| その他 |  emd_23081_additional_1.map.gz emd_23081_additional_1.map.gz emd_23081_additional_2.map.gz emd_23081_additional_2.map.gz emd_23081_additional_3.map.gz emd_23081_additional_3.map.gz | 96.2 MB 96.1 MB 1.2 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23081 http://ftp.pdbj.org/pub/emdb/structures/EMD-23081 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23081 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23081 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23081_validation.pdf.gz emd_23081_validation.pdf.gz | 793.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23081_full_validation.pdf.gz emd_23081_full_validation.pdf.gz | 793.5 KB | 表示 | |
| XML形式データ |  emd_23081_validation.xml.gz emd_23081_validation.xml.gz | 8.5 KB | 表示 | |
| CIF形式データ |  emd_23081_validation.cif.gz emd_23081_validation.cif.gz | 9.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23081 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23081 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23081 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23081 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23081.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23081.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HPV16 quasivirus capsid. Recombined map from pentavalent and hexavalent subvolume refinements. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
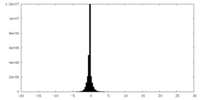
-追加マップ: HPV16 quasivirus pentavalent capsomer subvolume refinement.
| ファイル | emd_23081_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HPV16 quasivirus pentavalent capsomer subvolume refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
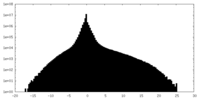
-追加マップ: HPV16 quasivirus hexavalent capsomer subvolume refinement.
| ファイル | emd_23081_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HPV16 quasivirus hexavalent capsomer subvolume refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
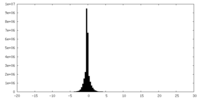
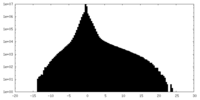
-追加マップ: HPV16 quasivirus icosahedral refinement from which pentavalent and...
| ファイル | emd_23081_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HPV16 quasivirus icosahedral refinement from which pentavalent and hexavalent subvolumes were extracted. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human papillomavirus type 16
| 全体 | 名称:  Human papillomavirus type 16 (パピローマウイルス) Human papillomavirus type 16 (パピローマウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human papillomavirus type 16
| 超分子 | 名称: Human papillomavirus type 16 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 333760 / 生物種: Human papillomavirus type 16 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Major capsid protein L1
| 分子 | 名称: Major capsid protein L1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human papillomavirus type 16 (パピローマウイルス) Human papillomavirus type 16 (パピローマウイルス) |
| 分子量 | 理論値: 56.098617 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSLWLPSEAT VYLPPVPVSK VVSTDEYVAR TNIYYHAGTS RLLAVGHPYF PIKKPNNNKI LVPKVSGLQY RVFRIHLPDP NKFGFPDTS FYNPDTQRLV WACVGVEVGR GQPLGVGISG HPLLNKLDDT ENASAYAANA GVDNRECISM DYKQTQLCLI G CKPPIGEH ...文字列: MSLWLPSEAT VYLPPVPVSK VVSTDEYVAR TNIYYHAGTS RLLAVGHPYF PIKKPNNNKI LVPKVSGLQY RVFRIHLPDP NKFGFPDTS FYNPDTQRLV WACVGVEVGR GQPLGVGISG HPLLNKLDDT ENASAYAANA GVDNRECISM DYKQTQLCLI G CKPPIGEH WGKGSPCTNV AVNPGDCPPL ELINTVIQDG DMVDTGFGAM DFTTLQANKS EVPLDICTSI CKYPDYIKMV SE PYGDSLF FYLRREQMFV RHLFNRAGAV GENVPDDLYI KGSGSTANLA SSNYFPTPSG SMVTSDAQIF NKPYWLQRAQ GHN NGICWG NQLFVTVVDT TRSTNMSLCA AISTSETTYK NTNFKEYLRH GEEYDLQFIF QLCKITLTAD VMTYIHSMNS TILE DWNFG LQPPPGGTLE DTYRFVTSQA IACQKHTPPA PKEDPLKKYT FWEVNLKEKF SADLDQFPLG RKFLLQAGLK AKPKF TLGK RKATPTTSST STTAKRKKR UniProtKB: Major capsid protein L1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 1.0 X / 構成要素 - 名称: PBS |
|---|---|
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 10143 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X