+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23058 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The stress-sensing domain of activated IRE1a forms helical filaments in narrow ER membrane tubes | ||||||||||||
 マップデータ マップデータ | STA of human IRE1 lumenal domain captured in situ using cryo-CLEM and cryo-ET. Helical symmetry applied sym=H2.5:2:45:5, with 653 model points --keep=0.8 and --mask=Auto | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 15.0 Å | ||||||||||||
 データ登録者 データ登録者 | Carter SD / Tran NH / De Maziere A / Ashkenazi A / Klumpermann J / Walter P / Jensen GJ | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: The stress-sensing domain of activated IRE1α forms helical filaments in narrow ER membrane tubes. 著者: Ngoc-Han Tran / Stephen D Carter / Ann De Mazière / Avi Ashkenazi / Judith Klumperman / Peter Walter / Grant J Jensen /   要旨: The signaling network of the unfolded protein response (UPR) adjusts the protein-folding capacity of the endoplasmic reticulum (ER) according to need. The most conserved UPR sensor, IRE1α, spans the ...The signaling network of the unfolded protein response (UPR) adjusts the protein-folding capacity of the endoplasmic reticulum (ER) according to need. The most conserved UPR sensor, IRE1α, spans the ER membrane and activates through oligomerization. IRE1α oligomers accumulate in dynamic foci. We determined the in situ structure of IRE1α foci by cryogenic correlated light and electron microscopy combined with electron cryo-tomography and complementary immuno–electron microscopy in mammalian cell lines. IRE1α foci localized to a network of narrow anastomosing ER tubes (diameter, ~28 nm) with complex branching. The lumen of the tubes contained protein filaments, which were likely composed of arrays of IRE1α lumenal domain dimers that were arranged in two intertwined, left-handed helices. This specialized ER subdomain may play a role in modulating IRE1α signaling. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
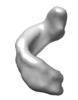
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23058.map.gz emd_23058.map.gz | 662.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23058-v30.xml emd-23058-v30.xml emd-23058.xml emd-23058.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23058.png emd_23058.png | 27.7 KB | ||
| マスクデータ |  emd_23058_msk_1.map emd_23058_msk_1.map | 687 KB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23058 http://ftp.pdbj.org/pub/emdb/structures/EMD-23058 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23058 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23058 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23058_validation.pdf.gz emd_23058_validation.pdf.gz | 301.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23058_full_validation.pdf.gz emd_23058_full_validation.pdf.gz | 301 KB | 表示 | |
| XML形式データ |  emd_23058_validation.xml.gz emd_23058_validation.xml.gz | 4.5 KB | 表示 | |
| CIF形式データ |  emd_23058_validation.cif.gz emd_23058_validation.cif.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23058 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23058 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23058 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23058 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23058.map.gz / 形式: CCP4 / 大きさ: 686.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23058.map.gz / 形式: CCP4 / 大きさ: 686.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | STA of human IRE1 lumenal domain captured in situ using cryo-CLEM and cryo-ET. Helical symmetry applied sym=H2.5:2:45:5, with 653 model points --keep=0.8 and --mask=Auto | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.52 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23058_msk_1.map emd_23058_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
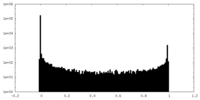
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Double-helical arrangement of human IRE1a luminal domain in 30 nm...
| 全体 | 名称: Double-helical arrangement of human IRE1a luminal domain in 30 nm ER tubes |
|---|---|
| 要素 |
|
-超分子 #1: Double-helical arrangement of human IRE1a luminal domain in 30 nm...
| 超分子 | 名称: Double-helical arrangement of human IRE1a luminal domain in 30 nm ER tubes タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Cryo-CLEM was used to target IRE1a fused to fluorescent protein mNeonGreen (mNG) for cryo-ET imaging. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: ER Homo sapiens (ヒト) / Organelle: ER |
-分子 #1: IRE1 lumenal domain
| 分子 | 名称: IRE1 lumenal domain / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 配列 | 文字列: MPARRLLLLL TLLLPGLGIF GSTSTVTLPE TLLFVSTLDG SLHAVSKRTG SIKWTLKEDP VLQVPTHVEE PAFLPDPNDG SLYTLGSKNN EGLTKLPFTI PELVQASPCR SSDGILYMGK KQDIWYVIDL LTGEKQQTLS SAFADSLCPS TSLLYLGRTE YTITMYDTKT ...文字列: MPARRLLLLL TLLLPGLGIF GSTSTVTLPE TLLFVSTLDG SLHAVSKRTG SIKWTLKEDP VLQVPTHVEE PAFLPDPNDG SLYTLGSKNN EGLTKLPFTI PELVQASPCR SSDGILYMGK KQDIWYVIDL LTGEKQQTLS SAFADSLCPS TSLLYLGRTE YTITMYDTKT RELRWNATYF DYAASLPEDD VDYKMSHFVS NGDGLVVTVD SESGDVLWIQ NYASPVVAFY VWQREGLRKV MHINVAVETL RYLTFMSGEV GRITKWKYPF PKETEAKSKL TPTLYVGKYS TSLYASPSMV HEGVAVVPRG STLPLLEGPQ TDGVTIGDKG ECVITPSTDV KFDPGLKSKN KLNYLRNYWL LIGHHETPLS ASTKMLERFP NNLPKHRENV IPADSEKKSF EEVINLVDQT SENAPTTVSR DVEEKPAHAP ARPEAPVDSM LKD |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 5.0 nm |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 90 % / チャンバー内温度: 310.15 K / 装置: FEI VITROBOT MARK III 詳細: Manual blotting from the gold side of the grid (opposite side to cells).. |
| 詳細 | in situ imaging in a near-native state |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.5 e/Å2 詳細: Images were collected using a bidirectional tilt-scheme. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 倍率(公称値): 34000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 32.6 Å 想定した対称性 - らせんパラメータ - ΔΦ: 45 ° 想定した対称性 - らせんパラメータ - 軸対称性: C2 (2回回転対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 15.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: EMAN2 (ver. 2.3) / 使用したサブトモグラム数: 653 |
|---|---|
| 抽出 | トモグラム数: 3 / 使用した粒子像数: 653 / 手法: Manually every ~85A along the filament / ソフトウェア - 名称: EMAN2 (ver. 2.3) / ソフトウェア - 詳細: e2boxer.py / 詳細: e2boxer.py |
| CTF補正 | ソフトウェア - 名称: EMAN2 (ver. 2.3) / ソフトウェア - 詳細: CTF / 詳細: CTF correction was performed in EMAN2 |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X