+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22876 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the Desulfovibrio vulgaris Type I-C Apo Cascade | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / Cascade / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) / Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) /  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア) | |||||||||
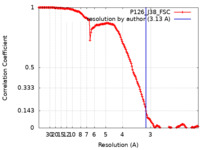
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.13 Å | |||||||||
 データ登録者 データ登録者 | O'Brien R / Wrapp D / Bravo JPK / Schwartz E / Taylor D | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural basis for assembly of non-canonical small subunits into type I-C Cascade. 著者: Roisin E O'Brien / Inês C Santos / Daniel Wrapp / Jack P K Bravo / Evan A Schwartz / Jennifer S Brodbelt / David W Taylor /  要旨: Bacteria and archaea employ CRISPR (clustered, regularly, interspaced, short palindromic repeats)-Cas (CRISPR-associated) systems as a type of adaptive immunity to target and degrade foreign nucleic ...Bacteria and archaea employ CRISPR (clustered, regularly, interspaced, short palindromic repeats)-Cas (CRISPR-associated) systems as a type of adaptive immunity to target and degrade foreign nucleic acids. While a myriad of CRISPR-Cas systems have been identified to date, type I-C is one of the most commonly found subtypes in nature. Interestingly, the type I-C system employs a minimal Cascade effector complex, which encodes only three unique subunits in its operon. Here, we present a 3.1 Å resolution cryo-EM structure of the Desulfovibrio vulgaris type I-C Cascade, revealing the molecular mechanisms that underlie RNA-directed complex assembly. We demonstrate how this minimal Cascade utilizes previously overlooked, non-canonical small subunits to stabilize R-loop formation. Furthermore, we describe putative PAM and Cas3 binding sites. These findings provide the structural basis for harnessing the type I-C Cascade as a genome-engineering tool. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22876.map.gz emd_22876.map.gz | 6.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22876-v30.xml emd-22876-v30.xml emd-22876.xml emd-22876.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22876_fsc.xml emd_22876_fsc.xml | 16.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22876.png emd_22876.png | 163.8 KB | ||
| Filedesc metadata |  emd-22876.cif.gz emd-22876.cif.gz | 6.7 KB | ||
| その他 |  emd_22876_half_map_1.map.gz emd_22876_half_map_1.map.gz emd_22876_half_map_2.map.gz emd_22876_half_map_2.map.gz | 285.3 MB 285.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22876 http://ftp.pdbj.org/pub/emdb/structures/EMD-22876 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22876 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22876 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22876_validation.pdf.gz emd_22876_validation.pdf.gz | 871.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22876_full_validation.pdf.gz emd_22876_full_validation.pdf.gz | 871.4 KB | 表示 | |
| XML形式データ |  emd_22876_validation.xml.gz emd_22876_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_22876_validation.cif.gz emd_22876_validation.cif.gz | 27.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22876 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22876 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22876 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22876 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22876.map.gz / 形式: CCP4 / 大きさ: 6.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22876.map.gz / 形式: CCP4 / 大きさ: 6.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.047 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_22876_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
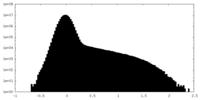
| 投影像・断面図 |
| ||||||||||||
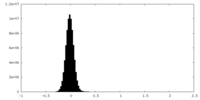
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_22876_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
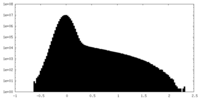
| 投影像・断面図 |
| ||||||||||||
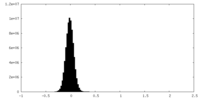
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Type I-C Apo Cascade
| 全体 | 名称: Type I-C Apo Cascade |
|---|---|
| 要素 |
|
-超分子 #1: Type I-C Apo Cascade
| 超分子 | 名称: Type I-C Apo Cascade / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Minimal CRISPR Cascade |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 |
| 分子量 | 理論値: 371 KDa |
-分子 #1: CRISPR-associated protein, CT1134 family
| 分子 | 名称: CRISPR-associated protein, CT1134 family / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 |
| 分子量 | 理論値: 25.250969 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TYGIRLRVWG DYACFTRPEM KVERVSYDVM PPSAARGILE AIHWKPAIRW IVDRIHVLRP IVFDNVRRNE VSSKIPKPNP ATAMRDRKP LYFLVDDGSN RQQRAATLLR NVDYVIEAHF ELTDKAGAED NAGKHLDIFR RRARAGQSFQ QPCLGCREFP A SFELLEGD ...文字列: TYGIRLRVWG DYACFTRPEM KVERVSYDVM PPSAARGILE AIHWKPAIRW IVDRIHVLRP IVFDNVRRNE VSSKIPKPNP ATAMRDRKP LYFLVDDGSN RQQRAATLLR NVDYVIEAHF ELTDKAGAED NAGKHLDIFR RRARAGQSFQ QPCLGCREFP A SFELLEGD VPLSCYAGEK RDLGYMLLDI DFERDMTPLF FKAVMEDGVI TPPSRTSPEV RA UniProtKB: pre-crRNA processing endonuclease |
-分子 #2: CRISPR-associated protein, TM1801 family
| 分子 | 名称: CRISPR-associated protein, TM1801 family / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 |
| 分子量 | 理論値: 32.358912 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTAIANRYEF VLLFDVENGN PNGDPDAGNM PRIDPETGHG LVTDVCLKRK IRNHVALTKE GAERFNIYIQ EKAILNETHE RAYTACDLK PEPKKLPKKV EDAKRVTDWM CTNFYDIRTF GAVMTTEVNC GQVRGPVQMA FARSVEPVVP QEVSITRMAV T TKAEAEKQ ...文字列: MTAIANRYEF VLLFDVENGN PNGDPDAGNM PRIDPETGHG LVTDVCLKRK IRNHVALTKE GAERFNIYIQ EKAILNETHE RAYTACDLK PEPKKLPKKV EDAKRVTDWM CTNFYDIRTF GAVMTTEVNC GQVRGPVQMA FARSVEPVVP QEVSITRMAV T TKAEAEKQ QGDNRTMGRK HIVPYGLYVA HGFISAPLAE KTGFSDEDLT LFWDALVNMF EHDRSAARGL MSSRKLIVFK HQ NRLGNAP AHKLFDLVKV SRAEGSSGPA RSFADYAVTV GQAPEGVEVK EML UniProtKB: CRISPR-associated protein, TM1801 family |
-分子 #3: CRISPR-associated protein, CT1133 family
| 分子 | 名称: CRISPR-associated protein, CT1133 family / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 |
| 分子量 | 理論値: 59.310883 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: WENTSYILGV DAKGKQERTD KCHAAFIAHI KAYCDTADQD LAAVLQFLEH GEKDLSAFPV SEEVIGSNIV FRIEGEPGFV HERPAARQA WANCLNRREQ GLCGQCLITG ERQKPIAQLH PSIKGGRDGV RGAQAVASIV SFNNTAFESY GKEQSINAPV S QEAAFSYV ...文字列: WENTSYILGV DAKGKQERTD KCHAAFIAHI KAYCDTADQD LAAVLQFLEH GEKDLSAFPV SEEVIGSNIV FRIEGEPGFV HERPAARQA WANCLNRREQ GLCGQCLITG ERQKPIAQLH PSIKGGRDGV RGAQAVASIV SFNNTAFESY GKEQSINAPV S QEAAFSYV TALNYLLNPS NRQKVTIADA TVVFWAERSS PAEDIFAGMF DPPSTTAKPE SSNGTPPEDS EEGSQPDTAR DD PHAAARM HDLLVAIRSG KRATDIMPDM DESVRFHVLG LSPNAARLSV RFWEVDTVGH MLDKVGRHYR ELEIIPQFNN EQE FPSLST LLRQTAVLNK TENISPVLAG GLFRAMLTGG PYPQSLLPAV LGRIRAEHAR PEDKSRYRLE VVTYYRAALI KAYL IRNRK LEVPVSLDPA RTDRPYLLGR LFAVLEKAQE DAVPGANATI KDRYLASASA NPGQVFHMLL KNASNHTAKL RKDPE RKGS AIHYEIMMQE IIDNISDFPV TMSSDEQGLF MIGYYHQRKA LFTKKNKEN UniProtKB: CRISPR-associated protein, CT1133 family |
-分子 #4: CRISPR-associated protein, CT1133 family
| 分子 | 名称: CRISPR-associated protein, CT1133 family / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 |
| 分子量 | 理論値: 14.017981 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VSLDPARTDR PYLLGRLFAV LEKAQEDAVP GANATIKDRY LASASANPGQ VFHMLLKNAS NHTAKLRKDP ERKGSAIHYE IMMQEIIDN ISDFPVTMSS DEQGLFMIGY YHQRKALFTK KNKEN UniProtKB: CRISPR-associated protein, CT1133 family |
-分子 #5: RNA (45-MER)
| 分子 | 名称: RNA (45-MER) / タイプ: rna / ID: 5 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 |
| 分子量 | 理論値: 14.457629 KDa |
| 配列 | 文字列: UGGAUUGAAA CGCCAUGCUC AGGCUGGCGA GUGCGCCACU CAUCA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 5400 / 平均電子線量: 33.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)