+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22738 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM reconstruction of SARS-CoV-2 receptor binding domain in complex with the Fab fragment of neutralizing antibody 46 | ||||||||||||||||||
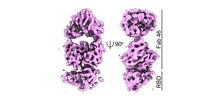
 マップデータ マップデータ | Fab 46 in complex with RBD domain of SARS-CoV-2 Spike protein. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Kucharska I / Tan YZ / Benlekbir S / Rubinstein JL / Julien JP | ||||||||||||||||||
| 資金援助 |  カナダ, 5件 カナダ, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Multivalency transforms SARS-CoV-2 antibodies into ultrapotent neutralizers. 著者: Edurne Rujas / Iga Kucharska / Yong Zi Tan / Samir Benlekbir / Hong Cui / Tiantian Zhao / Gregory A Wasney / Patrick Budylowski / Furkan Guvenc / Jocelyn C Newton / Taylor Sicard / Anthony ...著者: Edurne Rujas / Iga Kucharska / Yong Zi Tan / Samir Benlekbir / Hong Cui / Tiantian Zhao / Gregory A Wasney / Patrick Budylowski / Furkan Guvenc / Jocelyn C Newton / Taylor Sicard / Anthony Semesi / Krithika Muthuraman / Amy Nouanesengsy / Clare Burn Aschner / Katherine Prieto / Stephanie A Bueler / Sawsan Youssef / Sindy Liao-Chan / Jacob Glanville / Natasha Christie-Holmes / Samira Mubareka / Scott D Gray-Owen / John L Rubinstein / Bebhinn Treanor / Jean-Philippe Julien /    要旨: SARS-CoV-2, the virus responsible for COVID-19, has caused a global pandemic. Antibodies can be powerful biotherapeutics to fight viral infections. Here, we use the human apoferritin protomer as a ...SARS-CoV-2, the virus responsible for COVID-19, has caused a global pandemic. Antibodies can be powerful biotherapeutics to fight viral infections. Here, we use the human apoferritin protomer as a modular subunit to drive oligomerization of antibody fragments and transform antibodies targeting SARS-CoV-2 into exceptionally potent neutralizers. Using this platform, half-maximal inhibitory concentration (IC) values as low as 9 × 10 M are achieved as a result of up to 10,000-fold potency enhancements compared to corresponding IgGs. Combination of three different antibody specificities and the fragment crystallizable (Fc) domain on a single multivalent molecule conferred the ability to overcome viral sequence variability together with outstanding potency and IgG-like bioavailability. The MULTi-specific, multi-Affinity antiBODY (Multabody or MB) platform thus uniquely leverages binding avidity together with multi-specificity to deliver ultrapotent and broad neutralizers against SARS-CoV-2. The modularity of the platform also makes it relevant for rapid evaluation against other infectious diseases of global health importance. Neutralizing antibodies are a promising therapeutic for SARS-CoV-2. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Multivalency transforms SARS-CoV-2 antibodies into broad and ultrapotent neutralizers 著者: Rujas E / Kucharska I / Tan YZ / Benlekbir S / Cui H / Zhao T / Wasney G / Budylowski P / Guvenc F / Newton J / Semesi A / Muthuraman K / Nouanesengsy A / Prieto K / Bueler S / Youssef S / ...著者: Rujas E / Kucharska I / Tan YZ / Benlekbir S / Cui H / Zhao T / Wasney G / Budylowski P / Guvenc F / Newton J / Semesi A / Muthuraman K / Nouanesengsy A / Prieto K / Bueler S / Youssef S / Liao-Chan S / Glanville J / Christie-Holmes N / Mubareka S / Gray-Owen SD / Rubinstein JL / Treanor B / Julien JP | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22738.map.gz emd_22738.map.gz | 28.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22738-v30.xml emd-22738-v30.xml emd-22738.xml emd-22738.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22738.png emd_22738.png | 178.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22738 http://ftp.pdbj.org/pub/emdb/structures/EMD-22738 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22738 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22738 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22738_validation.pdf.gz emd_22738_validation.pdf.gz | 352.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22738_full_validation.pdf.gz emd_22738_full_validation.pdf.gz | 352.3 KB | 表示 | |
| XML形式データ |  emd_22738_validation.xml.gz emd_22738_validation.xml.gz | 5.9 KB | 表示 | |
| CIF形式データ |  emd_22738_validation.cif.gz emd_22738_validation.cif.gz | 6.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22738 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22738 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22738 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22738 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22738.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22738.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Fab 46 in complex with RBD domain of SARS-CoV-2 Spike protein. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 receptor binding domain in complex with the Fab fragme...
| 全体 | 名称: SARS-CoV-2 receptor binding domain in complex with the Fab fragment of neutralizing antibody 46 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 receptor binding domain in complex with the Fab fragme...
| 超分子 | 名称: SARS-CoV-2 receptor binding domain in complex with the Fab fragment of neutralizing antibody 46 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Fab fragment of neutralizing antibody 46
| 超分子 | 名称: Fab fragment of neutralizing antibody 46 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: SARS-CoV-2 receptor binding domain
| 超分子 | 名称: SARS-CoV-2 receptor binding domain / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Fab 46 heavy chain
| 分子 | 名称: Fab 46 heavy chain / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLLESGGG LVQPGRSLRL SCAASGFTFS SYAMSWVRQA PGKGLEWVST IYSGGSTYY ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCARGD S RDAFDIWG QGTMVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD YF PEPVTVS WNSGALTSGV ...文字列: EVQLLESGGG LVQPGRSLRL SCAASGFTFS SYAMSWVRQA PGKGLEWVST IYSGGSTYY ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCARGD S RDAFDIWG QGTMVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD YF PEPVTVS WNSGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YIC NVNHKP SNTKVDKKVE PKSC |
-分子 #2: Fab 46 light chain
| 分子 | 名称: Fab 46 light chain / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ITCRASQSIS SWLAWYQQKP GKAPKLLIYD ASNLETGVP SRFSGSGSGT DFTLTISSLQ PEDFATYYCQ QSYSTPFTFG P GTKVDIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KV DNALQSG NSQESVTEQD ...文字列: DIQMTQSPSS LSASVGDRVT ITCRASQSIS SWLAWYQQKP GKAPKLLIYD ASNLETGVP SRFSGSGSGT DFTLTISSLQ PEDFATYYCQ QSYSTPFTFG P GTKVDIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KV DNALQSG NSQESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQG LSSPVT KSFNRGEC |
-分子 #3: Receptor Binding Domain of SARS-CoV-2 Spike
| 分子 | 名称: Receptor Binding Domain of SARS-CoV-2 Spike / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RVQPTESIVR FPNITNLCPF GEVFNATRFA SVYAWNRKRI SNCVADYSVL YNSASFSTFK CYGVSPTKLN DLCFTNVYAD SFVIRGDEVR QIAPGQTGKI ADYNYKLPDD FTGCVIAWNS NNLDSKVGGN YNYLYRLFRK SNLKPFERDI STEIYQAGST PCNGVEGFNC ...文字列: RVQPTESIVR FPNITNLCPF GEVFNATRFA SVYAWNRKRI SNCVADYSVL YNSASFSTFK CYGVSPTKLN DLCFTNVYAD SFVIRGDEVR QIAPGQTGKI ADYNYKLPDD FTGCVIAWNS NNLDSKVGGN YNYLYRLFRK SNLKPFERDI STEIYQAGST PCNGVEGFNC YFPLQSYGFQ PTNGVGYQPY RVVVLSFELL HAPATVCGPK KSTNLVKNKC VNFHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Homemade / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: glow discharged with PELCO easiGlow (Ted Pella) | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 4722 / 平均露光時間: 9.6 sec. / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















