+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22597 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Focused cryo-EM map of rabbit RyR1 central and transmembrane domains in the presence of AMP-PCP in nanodisc | |||||||||||||||||||||
 マップデータ マップデータ | Masked full map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 生物種 |   | |||||||||||||||||||||
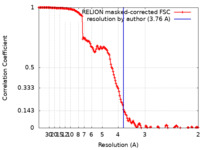
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.76 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Nayak AR / Samso M | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Ca inactivation of the mammalian ryanodine receptor type 1 in a lipidic environment revealed by cryo-EM. 著者: Ashok R Nayak / Montserrat Samsó /  要旨: Activation of the intracellular Ca channel ryanodine receptor (RyR) triggers a cytosolic Ca surge, while elevated cytosolic Ca inhibits the channel in a negative feedback mechanism. Cryogenic ...Activation of the intracellular Ca channel ryanodine receptor (RyR) triggers a cytosolic Ca surge, while elevated cytosolic Ca inhibits the channel in a negative feedback mechanism. Cryogenic electron microscopy of rabbit RyR1 embedded in nanodiscs under partially inactivating Ca conditions revealed an open and a closed-inactivated conformation. Ca binding to the high-affinity site engages the central and C-terminal domains into a block, which pries the S6 four-helix bundle open. Further rotation of this block pushes S6 toward the central axis, closing (inactivating) the channel. Main characteristics of the Ca-inactivated conformation are downward conformation of the cytoplasmic assembly and tightly knit subunit interface contributed by a fully occupied Ca activation site, two inter-subunit resolved lipids, and two salt bridges between the EF hand domain and the S2-S3 loop validated by disease-causing mutations. The structural insight illustrates the prior Ca activation prerequisite for Ca inactivation and provides for a seamless transition from inactivated to closed conformations. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22597.map.gz emd_22597.map.gz | 17.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22597-v30.xml emd-22597-v30.xml emd-22597.xml emd-22597.xml | 25.1 KB 25.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22597_fsc.xml emd_22597_fsc.xml | 15.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22597.png emd_22597.png | 195.4 KB | ||
| マスクデータ |  emd_22597_msk_1.map emd_22597_msk_1.map | 307.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22597_additional_1.map.gz emd_22597_additional_1.map.gz emd_22597_additional_2.map.gz emd_22597_additional_2.map.gz emd_22597_half_map_1.map.gz emd_22597_half_map_1.map.gz emd_22597_half_map_2.map.gz emd_22597_half_map_2.map.gz | 238.9 MB 239.3 MB 16 MB 16 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22597 http://ftp.pdbj.org/pub/emdb/structures/EMD-22597 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22597 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22597 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22597_validation.pdf.gz emd_22597_validation.pdf.gz | 446.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22597_full_validation.pdf.gz emd_22597_full_validation.pdf.gz | 446.2 KB | 表示 | |
| XML形式データ |  emd_22597_validation.xml.gz emd_22597_validation.xml.gz | 23.2 KB | 表示 | |
| CIF形式データ |  emd_22597_validation.cif.gz emd_22597_validation.cif.gz | 30.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22597 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22597 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22597 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22597 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22597.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22597.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked full map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22597_msk_1.map emd_22597_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
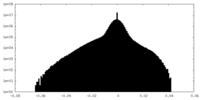
| 密度ヒストグラム |
-追加マップ: Unmasked and unfiltered half map1 up to Nyquist frequency
| ファイル | emd_22597_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked and unfiltered half map1 up to Nyquist frequency | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
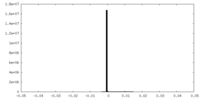
| 密度ヒストグラム |
-追加マップ: Unmasked and unfiltered half map2 up to Nyquist frequency
| ファイル | emd_22597_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked and unfiltered half map2 up to Nyquist frequency | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
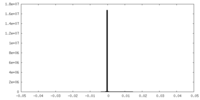
| 密度ヒストグラム |
-ハーフマップ: Masked half map2
| ファイル | emd_22597_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
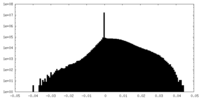
| 密度ヒストグラム |
-ハーフマップ: Masked half map1
| ファイル | emd_22597_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rabbit RyR1 with AMP-PCP
| 全体 | 名称: Rabbit RyR1 with AMP-PCP |
|---|---|
| 要素 |
|
-超分子 #1: Rabbit RyR1 with AMP-PCP
| 超分子 | 名称: Rabbit RyR1 with AMP-PCP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Purified RyR1 was reconstituted with membrane scaffold protein, MSP1E3D1, and POPC. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 2.26 MDa |
-分子 #1: RyR1
| 分子 | 名称: RyR1 / タイプ: protein_or_peptide / ID: 1 詳細: Rabbit RyR1 corresponding to amino acid sequence 3668-5037 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: SFEDRMIDDL SKAGEQEEEE EEVEEKKPDP LHQLVLHFSR TALTEKSKLD EDYLYMAYAD IMAKSCHLEE GGENEEVEV SFEEKEMEKQ RLLYQQSRLH TRGAAEMVLQ MISACKGETG AMVSSTLKLG ISILNGGNAE V QQKMLDYL KDKKEVGFFQ SIQALMQTCS ...文字列: SFEDRMIDDL SKAGEQEEEE EEVEEKKPDP LHQLVLHFSR TALTEKSKLD EDYLYMAYAD IMAKSCHLEE GGENEEVEV SFEEKEMEKQ RLLYQQSRLH TRGAAEMVLQ MISACKGETG AMVSSTLKLG ISILNGGNAE V QQKMLDYL KDKKEVGFFQ SIQALMQTCS VLDLNAFERQ NKAEGLGMVN EDGTVINRQN GEKVMADDEF TQ DLFRFLQ LLCEGHNNDF QNYLRTQTGN TTTINIIICT VDYLLRLQES ISDFYWYYSG KDVIEEQGKR NFS KAMSVA KQVFNSLTEY IQGPCTGNQQ SLAHSRLWDA VVGFLHVFAH MMMKLAQDSS QIELLKELLD LQKD MVVML LSLLEGNVVN GMIARQMVDM LVESSSNVEM ILKFFDMFLK LKDIVGSEAF QDYVTDPRGL ISKKD FQKA MDSQKQFTGP EIQFLLSCSE ADENEMINFE EFANRFQEPA RDIGFNVAVL LTNLSEHV P HDPRLRNFLE LAESILEYFR PYLGRAEIMG ASRRIERIYF EISETNRAQW EMPQVKESKR QFIFDVVNE GGEAEKMELF VSFCEDTIFE MQIAAQISEF WGELEVQRVK FLNYLSRNFY TLRFLALFLA FAINFILLF YKVSDSPPAN MVYYFLEEST GYMEPALWCL SLLHTLVAFL CIIGYNCLKV PLVIFKREKE LARKLEFDGL YITEQPGDD DVKGQWDRLV LNTPSFPSNY WDKFVKRKVL DKHGDIFGRE RIAELLGMDL ASLEITAHNE R KPDPPPGL LTWLMSIDVK YQIWKFGVIF TDNSFLYLGW YMVMSLLGHY NNFFFAAHLL DIAMGVKTLR TI LSSVTHN GKQLVMTVGL LAVVVYLYTV VAFNFFRKFY NKSEDEDEPD MKCDDMMTCY LFHMYVGVRA GGG IGDEIE DPAGDEYELY RVVFDITFFF FVIVILLAII QGLIIDAFGE LRDQQEQVKE DMETKCFICG IGSD YFDTT PHGFETHTLE EHNLANYMFF LMYLINKDET EHTGQESYVW KMYQERCWDF FPAGDCFRKQ YEDQL S |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.35 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Sample was blotted for 1 second on both sides with Whatman hardened ashless filter paper with blot force 2.. | |||||||||||||||||||||
| 詳細 | Purified RyR1 was reconstituted with membrane scaffold protein, MSP1E3D1, and POPC in 1:2:50 molar ratio. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 1959 / 平均電子線量: 70.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)