+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22447 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Plant Mitochondrial complex III2 from Vigna radiata | |||||||||
 マップデータ マップデータ | Plant Mitochondrial complex IV from Vigna radiata | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | mitochondria / respiration / bioenergetics / plants / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報respiratory chain complex IV / mitochondrial envelope / mitochondrial electron transport, cytochrome c to oxygen / cytochrome-c oxidase activity / ATP synthesis coupled electron transport / enzyme regulator activity / mitochondrial inner membrane / copper ion binding / mitochondrion / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Vigna radiata (リョクトウ) / Vigna radiata (リョクトウ) /  Vigna radiata var. radiata (リョクトウ) Vigna radiata var. radiata (リョクトウ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Maldonado M / Letts JA | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Atomic structures of respiratory complex III, complex IV, and supercomplex III-IV from vascular plants. 著者: Maria Maldonado / Fei Guo / James A Letts /  要旨: Mitochondrial complex III (CIII) and complex IV (CIV), which can associate into a higher-order supercomplex (SC III+IV), play key roles in respiration. However, structures of these plant complexes ...Mitochondrial complex III (CIII) and complex IV (CIV), which can associate into a higher-order supercomplex (SC III+IV), play key roles in respiration. However, structures of these plant complexes remain unknown. We present atomic models of CIII, CIV, and SC III+IV from determined by single-particle cryoEM. The structures reveal plant-specific differences in the MPP domain of CIII and define the subunit composition of CIV. Conformational heterogeneity analysis of CIII revealed long-range, coordinated movements across the complex, as well as the motion of CIII's iron-sulfur head domain. The CIV structure suggests that, in plants, proton translocation does not occur via the H channel. The supercomplex interface differs significantly from that in yeast and bacteria in its interacting subunits, angle of approach and limited interactions in the mitochondrial matrix. These structures challenge long-standing assumptions about the plant complexes and generate new mechanistic hypotheses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22447.map.gz emd_22447.map.gz | 480.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22447-v30.xml emd-22447-v30.xml emd-22447.xml emd-22447.xml | 30.3 KB 30.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22447_fsc.xml emd_22447_fsc.xml | 18.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22447.png emd_22447.png | 84.6 KB | ||
| マスクデータ |  emd_22447_msk_1.map emd_22447_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22447.cif.gz emd-22447.cif.gz | 7.8 KB | ||
| その他 |  emd_22447_half_map_1.map.gz emd_22447_half_map_1.map.gz emd_22447_half_map_2.map.gz emd_22447_half_map_2.map.gz | 411.5 MB 411.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22447 http://ftp.pdbj.org/pub/emdb/structures/EMD-22447 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22447 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22447 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22447_validation.pdf.gz emd_22447_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22447_full_validation.pdf.gz emd_22447_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_22447_validation.xml.gz emd_22447_validation.xml.gz | 25.9 KB | 表示 | |
| CIF形式データ |  emd_22447_validation.cif.gz emd_22447_validation.cif.gz | 34.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22447 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22447 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22447 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22447 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7jroMC  7jrgC  7jrpC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10586 (タイトル: Cryo electron micrographs of digitonin-solubilized, amphipol-stabilized, sucrose-gradient-purified V. radiata mitochondrial membranes - mixed fraction containing CI*, CIII2 and SC III2+IV EMPIAR-10586 (タイトル: Cryo electron micrographs of digitonin-solubilized, amphipol-stabilized, sucrose-gradient-purified V. radiata mitochondrial membranes - mixed fraction containing CI*, CIII2 and SC III2+IVData size: 6.7 TB Data #1: Raw movies of mixed sample used for determination of mung bean respiratory CI*, CIII2, CIV and SC III2+IV [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22447.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22447.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Plant Mitochondrial complex IV from Vigna radiata | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8332 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
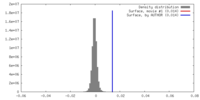
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22447_msk_1.map emd_22447_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
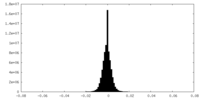
| 密度ヒストグラム |
-ハーフマップ: Plant Mitochondrial complex IV from Vigna radiata. Half map 1.
| ファイル | emd_22447_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Plant Mitochondrial complex IV from Vigna radiata. Half map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
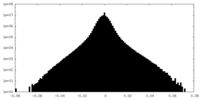
| 密度ヒストグラム |
-ハーフマップ: Plant Mitochondrial complex IV from Vigna radiata. Half map 2.
| ファイル | emd_22447_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Plant Mitochondrial complex IV from Vigna radiata. Half map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
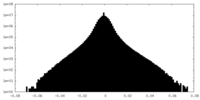
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Mitochondrial Respiratory Complex IV
+超分子 #1: Mitochondrial Respiratory Complex IV
+分子 #1: COX1
+分子 #2: Cytochrome c oxidase subunit 2
+分子 #3: COX3
+分子 #4: COX4
+分子 #5: cytochrome c oxidase subunit 5b-2, mitochondrial
+分子 #6: cytochrome c oxidase subunit 6a, mitochondrial
+分子 #7: cytochrome c oxidase subunit 6b-1
+分子 #8: Cytochrome c oxidase subunit 5C
+分子 #9: COX7a
+分子 #10: COX7c
+分子 #11: HEME-A
+分子 #12: COPPER (II) ION
+分子 #13: MAGNESIUM ION
+分子 #14: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
+分子 #15: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #16: CARDIOLIPIN
+分子 #17: DINUCLEAR COPPER ION
+分子 #18: ZINC ION
+分子 #19: LYSINE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 288 K / 装置: FEI VITROBOT MARK III | |||||||||||||||
| 詳細 | This sample was monodisperse on size exclusion chromatography but was a mixture of different mitochondrial respiratory complexes |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 9816 / 平均露光時間: 3.0 sec. / 平均電子線量: 86.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 60010 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)