+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22384 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full-length three-dimensional structure of the influenza A virus M1 protein and its organization into a matrix layer | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | influenza A / virus / matrix layer / M1 protein / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Assembly of Viral Components at the Budding Site / Influenza Infection / Fusion of the Influenza Virion to the Host Cell Endosome / Release / Budding / Packaging of Eight RNA Segments / Uncoating of the Influenza Virion / Entry of Influenza Virion into Host Cell via Endocytosis / Viral RNP Complexes in the Host Cell Nucleus / NEP/NS2 Interacts with the Cellular Export Machinery ...Assembly of Viral Components at the Budding Site / Influenza Infection / Fusion of the Influenza Virion to the Host Cell Endosome / Release / Budding / Packaging of Eight RNA Segments / Uncoating of the Influenza Virion / Entry of Influenza Virion into Host Cell via Endocytosis / Viral RNP Complexes in the Host Cell Nucleus / NEP/NS2 Interacts with the Cellular Export Machinery / Viral mRNA Translation / viral budding from plasma membrane / structural constituent of virion / host cell nucleus / virion membrane / RNA binding / extracellular region / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) / Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) /  Influenza A virus (strain A/Puerto Rico/8/1934 H1N1) (A型インフルエンザウイルス) Influenza A virus (strain A/Puerto Rico/8/1934 H1N1) (A型インフルエンザウイルス) | ||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Su Z / Pintilie G | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: PLoS Biol / 年: 2020 ジャーナル: PLoS Biol / 年: 2020タイトル: Full-length three-dimensional structure of the influenza A virus M1 protein and its organization into a matrix layer. 著者: Lisa Selzer / Zhaoming Su / Grigore D Pintilie / Wah Chiu / Karla Kirkegaard /   要旨: Matrix proteins are encoded by many enveloped viruses, including influenza viruses, herpes viruses, and coronaviruses. Underneath the viral envelope of influenza virus, matrix protein 1 (M1) forms an ...Matrix proteins are encoded by many enveloped viruses, including influenza viruses, herpes viruses, and coronaviruses. Underneath the viral envelope of influenza virus, matrix protein 1 (M1) forms an oligomeric layer critical for particle stability and pH-dependent RNA genome release. However, high-resolution structures of full-length monomeric M1 and the matrix layer have not been available, impeding antiviral targeting and understanding of the pH-dependent transitions involved in cell entry. Here, purification and extensive mutagenesis revealed protein-protein interfaces required for the formation of multilayered helical M1 oligomers similar to those observed in virions exposed to the low pH of cell entry. However, single-layered helical oligomers with biochemical and ultrastructural similarity to those found in infectious virions before cell entry were observed upon mutation of a single amino acid. The highly ordered structure of the single-layered oligomers and their likeness to the matrix layer of intact virions prompted structural analysis by cryo-electron microscopy (cryo-EM). The resulting 3.4-Å-resolution structure revealed the molecular details of M1 folding and its organization within the single-shelled matrix. The solution of the full-length M1 structure, the identification of critical assembly interfaces, and the development of M1 assembly assays with purified proteins are crucial advances for antiviral targeting of influenza viruses. #1:  ジャーナル: Plos Biol. / 年: 2020 ジャーナル: Plos Biol. / 年: 2020タイトル: Full-length three-dimensional structure of the influenza A virus M1 protein and its organization into a matrix layer 著者: Selzer L / Su Z / Pintilie G / Chiu W / Kirkegaard K | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22384.map.gz emd_22384.map.gz | 71 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22384-v30.xml emd-22384-v30.xml emd-22384.xml emd-22384.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22384.png emd_22384.png | 287.8 KB | ||
| Filedesc metadata |  emd-22384.cif.gz emd-22384.cif.gz | 6.3 KB | ||
| その他 |  emd_22384_additional.map.gz emd_22384_additional.map.gz emd_22384_additional_1.map.gz emd_22384_additional_1.map.gz | 658.3 KB 658.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22384 http://ftp.pdbj.org/pub/emdb/structures/EMD-22384 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22384 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22384 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22384_validation.pdf.gz emd_22384_validation.pdf.gz | 558.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22384_full_validation.pdf.gz emd_22384_full_validation.pdf.gz | 558.4 KB | 表示 | |
| XML形式データ |  emd_22384_validation.xml.gz emd_22384_validation.xml.gz | 7.7 KB | 表示 | |
| CIF形式データ |  emd_22384_validation.cif.gz emd_22384_validation.cif.gz | 8.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22384 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22384 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22384 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22384 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22384.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22384.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Segmented M1 protein
| ファイル | emd_22384_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Segmented M1 protein | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
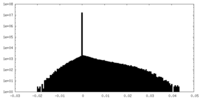
| 密度ヒストグラム |
-追加マップ: Segmented M1 protein
| ファイル | emd_22384_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Segmented M1 protein | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
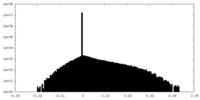
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : M1-V97K oligomer
| 全体 | 名称: M1-V97K oligomer |
|---|---|
| 要素 |
|
-超分子 #1: M1-V97K oligomer
| 超分子 | 名称: M1-V97K oligomer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) |
| 分子量 | 理論値: 143 kDa/nm |
-分子 #1: Matrix protein 1
| 分子 | 名称: Matrix protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (strain A/Puerto Rico/8/1934 H1N1) (A型インフルエンザウイルス) Influenza A virus (strain A/Puerto Rico/8/1934 H1N1) (A型インフルエンザウイルス)株: A/Puerto Rico/8/1934 H1N1 |
| 分子量 | 理論値: 27.827152 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SLLTEVETYV LSIIPSGPLK AEIAQRLEDV FAGKNTDLEV LMEWLKTRPI LSPLTKGILG FVFTLTVPSE RGLQRRRFVQ NALNGNGDP NNMDKAKKLY RKLKREITFH GAKEISLSYS AGALASCMGL IYNRMGAVTT EVAFGLVCAT CEQIADSQHR S HRQMVTTT ...文字列: SLLTEVETYV LSIIPSGPLK AEIAQRLEDV FAGKNTDLEV LMEWLKTRPI LSPLTKGILG FVFTLTVPSE RGLQRRRFVQ NALNGNGDP NNMDKAKKLY RKLKREITFH GAKEISLSYS AGALASCMGL IYNRMGAVTT EVAFGLVCAT CEQIADSQHR S HRQMVTTT NPLIRHENRM VLASTTAKAM EQMAGSSEQA AEAMEVASQA RQMVQAMRTI GTHPSSSAGL KNDLLENLQA YQ KRMGVQM QRFK UniProtKB: Matrix protein 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 3 seconds once before plunging.. | |||||||||
| 詳細 | Influenza A virus matrix protein with V97K mutation assembled in vitro |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 78.0 K / 最高: 78.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 2 / 実像数: 440 / 平均露光時間: 4.0 sec. / 平均電子線量: 36.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 4.3 µm / 最小 デフォーカス(補正後): 0.3 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用したクラス数: 200 想定した対称性 - らせんパラメータ - Δz: 1.96 Å 想定した対称性 - らせんパラメータ - ΔΦ: 17.1 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0.6) / 使用した粒子像数: 56602 |
|---|---|
| Segment selection | 選択した数: 2268 / ソフトウェア - 名称: EMAN2 (ver. 2.23) |
| 初期モデル | モデルのタイプ: INSILICO MODEL / In silico モデル: EMAN2.23 詳細: Created a cylindrical density map in EMAN2 and used it as an initial model. |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: RELION (ver. 3.0.6) |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)