| 登録情報 | データベース: EMDB / ID: EMD-22243
|
|---|
| タイトル | Full-length Hsc82 in complex with two Aha1 CTD in the presence of ATPgammaS |
|---|
 マップデータ マップデータ | Hsc82 in complex with two Aha1 CTD in the presence of ATPgammaS |
|---|
 試料 試料 | - 複合体: Full-length Hsc82 in complex with Aha1-CTD in the presence of ATPgammaS
- タンパク質・ペプチド: ATP-dependent molecular chaperone HSC82
- タンパク質・ペプチド: Hsp90 co-chaperone AHA1
- リガンド: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
- リガンド: MAGNESIUM ION
- リガンド: POTASSIUM ION
|
|---|
 キーワード キーワード | Co-chaperone / activator / CHAPERONE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / eNOS activation / Extra-nuclear estrogen signaling / HSF1-dependent transactivation / VEGFR2 mediated vascular permeability / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / HSF1 activation / response to oxygen levels / box C/D snoRNP assembly / ATPase activator activity ...Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / eNOS activation / Extra-nuclear estrogen signaling / HSF1-dependent transactivation / VEGFR2 mediated vascular permeability / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / HSF1 activation / response to oxygen levels / box C/D snoRNP assembly / ATPase activator activity / proteasome assembly / Neutrophil degranulation / telomere maintenance / ATP-dependent protein folding chaperone / protein import into nucleus / unfolded protein binding / protein folding / protein-folding chaperone binding / cellular response to heat / protein stabilization / perinuclear region of cytoplasm / protein-containing complex / ATP hydrolysis activity / mitochondrion / ATP binding / nucleus / plasma membrane / cytoplasm / cytosol類似検索 - 分子機能 Activator of Hsp90 ATPase, N-terminal / Activator of Hsp90 ATPase, Aha1 / Activator of Hsp90 ATPase, N-terminal / Activator of Hsp90 ATPase, N-terminal / Activator of Hsp90 ATPase homologue 1-like / Activator of Hsp90 ATPase homolog 1-like protein / START-like domain superfamily / Heat shock protein Hsp90, conserved site / Heat shock hsp90 proteins family signature. / HSP90, C-terminal domain ...Activator of Hsp90 ATPase, N-terminal / Activator of Hsp90 ATPase, Aha1 / Activator of Hsp90 ATPase, N-terminal / Activator of Hsp90 ATPase, N-terminal / Activator of Hsp90 ATPase homologue 1-like / Activator of Hsp90 ATPase homolog 1-like protein / START-like domain superfamily / Heat shock protein Hsp90, conserved site / Heat shock hsp90 proteins family signature. / HSP90, C-terminal domain / Heat shock protein Hsp90, N-terminal / Heat shock protein Hsp90 family / Hsp90 protein / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase superfamily / Ribosomal protein S5 domain 2-type fold類似検索 - ドメイン・相同性 ATP-dependent molecular chaperone HSC82 / Hsp90 co-chaperone AHA1類似検索 - 構成要素 |
|---|
| 生物種 |   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
|---|
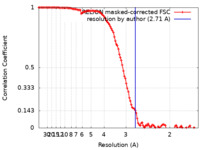
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.71 Å |
|---|
 データ登録者 データ登録者 | Liu YX / Sun M |
|---|
| 資金援助 |  米国, 5件 米国, 5件 | Organization | Grant number | 国 |
|---|
| Howard Hughes Medical Institute (HHMI) | |  米国 米国 | | American Heart Association | |  米国 米国 | | National Institutes of Health/National Cancer Institute (NIH/NCI) | U54CA209891 |  米国 米国 | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | S10OD020054 |  米国 米国 | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | S10OD021741 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Cryo-EM structures reveal a multistep mechanism of Hsp90 activation by co-chaperone Aha1
著者: Liu YX / Sun M / Myasnikov AG / Elnatan D / Agard DA |
|---|
| 履歴 | | 登録 | 2020年6月28日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2021年6月30日 | - |
|---|
| マップ公開 | 2021年6月30日 | - |
|---|
| 更新 | 2024年3月6日 | - |
|---|
| 現状 | 2024年3月6日 | 処理サイト: RCSB / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 米国, 5件
米国, 5件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_22243.map.gz
emd_22243.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-22243-v30.xml
emd-22243-v30.xml emd-22243.xml
emd-22243.xml EMDBヘッダ
EMDBヘッダ emd_22243_fsc.xml
emd_22243_fsc.xml FSCデータファイル
FSCデータファイル emd_22243.png
emd_22243.png emd-22243.cif.gz
emd-22243.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-22243
http://ftp.pdbj.org/pub/emdb/structures/EMD-22243 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22243
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22243 emd_22243_validation.pdf.gz
emd_22243_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_22243_full_validation.pdf.gz
emd_22243_full_validation.pdf.gz emd_22243_validation.xml.gz
emd_22243_validation.xml.gz emd_22243_validation.cif.gz
emd_22243_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22243
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22243 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22243
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22243 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_22243.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_22243.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素





 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー
































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)