+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21911 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
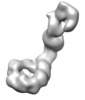
| タイトル | Mdn1-DeltaC alone | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 23.56 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Mickolajczyk KJ / Niu Y | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: Long-range intramolecular allostery and regulation in the dynein-like AAA protein Mdn1. 著者: Keith J Mickolajczyk / Paul Dominic B Olinares / Yiming Niu / Nan Chen / Sara E Warrington / Yusuke Sasaki / Thomas Walz / Brian T Chait / Tarun M Kapoor /  要旨: Mdn1 is an essential mechanoenzyme that uses the energy from ATP hydrolysis to physically reshape and remodel, and thus mature, the 60S subunit of the ribosome. This massive (>500 kDa) protein has an ...Mdn1 is an essential mechanoenzyme that uses the energy from ATP hydrolysis to physically reshape and remodel, and thus mature, the 60S subunit of the ribosome. This massive (>500 kDa) protein has an N-terminal AAA (ATPase associated with diverse cellular activities) ring, which, like dynein, has six ATPase sites. The AAA ring is followed by large (>2,000 aa) linking domains that include an ∼500-aa disordered (D/E-rich) region, and a C-terminal substrate-binding MIDAS domain. Recent models suggest that intramolecular docking of the MIDAS domain onto the AAA ring is required for Mdn1 to transmit force to its ribosomal substrates, but it is not currently understood what role the linking domains play, or why tethering the MIDAS domain to the AAA ring is required for protein function. Here, we use chemical probes, single-particle electron microscopy, and native mass spectrometry to study the AAA and MIDAS domains separately or in combination. We find that Mdn1 lacking the D/E-rich and MIDAS domains retains ATP and chemical probe binding activities. Free MIDAS domain can bind to the AAA ring of this construct in a stereo-specific bimolecular interaction, and, interestingly, this binding reduces ATPase activity. Whereas intramolecular MIDAS docking appears to require a treatment with a chemical inhibitor or preribosome binding, bimolecular MIDAS docking does not. Hence, tethering the MIDAS domain to the AAA ring serves to prevent, rather than promote, MIDAS docking in the absence of inducing signals. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21911.map.gz emd_21911.map.gz | 10.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21911-v30.xml emd-21911-v30.xml emd-21911.xml emd-21911.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21911.png emd_21911.png | 32.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21911 http://ftp.pdbj.org/pub/emdb/structures/EMD-21911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21911 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21911_validation.pdf.gz emd_21911_validation.pdf.gz | 78 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21911_full_validation.pdf.gz emd_21911_full_validation.pdf.gz | 77.1 KB | 表示 | |
| XML形式データ |  emd_21911_validation.xml.gz emd_21911_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21911 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21911 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21911.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21911.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.71 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mdn1(1-3911)
| 全体 | 名称: Mdn1(1-3911) |
|---|---|
| 要素 |
|
-超分子 #1: Mdn1(1-3911)
| 超分子 | 名称: Mdn1(1-3911) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Map with no bound MIDAS protein |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  組換株: Hi5 |
| 分子量 | 実験値: 453 KDa |
-分子 #1: Mdn1(1-3911)
| 分子 | 名称: Mdn1(1-3911) / タイプ: protein_or_peptide / ID: 1 / 詳細: Rbin-1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTENL YFQGAMGIRN SKAYVDMDVL IEWVAIYPQI YDILEHINYV PSNTLQRLRL HQPWSKIDYD VWFLYASDEI RETCKVKYYG ETKTYGEVFV LENERISQLH RLFVSWTVSE RAEHLKNLLF DAGLSNLPLV ELGGNVFFNS HVPLPCSLVL ...文字列: MSYYHHHHHH DYDIPTTENL YFQGAMGIRN SKAYVDMDVL IEWVAIYPQI YDILEHINYV PSNTLQRLRL HQPWSKIDYD VWFLYASDEI RETCKVKYYG ETKTYGEVFV LENERISQLH RLFVSWTVSE RAEHLKNLLF DAGLSNLPLV ELGGNVFFNS HVPLPCSLVL TKSTQENLNR ITPYLVQKRP ILLAGPEGIG KKFLITQIAA KLGQQIIRIH LSDSTDPKML IGTYTSPKPG EFEWQPGVLT QAVITGKWIL FTNIEHAPSE VLSVLLPLLE KRQLVIPSRG ETIYAKGSFQ MFATSSMKTK ILGQRLWQIL DLTYQPDECV EVVSTLYPVL SIICPTLYSV YKDIFDLFSQ RSFLATSKIY RRLCLRDFYK FIKRVAFLYH KFMIPSDHVV ISQELQDAVF KEAIDMFGAF IPSRDGFDLV VRNVAIELNI PPEKALQLRY SIPVFQNLEH NINIGRCSLK KLSTIRSCST NSYAFTSSSL GLLEQLAAGV QTNEPLLLVG ETGTGKTTTI QLLAGLLGQK VTVINMSQQT ESSDMLGGYK PINASTLGLP LHERFIDIFE QTFSSKKNAK FISMASTSAR RFRWKTCLKI WKEACKLSKT VLDGQQPLPN PQKRQKRLSN QVELRNQWAK FEKEVEDFEK VLTGGSNGFM FSFVEGALVK AVRSGHWVLL DEINLASLET LEPIGQLLSS YESGILLSER GDITPITPHK NFRLFGCMNP STDVGKRELE PSFRSRFTEI YVHSPDQNLD DLLSIIQKYI GSLCIGNEHV IREVAELYQV AKSLSLDGSL VDGAGQRPHY TVRTLSRTLS YVTEIAPIYG LRRSLYEGFC MSFLTLLDHT SESLLYNHVV RFTLGELNRD QQNAILKQIP KVPDHSSYIA FCHYWLRRGS FPVEEQEHYI ITPFVQKNLL NIARACSTRM FPILIQGPTS SGKTSMIEYV AKKTGHKFVR INNHEHTDLQ EYIGTYVTDD NGSLSFREGV LVEALRNGYW IVLDELNLAP TDVLEALNRL LDDNRELFIP ETQVLVKPHP EFMLFATQNP PGVYAGRKHL SRAFRNRFLE IHFDDIPENE LETILHKRCK IAPSYAAKIV QVFRELSLRR QTTRIFEQKN SFATLRDLFR WAFREAVGYQ QLAENGYMLL AERARDQKDK LAVQEVIEKV MKVKIDTDGI YNLDSMEIFQ DMSLKEGPLS KVVWTRPMIR LFCLVWRCLL AKEPVLLVGD TGCGKTTVCQ ILAECLHKEL HIINAHQDTE NGDIIGAQRP VRNRSAVNYS LHSQLCEKFN VQESLDSIDD LIEKFEKLSS SEKNDNLSNL IERQIIKYRS LFEWHDGALV TAMKQGDFFL LDEISLADDS VLERLNSVLE LSRTLTLVEH SNAAVSLTAK DGFAFFATMN PGGDYGKKEL SPALRNRFTE IWVPPMVDTE DILKIVEGKL HNNKIELARP LVEYAKWHAN EYLYTDVISI RDVLSAVEFI NACEILDLNL VLFNAVSMVF IDALGSFTTF SLSNNLASLH AERQRCFAKL NELAGSNIMA SKSADISIKF SDSSFFIGDF GIPLGDSVES DSTYSLHTDT TLMNASKVLR ALQVLKPILL EGSPGVGKTS LITALARETG HQLVRINLSD QTDLMDLFGS DVPVEGGEGG QFAWRDAPFL AAMRNGHWVL LDELNLASQS VLEGLNACLD HRNEAYIPEL DKVFKAHPNF RVFAAQNPQH QGGGRKGLPR SFINRFSVVY VEALKEKDMI EIAACNYHQV NEDWRLKIIK FMFRLQDNIE KDISFGSFGS PWEFNLRDTL RWLQLLNDAP KYTCVSPADY LEVMVLHRMR TVEDRVRTCE LFKEVFDIDY EPRTIGFSLS SQCFKVGHSL LVRDVERQKT LLDSQNILQS QLPVLESVIT CINKKWPCIL VGDTATGKTC ILRLLAAIAG AKIKEMAVNS DTDTMDLIGE YEQIDISRKA SELFTDLSQQ LLNIVIKYRN FDNIFRETSL YTLTTTSFKT HSQAFTLLQK VVDQLDQLKI HETLVHSLGD IHEKARKLLA EFSASPAGRF EWFDGYLLKA VEEGHWFVLD NANLCSPAVL DRLNSLLEHK GVLIVNEKTT EDGHPKTIKP HPNFRLFLTV NPVYGELSRA MRNRGVEIFL LKEALTEIDK KQMSLLEPAP ISSAVDTLAS NISYIKYVFE TMGKIEIDGN YMYIAHAIIL ALFSPRQLKL LRKVLLTNPQ FSLSIKADAE LLLTLKNLVQ KIYCADYFNH MDLKASRFMD IYEYPVQLRE VVGLIQTIND FQSVILTSHL ELPETYASGL LFVSAHEILD LTEEVNRLAV STSNSTYLLK SASAVYHNVS SFKGSTPSLW NLLNQFSKFL IEIASANSNI VYKLSYDVIR HFLKLVVLWK NIYVWTNVPD CDISRFYCYT KMLGEWMFTL TEKTKLLESF LPKDSLEKFS ELQNLSTGLH MQAIWDKWHA FVPRTYDQWS LWNTVDKLLT QYVNANIPSI SMETTACEVV GTSLSLLNKV LVENEVGDIY SYLKILGKGV NELKSSKQVI LPENLVNLFN CLASLDLLHI FIKYTTSSFF LTDDFVRFIR VCFHSRISGN LLTLLHGISF DSTKAVAPVL TYFDFCSLTT GNILGRIALA FTSIDENANL ESANIFEHAR LALLQHFMDH SSLLAEDSST KMNLILLQRY AVIISIFLDQ GKCEKANDLI TKLSLPYEEL AENFVSILEA CKAFLVANSE FISYTYTERF IHSLRFLKDS WLSSNQQKML KNQGMAYIYF ASGMLLVYVP DKPFDPALLP LLTVESLRHY LESLYKESQI LEIAESLNSG KVNSVMRRLV STEISNTPNI DSSFSTVYRS LNESIVPLYS ELEFFMKSVV LNQYIFELAM RLSKESNIAV VEEAKSFVTK WKAYIERIRE AYPQFVDVYE LILSFISFMI YGIELLMFEA KRRLDERSQI LSTLILTLVD PSSFARSLSF DDVSNLIEQI KVLDLNDSIR FEIYLFLASR LCSEKQHSSD THSLANSFVL LANEFYIHNA KIKQKELEEI EEKNRLYRQR EFNFDKNDYL KVFINYDDEV EPEVEPEVVI ERKRFLQLQF AFWSLYNEIY SEKMNVIPLE QLMNTGSYLA KKIKVKNPDM IASSGFDIVS VVLMMGVKST NERQYWTPPV YNFYSDPNPS KAIEVRDLIK IVESRAISLI KNWPENFVLR GLKDAIDAIL NLSPFSPIAE YLSKLERVFH LLSEWEKLAS REYSLANEMD LIKKKIIDWR KFELSNWNNL LKLEEYKLSE RVYPRLYSIL QFIILKPFFE NSKFTKQNLC ESASIIVQFI TDLTVGEFQL CLKCLLSFSQ HAASLRICHG IDAMLLNIYH YFEQFLSKVS EAIHTQKQSL ENSIKERILL MSWKDTNVYA LKESAKKSHA ELFKVLHRYR EVLRQPVSSY LSQKHDWDSL LDTENNSAMW VAKKVNLSPS YIEKMDTEIM KLVPVRFSNT PTTLRLMWTL FANVEKPGST FTNMVSNLIT DARELMKLTP ETINDDNLSE IKHLKSRKHL LLTETFKTLK AFGLQYRVKA GIEENLSNLR NLLAVIPTFP VTSLSIEKVD RSLMKSLDFI PKFQTLAGHQ HNDLSVPEVQ KGVGLFNSML SLQLGERAQL VEFTNELLAL KNVYSEVGVN GSPLESFNNS SFNEVSSLGY DHDFENRAQA VSMLCQIYAI VIQKHSSISP TASFQSIGHE LSRFADLLSN KLFPSSIPLY ASADKVSSIR DQQKGINDLI EYCRKKRTEL PELSYCFKHL VSLQSLKSIS RTQVDLTNDE FLNLMNFVLN LFDSLLSSIE TATKNMRTFK ELAETSSFIE MSSCFSKVLR AFNLKFQSMK LSSLKEKLRS SSVDKMSCQL LMLFLPVCEQ FINLAESVLD YFINVHNSNL DSLSKISTLF FMVANNGFCS PDLPQEGKSN SGELESG |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl formate |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM10 |
|---|---|
| 撮影 | フィルム・検出器のモデル: OTHER / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 100 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















