+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21896 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full-length human ENaC ECD | |||||||||||||||
 マップデータ マップデータ | ENaC_FL sharpened EM map (Bfactor -101.4) | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | sodium channel / blood pressure / epithelial / salt transport / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sensory perception of salty taste / Sensory perception of salty taste / sensory perception of sour taste / neutrophil-mediated killing of bacterium / aldosterone metabolic process / leukocyte activation involved in inflammatory response / cellular response to vasopressin / sperm principal piece / sodium channel complex / epithelial fluid transport ...sensory perception of salty taste / Sensory perception of salty taste / sensory perception of sour taste / neutrophil-mediated killing of bacterium / aldosterone metabolic process / leukocyte activation involved in inflammatory response / cellular response to vasopressin / sperm principal piece / sodium channel complex / epithelial fluid transport / mucus secretion / sodium ion homeostasis / cellular response to aldosterone / renal system process / artery smooth muscle contraction / neutrophil activation involved in immune response / multicellular organismal-level water homeostasis / potassium ion homeostasis / intracellular sodium ion homeostasis / cellular response to acidic pH / sodium ion import across plasma membrane / ligand-gated sodium channel activity / motile cilium / response to food / erythrocyte homeostasis / ciliary membrane / WW domain binding / monoatomic ion channel activity / sodium ion transmembrane transport / cytoplasmic vesicle membrane / acrosomal vesicle / regulation of blood pressure / Stimuli-sensing channels / multicellular organism growth / gene expression / apical plasma membrane / response to xenobiotic stimulus / external side of plasma membrane / extracellular exosome / nucleoplasm / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||||||||
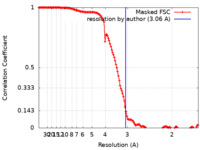
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.06 Å | |||||||||||||||
 データ登録者 データ登録者 | Posert R / Baconguis I / Noreng S / Bharadwaj A / Houser A | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Molecular principles of assembly, activation, and inhibition in epithelial sodium channel. 著者: Sigrid Noreng / Richard Posert / Arpita Bharadwaj / Alexandra Houser / Isabelle Baconguis /  要旨: The molecular bases of heteromeric assembly and link between Na self-inhibition and protease-sensitivity in epithelial sodium channels (ENaCs) are not fully understood. Previously, we demonstrated ...The molecular bases of heteromeric assembly and link between Na self-inhibition and protease-sensitivity in epithelial sodium channels (ENaCs) are not fully understood. Previously, we demonstrated that ENaC subunits - α, β, and γ - assemble in a counterclockwise configuration when viewed from outside the cell with the protease-sensitive GRIP domains in the periphery (Noreng et al., 2018). Here we describe the structure of ENaC resolved by cryo-electron microscopy at 3 Å. We find that a combination of precise domain arrangement and complementary hydrogen bonding network defines the subunit arrangement. Furthermore, we determined that the α subunit has a primary functional module consisting of the finger and GRIP domains. The module is bifurcated by the α2 helix dividing two distinct regulatory sites: Na and the inhibitory peptide. Removal of the inhibitory peptide perturbs the Na site via the α2 helix highlighting the critical role of the α2 helix in regulating ENaC function. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21896.map.gz emd_21896.map.gz | 204 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21896-v30.xml emd-21896-v30.xml emd-21896.xml emd-21896.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21896_fsc.xml emd_21896_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21896.png emd_21896.png | 88.7 KB | ||
| マスクデータ |  emd_21896_msk_1.map emd_21896_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21896.cif.gz emd-21896.cif.gz | 7.3 KB | ||
| その他 |  emd_21896_additional.map.gz emd_21896_additional.map.gz emd_21896_half_map_1.map.gz emd_21896_half_map_1.map.gz emd_21896_half_map_2.map.gz emd_21896_half_map_2.map.gz | 108.1 MB 200.4 MB 200.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21896 http://ftp.pdbj.org/pub/emdb/structures/EMD-21896 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21896 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21896 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21896_validation.pdf.gz emd_21896_validation.pdf.gz | 996 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21896_full_validation.pdf.gz emd_21896_full_validation.pdf.gz | 995.5 KB | 表示 | |
| XML形式データ |  emd_21896_validation.xml.gz emd_21896_validation.xml.gz | 21.8 KB | 表示 | |
| CIF形式データ |  emd_21896_validation.cif.gz emd_21896_validation.cif.gz | 28.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21896 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21896 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21896 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21896 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21896.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21896.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ENaC_FL sharpened EM map (Bfactor -101.4) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
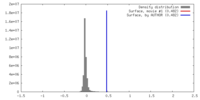
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21896_msk_1.map emd_21896_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
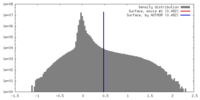
| 密度ヒストグラム |
-追加マップ: ENaC FL unsharpened EM map
| ファイル | emd_21896_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ENaC_FL unsharpened EM map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ENaC FL EM half map A
| ファイル | emd_21896_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ENaC_FL EM half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ENaC FL EM half map B
| ファイル | emd_21896_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ENaC_FL EM half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Full-length human ENaC heterotrimer bound with two high-affinity Fabs
| 全体 | 名称: Full-length human ENaC heterotrimer bound with two high-affinity Fabs |
|---|---|
| 要素 |
|
-超分子 #1: Full-length human ENaC heterotrimer bound with two high-affinity Fabs
| 超分子 | 名称: Full-length human ENaC heterotrimer bound with two high-affinity Fabs タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 320 KDa |
-分子 #1: Amiloride-sensitive sodium channel subunit alpha
| 分子 | 名称: Amiloride-sensitive sodium channel subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 75.780531 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEGNKLEEQD SSPPQSTPGL MKGNKREEQG LGPEPAAPQQ PTAEEEALIE FHRSYRELFE FFCNNTTIHG AIRLVCSQHN RMKTAFWAV LWLCTFGMMY WQFGLLFGEY FSYPVSLNIN LNSDKLVFPA VTICTLNPYR YPEIKEELEE LDRITEQTLF D LYKYSSFT ...文字列: MEGNKLEEQD SSPPQSTPGL MKGNKREEQG LGPEPAAPQQ PTAEEEALIE FHRSYRELFE FFCNNTTIHG AIRLVCSQHN RMKTAFWAV LWLCTFGMMY WQFGLLFGEY FSYPVSLNIN LNSDKLVFPA VTICTLNPYR YPEIKEELEE LDRITEQTLF D LYKYSSFT TLVAGSRSRR DLRGTLPHPL QRLRVPPPPH GARRARSVAS SLRDNNPQVD WKDWKIGFQL CNQNKSDCFY QT YSSGVDA VREWYRFHYI NILSRLPETL PSLEEDTLGN FIFACRFNQV SCNQANYSHF HHPMYGNCYT FNDKNNSNLW MSS MPGINN GLSLMLRAEQ NDFIPLLSTV TGARVMVHGQ DEPAFMDDGG FNLRPGVETS ISMRKETLDR LGGDYGDCTK NGSD VPVEN LYPSKYTQQV CIHSCFQESM IKECGCAYIF YPRPQNVEYC DYRKHSSWGY CYYKLQVDFS SDHLGCFTKC RKPCS VTSY QLSAGYSRWP SVTSQEWVFQ MLSRQNNYTV NNKRNGVAKV NIFFKELNYK TNSESPSVTM VTLLSNLGSQ WSLWFG SSV LSVVEMAELV FDLLVIMFLM LLRRFRSRYW SPGRGGRGAQ EVASTLASSP PSHFCPHPMS LSLSQPGPAP SPALTAP PP AYATLGPRPS PGGSAGASSS TCPLGGP UniProtKB: Epithelial sodium channel subunit alpha |
-分子 #2: Amiloride-sensitive sodium channel subunit beta
| 分子 | 名称: Amiloride-sensitive sodium channel subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 72.728891 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MHVKKYLLKG LHRLQKGPGY TYKELLVWYC DNTNTHGPKR IICEGPKKKA MWFLLTLLFA ALVCWQWGIF IRTYLSWEVS VSLSVGFKT MDFPAVTICN ASPFKYSKIK HLLKDLDELM EAVLERILAP ELSHANATRN LNFSIWNHTP LVLIDERNPH H PMVLDLFG ...文字列: MHVKKYLLKG LHRLQKGPGY TYKELLVWYC DNTNTHGPKR IICEGPKKKA MWFLLTLLFA ALVCWQWGIF IRTYLSWEVS VSLSVGFKT MDFPAVTICN ASPFKYSKIK HLLKDLDELM EAVLERILAP ELSHANATRN LNFSIWNHTP LVLIDERNPH H PMVLDLFG DNHNGLTSSS ASEKICNAHG CKMAMRLCSL NRTQCTFRNF TSATQALTEW YILQATNIFA QVPQQELVEM SY PGEQMIL ACLFGAEPCN YRNFTSIFYP HYGNCYIFNW GMTEKALPSA NPGTEFGLKL ILDIGQEDYV PFLASTAGVR LML HEQRSY PFIRDEGIYA MSGTETSIGV LVDKLQRMGE PYSPCTVNGS EVPVQNFYSD YNTTYSIQAC LRSCFQDHMI RNCN CGHYL YPLPRGEKYC NNRDFPDWAH CYSDLQMSVA QRETCIGMCK ESCNDTQYKM TISMADWPSE ASEDWIFHVL SQERD QSTN ITLSRKGIVK LNIYFQEFNY RTIEESAANN IVWLLSNLGG QFGFWMGGSV LCLIEFGEII IDFVWITIIK LVALAK SLR QRRAQASYAG PPPTVAELVE AHTNFGFQPD TAPRSPNTGP YPSEQALPIP GTPPPNYDSL RLQPLDVIES DSEGDAI UniProtKB: Epithelial sodium channel subunit beta |
-分子 #3: Amiloride-sensitive sodium channel subunit gamma
| 分子 | 名称: Amiloride-sensitive sodium channel subunit gamma / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 74.352984 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAPGEKIKAK IKKNLPVTGP QAPTIKELMR WYCLNTNTHG CRRIVVSRGR LRRLLWIGFT LTAVALILWQ CALLVFSFYT VSVSIKVHF RKLDFPAVTI CNINPYKYST VRHLLADLEQ ETREALKSLY GFPESRKRRE AESWNSVSEG KQPRFSHRIP L LIFDQDEK ...文字列: MAPGEKIKAK IKKNLPVTGP QAPTIKELMR WYCLNTNTHG CRRIVVSRGR LRRLLWIGFT LTAVALILWQ CALLVFSFYT VSVSIKVHF RKLDFPAVTI CNINPYKYST VRHLLADLEQ ETREALKSLY GFPESRKRRE AESWNSVSEG KQPRFSHRIP L LIFDQDEK GKARDFFTGR KRKVGGSIIH KASNVMHIES KQVVGFQLCS NDTSDCATYT FSSGINAIQE WYKLHYMNIM AQ VPLEKKI NMSYSAEELL VTCFFDGVSC DARNFTLFHH PMHGNCYTFN NRENETILST SMGGSEYGLQ VILYINEEEY NPF LVSSTG AKVIIHRQDE YPFVEDVGTE IETAMVTSIG MHLTESFKLS EPYSQCTEDG SDVPIRNIYN AAYSLQICLH SCFQ TKMVE KCGCAQYSQP LPPAANYCNY QQHPNWMYCY YQLHRAFVQE ELGCQSVCKE ACSFKEWTLT TSLAQWPSVV SEKWL LPVL TWDQGRQVNK KLNKTDLAKL LIFYKDLNQR SIMESPANSI EMLLSNFGGQ LGLWMSCSVV CVIEIIEVFF IDFFSI IAR RQWQKAKEWW AWKQAPPCPE APRSPQGQDN PALDIDDDLP TFNSALHLPP ALGTQVPGTP PPKYNTLRLE RAFSNQL TD TQMLDEL UniProtKB: Epithelial sodium channel subunit gamma |
-分子 #4: 7B1 Fab
| 分子 | 名称: 7B1 Fab / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 10.060393 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #5: 10D4 Fab
| 分子 | 名称: 10D4 Fab / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.805078 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK) |
-分子 #7: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 285 K / 装置: FEI VITROBOT MARK III 詳細: 3.5 uL applied, manual blot, fresh 3.5 uL applied, vitrobot blot and freeze. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)