+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21456 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer | ||||||||||||
 マップデータ マップデータ | M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Cryo-EM / broadly neutralizing antibody / bNAb / Fab / human immunodeficiency virus / HIV-1 / CH505 / SOSIP / Env / IMMUNE SYSTEM | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.86 Å | ||||||||||||
 データ登録者 データ登録者 | Chan K-W / Kong XP | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2020 ジャーナル: Cell Host Microbe / 年: 2020タイトル: VSV-Displayed HIV-1 Envelope Identifies Broadly Neutralizing Antibodies Class-Switched to IgG and IgA. 著者: Manxue Jia / Rachel A Liberatore / Yicheng Guo / Kun-Wei Chan / Ruimin Pan / Hong Lu / Eric Waltari / Eva Mittler / Kartik Chandran / Andrés Finzi / Daniel E Kaufmann / Michael S Seaman / ...著者: Manxue Jia / Rachel A Liberatore / Yicheng Guo / Kun-Wei Chan / Ruimin Pan / Hong Lu / Eric Waltari / Eva Mittler / Kartik Chandran / Andrés Finzi / Daniel E Kaufmann / Michael S Seaman / David D Ho / Lawrence Shapiro / Zizhang Sheng / Xiang-Peng Kong / Paul D Bieniasz / Xueling Wu /   要旨: The HIV-1 envelope (Env) undergoes conformational changes during infection. Broadly neutralizing antibodies (bNAbs) are typically isolated by using soluble Env trimers, which do not capture all Env ...The HIV-1 envelope (Env) undergoes conformational changes during infection. Broadly neutralizing antibodies (bNAbs) are typically isolated by using soluble Env trimers, which do not capture all Env states. To address these limitations, we devised a vesicular stomatitis virus (VSV)-based probe to display membrane-embedded Env trimers and isolated five bNAbs from two chronically infected donors, M4008 and M1214. Donor B cell receptor (BCR) repertoires identified two bNAb lineages, M4008_N1 and M1214_N1, that class-switched to immunoglobulin G (IgG) and IgA. Variants of these bNAbs reconstituted as IgA demonstrated broadly neutralizing activity, and the IgA fraction of M1214 plasma conferred neutralization. M4008_N1 epitope mapping revealed a glycan-independent V3 epitope conferring tier 2 virus neutralization. A 4.86-Å-resolution cryogenic electron microscopy (cryo-EM) structure of M1214_N1 complexed with CH505 SOSIP revealed another elongated epitope, the V2V5 corridor, extending from V2 to V5. Overall, the VSV probe identified bNAb lineages with neutralizing IgG and IgA members targeting distinct sites of HIV-1 Env vulnerability. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21456.map.gz emd_21456.map.gz | 167.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21456-v30.xml emd-21456-v30.xml emd-21456.xml emd-21456.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21456.png emd_21456.png | 38.7 KB | ||
| Filedesc metadata |  emd-21456.cif.gz emd-21456.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21456 http://ftp.pdbj.org/pub/emdb/structures/EMD-21456 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21456 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21456 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21456_validation.pdf.gz emd_21456_validation.pdf.gz | 446 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21456_full_validation.pdf.gz emd_21456_full_validation.pdf.gz | 445.6 KB | 表示 | |
| XML形式データ |  emd_21456_validation.xml.gz emd_21456_validation.xml.gz | 6.8 KB | 表示 | |
| CIF形式データ |  emd_21456_validation.cif.gz emd_21456_validation.cif.gz | 7.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21456 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21456 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21456 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21456 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21456.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21456.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
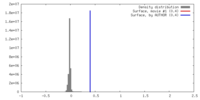
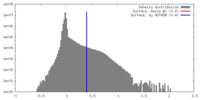
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer
| 全体 | 名称: M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer |
|---|---|
| 要素 |
|
-超分子 #1: M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer
| 超分子 | 名称: M1214_N1 Fab in complex with CH505 TF chimeric SOSIP.664 Env trimer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: chimeric CH505TF SOSIP664.R6 E64K A316W
| 超分子 | 名称: chimeric CH505TF SOSIP664.R6 E64K A316W / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: Fab fragment of the M1214_N1 IgG
| 超分子 | 名称: Fab fragment of the M1214_N1 IgG / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glycoprotein 120
| 分子 | 名称: Glycoprotein 120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.540402 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK EAKTTLFCAS DAKAYEKKVH NVWATHACVP TDPNPQEMVL KNVTENFNM WKNDMVDQMH EDVISLWDQS LKPCVKLTPL CVTLNCTNAT ASNSSIIEGM KNCSFNITTE LRDKREKKNA L FYKLDIVQ ...文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK EAKTTLFCAS DAKAYEKKVH NVWATHACVP TDPNPQEMVL KNVTENFNM WKNDMVDQMH EDVISLWDQS LKPCVKLTPL CVTLNCTNAT ASNSSIIEGM KNCSFNITTE LRDKREKKNA L FYKLDIVQ LDGNSSQYRL INCNTSVITQ ACPKVSFDPI PIHYCAPAGY AILKCNNKTF TGTGPCNNVS TVQCTHGIKP VV STQLLLN GSLAEGEIII RSENITNNVK TIIVHLNESV KIECTRPNNK TRTSIRIGPG QWFYATGQVI GDIREAYCNI NES KWNETL QRVSKKLKEY FPHKNITFQP SSGGDLEITT HSFNCGGEFF YCNTSSLFNR TYMANSTDMA NSTETNSTRT ITIH CRIKQ IINMWQEVGR AMYAPPIAGN ITCISNITGL LLTRDGGKNN TETFRPGGGN MKDNWRSELY KYKVVKIEPL GVAPT RCKR RVV UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Glycoprotein 41
| 分子 | 名称: Glycoprotein 41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 18.146699 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GRRRRRRAVG IGAVFLGFLG AAGSTMGAAS MTLTVQARNL LSGIVQQQSN LLRAPEAQQH LLKLTVWGIK QLQARVLAVE RYLRDQQLL GIWGCSGKLI CCTNVPWNSS WSNRNLSEIW DNMTWLQWDK EISNYTQIIY GLLEESQNQQ EKNEQDLLAL D UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: M1214 N1 Fab heavy chain
| 分子 | 名称: M1214 N1 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.210143 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DRLFQSGGGV SRPGGSLRVN CGASGFTVRT HYMYWLRQSP GKGLEWVAFM NSGGSVSYVD SVRGRFSVSR DNPANAMVLQ MDALKIEDT GTYYCARELR EAWYGDLRDY SGLDVWGRGT IVSISSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS ...文字列: DRLFQSGGGV SRPGGSLRVN CGASGFTVRT HYMYWLRQSP GKGLEWVAFM NSGGSVSYVD SVRGRFSVSR DNPANAMVLQ MDALKIEDT GTYYCARELR EAWYGDLRDY SGLDVWGRGT IVSISSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPK |
-分子 #4: M1214 N1 Fab light chain
| 分子 | 名称: M1214 N1 Fab light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.87642 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSALAQPPSV SGSPGQSVTI TCTGINDYGA AYKFVSWYQQ HPGKEPRLIM KNVKDRWSVT PNRFSGSTSG NTASLTISNL QSDDEAQYF CAVYAGGFTF PRLGGGTKLS VLSQPKAAPS VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP V KAGVETTT ...文字列: QSALAQPPSV SGSPGQSVTI TCTGINDYGA AYKFVSWYQQ HPGKEPRLIM KNVKDRWSVT PNRFSGSTSG NTASLTISNL QSDDEAQYF CAVYAGGFTF PRLGGGTKLS VLSQPKAAPS VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP V KAGVETTT PSKQSNNKYA ASSYLSLTPE QWKSHRSYSC QVTHEGSTVE KTVAPT |
-分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 11 / コピー数: 24 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)