+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21382 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM map of an MTA-HDAC-MBD complex | |||||||||
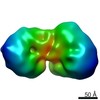
 マップデータ マップデータ | Negative stain EM map of an MTA-HDAC-MBD complex. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
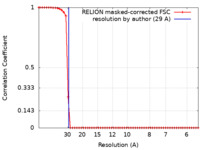
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 29.0 Å | |||||||||
 データ登録者 データ登録者 | Silva APG / Low JKK / Tabar MS / Torrado M / Schmidberger JW / Brillault L / Landsberg MJ / Mackay JP | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2020 ジャーナル: Cell Rep / 年: 2020タイトル: The Nucleosome Remodeling and Deacetylase Complex Has an Asymmetric, Dynamic, and Modular Architecture. 著者: Jason K K Low / Ana P G Silva / Mehdi Sharifi Tabar / Mario Torrado / Sarah R Webb / Benjamin L Parker / Maryam Sana / Callum Smits / Jason W Schmidberger / Lou Brillault / Matthew J Jackman ...著者: Jason K K Low / Ana P G Silva / Mehdi Sharifi Tabar / Mario Torrado / Sarah R Webb / Benjamin L Parker / Maryam Sana / Callum Smits / Jason W Schmidberger / Lou Brillault / Matthew J Jackman / David C Williams / Gerd A Blobel / Sandra B Hake / Nicholas E Shepherd / Michael J Landsberg / Joel P Mackay /    要旨: The nucleosome remodeling and deacetylase (NuRD) complex is essential for metazoan development but has been refractory to biochemical analysis. We present an integrated analysis of the native ...The nucleosome remodeling and deacetylase (NuRD) complex is essential for metazoan development but has been refractory to biochemical analysis. We present an integrated analysis of the native mammalian NuRD complex, combining quantitative mass spectrometry, cross-linking, protein biochemistry, and electron microscopy to define the architecture of the complex. NuRD is built from a 2:2:4 (MTA, HDAC, and RBBP) deacetylase module and a 1:1:1 (MBD, GATAD2, and Chromodomain-Helicase-DNA-binding [CHD]) remodeling module, and the complex displays considerable structural dynamics. The enigmatic GATAD2 controls the asymmetry of the complex and directly recruits the CHD remodeler. The MTA-MBD interaction acts as a point of functional switching, with the transcriptional regulator PWWP2A competing with MBD for binding to the MTA-HDAC-RBBP subcomplex. Overall, our data address the long-running controversy over NuRD stoichiometry, provide imaging of the mammalian NuRD complex, and establish the biochemical mechanism by which PWWP2A can regulate NuRD composition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21382.map.gz emd_21382.map.gz | 478.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21382-v30.xml emd-21382-v30.xml emd-21382.xml emd-21382.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21382_fsc.xml emd_21382_fsc.xml | 4.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21382.png emd_21382.png | 20.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21382 http://ftp.pdbj.org/pub/emdb/structures/EMD-21382 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21382 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21382 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21382_validation.pdf.gz emd_21382_validation.pdf.gz | 297.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21382_full_validation.pdf.gz emd_21382_full_validation.pdf.gz | 297.4 KB | 表示 | |
| XML形式データ |  emd_21382_validation.xml.gz emd_21382_validation.xml.gz | 8.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21382 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21382 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21382 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21382 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10539 (タイトル: Negative-stained transmission electron microscopy of the MTA-HDAC-MBD core complex purified from EXPI cells EMPIAR-10539 (タイトル: Negative-stained transmission electron microscopy of the MTA-HDAC-MBD core complex purified from EXPI cellsData size: 20.3 Data #1: micrograph of the MTA-HDAC-MBDcc core compelx of NuRD [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21382.map.gz / 形式: CCP4 / 大きさ: 12.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21382.map.gz / 形式: CCP4 / 大きさ: 12.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain EM map of an MTA-HDAC-MBD complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.79 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : MHm complex
| 全体 | 名称: MHm complex |
|---|---|
| 要素 |
|
-超分子 #1: MHm complex
| 超分子 | 名称: MHm complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Complex formed by HA-MTA2(1-429), HA-HDAC1 and FLAG-MBD3cc that were recombinantly expressed in HEK293 cells. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
| 分子量 | 実験値: 300 KDa |
-超分子 #2: Metastasis-associated protein MTA2,
| 超分子 | 名称: Metastasis-associated protein MTA2, / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1, #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-超分子 #3: Histone deacetylase 1
| 超分子 | 名称: Histone deacetylase 1 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Metastasis-associated protein MTA2
| 分子 | 名称: Metastasis-associated protein MTA2 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAANMYRVGD YVYFENSSSN PYLVRRIEEL NKTANGNVEA KVVCLFRRRD ISSSLNSLAD SNAREFEEE SKQPGVSEQQ RHQLKHRELF LSRQFESLPA THIRGKCSVT LLNETDILNQ Y LDKEDCFF YSLVFDPVQK TLLADQGEIR VGCKFQAEIP DRLAEGESDN ...文字列: MAANMYRVGD YVYFENSSSN PYLVRRIEEL NKTANGNVEA KVVCLFRRRD ISSSLNSLAD SNAREFEEE SKQPGVSEQQ RHQLKHRELF LSRQFESLPA THIRGKCSVT LLNETDILNQ Y LDKEDCFF YSLVFDPVQK TLLADQGEIR VGCKFQAEIP DRLAEGESDN RNQQKMEMKV WD PDNPLTD RQIDQFLVVA RAVGTFARAL DCSSSIRQPS LHMSAAAASR DITLFHAMDT LQR NGYDLA KAMSTLVPQG GPVLCRDEME EWSASEAMLF EEALEKYGKD FNDIRQDFLP WKSL ASIVQ FYYMWKTTDR YIQQKRLKAA EADSKLKQVY IPTYTKPNPN QIISVGSKPG MNGAG FQKG LTCESCHTTQ SAQWYAWGPP NMQCRLCASC WIYWKKYGGL KTPTQLEGAA RGTTEP HSR GHLSRP |
-分子 #2: Histone deacetylase 1
| 分子 | 名称: Histone deacetylase 1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAQTQGTRRK VCYYYDGDVG NYYYGQGHPM KPHRIRMTHN LLLNYGLYRK MEIYRPHKAN AEEMTKYHS DDYIKFLRSI RPDNMSEYSK QMQRFNVGED CPVFDGLFEF CQLSTGGSVA S AVKLNKQQ TDIAVNWAGG LHHAKKSEAS GFCYVNDIVL AILELLKYHQ ...文字列: MAQTQGTRRK VCYYYDGDVG NYYYGQGHPM KPHRIRMTHN LLLNYGLYRK MEIYRPHKAN AEEMTKYHS DDYIKFLRSI RPDNMSEYSK QMQRFNVGED CPVFDGLFEF CQLSTGGSVA S AVKLNKQQ TDIAVNWAGG LHHAKKSEAS GFCYVNDIVL AILELLKYHQ RVLYIDIDIH HG DGVEEAF YTTDRVMTVS FHKYGEYFPG TGDLRDIGAG KGKYYAVNYP LRDGIDDESY EAI FKPVMS KVMEMFQPSA VVLQCGSDSL SGDRLGCFNL TIKGHAKCVE FVKSFNLPML MLGG GGYTI RNVARCWTYE TAVALDTEIP NELPYNDYFE YFGPDFKLHI SPSNMTNQNT NEYLE KIKQ RLFENLRMLP HAPGVQMQAI PEDAIPEESG DEDEDDPDKR ISICSSDKRI ACEEEF SDS EEEGEGGRKN SSNFKKAKRV KTEDEKEKDP EEKKEVTEEE KTKEEKPEAK GVKEEVK LA |
-分子 #3: Methyl-CpG-binding protein MBD3/GATA zinc finger domain-containin...
| 分子 | 名称: Methyl-CpG-binding protein MBD3/GATA zinc finger domain-containing protein 2A fusion タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MERKRWECPA LPQGWEREEV PRRSGLSAGH RDVFYYSPSG KKFRSKPQLA RYLGGSMDLS TFDFRTGKM LMNKMNKSRQ RVRYDSSNQV KGKPDLNTAL PVRQTASIFK QPVTKITNHP S NKVKSDPQ KAVDQPRQLF WEKKLSGLSA FDIAEELVRT MDLPKGLQGV ...文字列: MERKRWECPA LPQGWEREEV PRRSGLSAGH RDVFYYSPSG KKFRSKPQLA RYLGGSMDLS TFDFRTGKM LMNKMNKSRQ RVRYDSSNQV KGKPDLNTAL PVRQTASIFK QPVTKITNHP S NKVKSDPQ KAVDQPRQLF WEKKLSGLSA FDIAEELVRT MDLPKGLQGV GPGCTDETLL SA IASALHT STLPITGQLS AAVEKNPGVW LNTAQPLCKA FMVTDDDIRK QEELVQQVRK RLE EALMAD MLAHVEELAR DGEAPLDKAC AEEEEEEEEE EEEPEPERVS PEERERMIKQ LKEELRLEEA KLVLLKKLRQ SQIQKEATAQ K |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.027 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate 詳細: Sample (5 microliters) was applied to a glow-discharged, carbon-coated 400-mesh copper grid (GSCu400CC, ProSciTech). After 2 minutes incubation time, the grid was blotted and washed with ten ...詳細: Sample (5 microliters) was applied to a glow-discharged, carbon-coated 400-mesh copper grid (GSCu400CC, ProSciTech). After 2 minutes incubation time, the grid was blotted and washed with ten drops of distilled water, blotting on a filter paper in between washes. The grid was then washed in a drop of 1 % (w/v) uranyl acetate, blotted and subsequently incubated in another drop of 1% (w/v) uranyl acetate solution for 30 seconds, blotted and allowed to air dry at room temperature. | ||||||||||||
| グリッド | 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 詳細 | Sample was a sample from GraFix purification. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 撮影 | フィルム・検出器のモデル: OTHER / 撮影したグリッド数: 1 / 実像数: 325 / 平均電子線量: 10.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)