+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21136 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mouse retromer (VPS26/VPS35/VPS29) heterotrimer | |||||||||
 マップデータ マップデータ | Mouse retromer (VPS26/VPS35/VPS29) heterotrimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | retromer / membrane trafficking / endosomal trafficking / membrane coat complexes / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報WNT ligand biogenesis and trafficking / neurotransmitter receptor transport, endosome to plasma membrane / negative regulation of protein localization / regulation of dendritic spine maintenance / mitochondrion-derived vesicle / negative regulation of protein homooligomerization / tubular endosome / regulation of terminal button organization / positive regulation of Wnt protein secretion / mitochondrion to lysosome vesicle-mediated transport ...WNT ligand biogenesis and trafficking / neurotransmitter receptor transport, endosome to plasma membrane / negative regulation of protein localization / regulation of dendritic spine maintenance / mitochondrion-derived vesicle / negative regulation of protein homooligomerization / tubular endosome / regulation of terminal button organization / positive regulation of Wnt protein secretion / mitochondrion to lysosome vesicle-mediated transport / vacuolar protein processing / retromer, cargo-selective complex / Golgi to vacuole transport / protein localization to organelle / positive regulation of locomotion involved in locomotory behavior / negative regulation of lysosomal protein catabolic process / negative regulation of late endosome to lysosome transport / vesicle-mediated transport in synapse / positive regulation of dopamine biosynthetic process / mitochondrial fragmentation involved in apoptotic process / positive regulation of dopamine receptor signaling pathway / neurotransmitter receptor transport, endosome to postsynaptic membrane / protein localization to endosome / retromer complex / voluntary musculoskeletal movement / dopaminergic synapse / transcytosis / regulation of synapse maturation / endocytic recycling / retrograde transport, endosome to Golgi / positive regulation of protein localization to cell periphery / lysosome organization / positive regulation of mitochondrial fission / regulation of postsynapse assembly / D1 dopamine receptor binding / intracellular protein transport / modulation of chemical synaptic transmission / protein destabilization / negative regulation of inflammatory response / positive regulation of protein catabolic process / positive regulation of canonical Wnt signaling pathway / late endosome / protein transport / presynapse / vesicle / early endosome / lysosome / postsynapse / neuron projection / postsynaptic density / endosome / endosome membrane / negative regulation of gene expression / neuronal cell body / synapse / positive regulation of gene expression / perinuclear region of cytoplasm / glutamatergic synapse / mitochondrion / metal ion binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
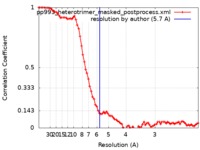
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.7 Å | |||||||||
 データ登録者 データ登録者 | Kendall AK / Jackson LP | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2020 ジャーナル: Structure / 年: 2020タイトル: Mammalian Retromer Is an Adaptable Scaffold for Cargo Sorting from Endosomes. 著者: Amy K Kendall / Boyang Xie / Peng Xu / Jue Wang / Rodger Burcham / Meredith N Frazier / Elad Binshtein / Hui Wei / Todd R Graham / Terunaga Nakagawa / Lauren P Jackson /  要旨: Metazoan retromer (VPS26/VPS35/VPS29) associates with sorting nexins on endosomal tubules to sort proteins to the trans-Golgi network or plasma membrane. Mechanisms of metazoan retromer assembly ...Metazoan retromer (VPS26/VPS35/VPS29) associates with sorting nexins on endosomal tubules to sort proteins to the trans-Golgi network or plasma membrane. Mechanisms of metazoan retromer assembly remain undefined. We combine single-particle cryoelectron microscopy with biophysical methods to uncover multiple oligomer structures. 2D class averages reveal mammalian heterotrimers; dimers of trimers; tetramers of trimers; and flat chains. These species are further supported by biophysical solution studies. We provide reconstructions of all species, including key sub-structures (∼5 Å resolution). Local resolution variation suggests that heterotrimers and dimers adopt multiple conformations. Our structures identify a flexible, highly conserved electrostatic dimeric interface formed by VPS35 subunits. We generate structure-based mutants to disrupt this interface in vitro. Equivalent mutations in yeast demonstrate a mild cargo-sorting defect. Our data suggest the metazoan retromer is an adaptable and plastic scaffold that accommodates interactions with different sorting nexins to sort multiple cargoes from endosomes their final destinations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21136.map.gz emd_21136.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21136-v30.xml emd-21136-v30.xml emd-21136.xml emd-21136.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21136_fsc.xml emd_21136_fsc.xml | 8.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21136.png emd_21136.png | 140.2 KB | ||
| Filedesc metadata |  emd-21136.cif.gz emd-21136.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21136 http://ftp.pdbj.org/pub/emdb/structures/EMD-21136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21136 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21136.map.gz / 形式: CCP4 / 大きさ: 46.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21136.map.gz / 形式: CCP4 / 大きさ: 46.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Mouse retromer (VPS26/VPS35/VPS29) heterotrimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.096 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mouse retromer (VPS26/VPS35/VPS29) tetramer of heterotrimers
| 全体 | 名称: Mouse retromer (VPS26/VPS35/VPS29) tetramer of heterotrimers |
|---|---|
| 要素 |
|
-超分子 #1: Mouse retromer (VPS26/VPS35/VPS29) tetramer of heterotrimers
| 超分子 | 名称: Mouse retromer (VPS26/VPS35/VPS29) tetramer of heterotrimers タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Vacuolar protein sorting-associated protein 35
| 分子 | 名称: Vacuolar protein sorting-associated protein 35 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 91.821727 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPTTQQSPQD EQEKLLDEAI QAVKVQSFQM KRCLDKNKLM DALKHASNML GELRTSMLSP KSYYELYMAI SDELHYLEVY LTDEFAKGR KVADLYELVQ YAGNIIPRLY LLITVGVVYV KSFPQSRKDI LKDLVEMCRG VQHPLRGLFL RNYLLQCTRN I LPDEGEPT ...文字列: MPTTQQSPQD EQEKLLDEAI QAVKVQSFQM KRCLDKNKLM DALKHASNML GELRTSMLSP KSYYELYMAI SDELHYLEVY LTDEFAKGR KVADLYELVQ YAGNIIPRLY LLITVGVVYV KSFPQSRKDI LKDLVEMCRG VQHPLRGLFL RNYLLQCTRN I LPDEGEPT DEETTGDISD SMDFVLLNFA EMNKLWVRMQ HQGHSRDREK RERERQELRI LVGTNLVRLS QLEGVNVERY KQ IVLTGIL EQVVNCRDAL AQEYLMECII QVFPDEFHLQ TLNPFLRACA ELHQNVNVKN IIIALIDRLA LFAHREDGPG IPA EIKLFD IFSQQVATVI QSRQDMPSED VVSLQVSLIN LAMKCYPDRV DYVDKVLETT VEIFNKLNLE HIATSSAVSK ELTR LLKIP VDTYNNILTV LKLKHFHPLF EYFDYESRKS MSCYVLSNVL DYNTEIVSQD QVDSIMNLVS TLIQDQPDQP VEDPD PEDF ADEQSLVGRF IHLLRSDDPD QQYLILNTAR KHFGAGGNQR IRFTLPPLVF AAYQLAFRYK ENSQMDDKWE KKCQKI FSF AHQTISALIK AELAELPLRL FLQGALAAGE IGFENHETVA YEFMSQAFSL YEDEISDSKA QLAAITLIIG TFERMKC FS EENHEPLRTQ CALAASKLLK KPDQGRAVST CAHLFWSGRN TDKNGEELHG GKRVMECLKK ALKIANQCMD PSLQVQLF I EILNRYIYFY EKENDAVTIQ VLNQLIQKIR EDLPNLESSE ETEQINKHFH NTLEHLRSRR ESPESEGPIY EGLIL UniProtKB: Vacuolar protein sorting-associated protein 35 |
-分子 #2: Vacuolar protein sorting-associated protein 26A
| 分子 | 名称: Vacuolar protein sorting-associated protein 26A / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 38.167789 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSFLGGFFGP ICEIDVALND GETRKMAEMK TEDGKVEKHY LFYDGESVSG KVNLAFKQPG KRLEHQGIRI EFVGQIELFN DKSNTHEFV NLVKELALPG ELTQSRSYDF EFMQVEKPYE SYIGANVRLR YFLKVTIVRR LTDLVKEYDL IVHQLATYPD V NNSIKMEV ...文字列: MSFLGGFFGP ICEIDVALND GETRKMAEMK TEDGKVEKHY LFYDGESVSG KVNLAFKQPG KRLEHQGIRI EFVGQIELFN DKSNTHEFV NLVKELALPG ELTQSRSYDF EFMQVEKPYE SYIGANVRLR YFLKVTIVRR LTDLVKEYDL IVHQLATYPD V NNSIKMEV GIEDCLHIEF EYNKSKYHLK DVIVGKIYFL LVRIKIQHME LQLIKKEITG IGPSTTTETE TIAKYEIMDG AP VKGESIP IRLFLAGYDP TPTMRDVNKK FSVRYFLNLV LVDEEDRRYF KQQEIILWRK APEKLRKQRT NFHQRFESPD SQA SAEQPE M UniProtKB: Vacuolar protein sorting-associated protein 26A |
-分子 #3: Vacuolar protein sorting-associated protein 29
| 分子 | 名称: Vacuolar protein sorting-associated protein 29 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 20.521668 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLVLVLGDLH IPHRCNSLPA KFKKLLVPGK IQHILCTGNL CTKESYDYLK TLAGDVHIVR GDFDENLNYP EQKVVTVGQF KIGLIHGHQ VIPWGDMASL ALLQRQFDVD ILISGHTHKF EAFEHENKFY INPGSATGAY NALETNIIPS FVLMDIQAST V VTYVYQLI GDDVKVERIE YKKS UniProtKB: Vacuolar protein sorting-associated protein 29 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 8.2 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 71.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)