+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20850 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
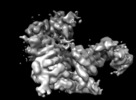
| タイトル | Focused map of type 3 IP3 receptor N-terminal domain revealing the presence of a self-binding peptide | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Azumaya CM / Linton EA / Risener CJ / Nakagawa T / Karakas E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2020 ジャーナル: J Biol Chem / 年: 2020タイトル: Cryo-EM structure of human type-3 inositol triphosphate receptor reveals the presence of a self-binding peptide that acts as an antagonist. 著者: Caleigh M Azumaya / Emily A Linton / Caitlin J Risener / Terunaga Nakagawa / Erkan Karakas /  要旨: Calcium-mediated signaling through inositol 1,4,5-triphosphate receptors (IPRs) is essential for the regulation of numerous physiological processes, including fertilization, muscle contraction, ...Calcium-mediated signaling through inositol 1,4,5-triphosphate receptors (IPRs) is essential for the regulation of numerous physiological processes, including fertilization, muscle contraction, apoptosis, secretion, and synaptic plasticity. Deregulation of IPRs leads to pathological calcium signaling and is implicated in many common diseases, including cancer and neurodegenerative, autoimmune, and metabolic diseases. Revealing the mechanism of activation and inhibition of this ion channel will be critical to an improved understanding of the biological processes that are controlled by IPRs. Here, we report structural findings of the human type-3 IPR (IPR-3) obtained by cryo-EM (at an overall resolution of 3.8 Å), revealing an unanticipated regulatory mechanism where a loop distantly located in the primary sequence occupies the IP-binding site and competitively inhibits IP binding. We propose that this inhibitory mechanism must differ qualitatively among IPR subtypes because of their diverse loop sequences, potentially serving as a key molecular determinant of subtype-specific calcium signaling in IPRs. In summary, our structural characterization of human IPR-3 provides critical insights into the mechanistic function of IPRs and into subtype-specific regulation of these important calcium-regulatory channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20850.map.gz emd_20850.map.gz | 138.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20850-v30.xml emd-20850-v30.xml emd-20850.xml emd-20850.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20850.png emd_20850.png | 83.9 KB | ||
| その他 |  emd_20850_additional.map.gz emd_20850_additional.map.gz emd_20850_additional_1.map.gz emd_20850_additional_1.map.gz emd_20850_half_map_1.map.gz emd_20850_half_map_1.map.gz emd_20850_half_map_2.map.gz emd_20850_half_map_2.map.gz | 138.8 MB 138.8 MB 15.3 MB 15.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20850 http://ftp.pdbj.org/pub/emdb/structures/EMD-20850 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20850 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20850 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20850_validation.pdf.gz emd_20850_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20850_full_validation.pdf.gz emd_20850_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_20850_validation.xml.gz emd_20850_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20850 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20850 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20850 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20850 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20850.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20850.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.247 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpened map
| ファイル | emd_20850_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
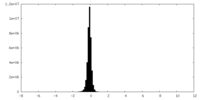
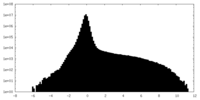
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_20850_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
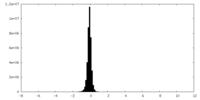
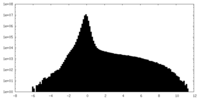
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_20850_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_20850_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : inositol 1,4,5-triphosphate receptor, type 3
| 全体 | 名称: inositol 1,4,5-triphosphate receptor, type 3 |
|---|---|
| 要素 |
|
-超分子 #1: inositol 1,4,5-triphosphate receptor, type 3
| 超分子 | 名称: inositol 1,4,5-triphosphate receptor, type 3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  組換株: Sf9 |
| 分子量 | 理論値: 1.2 MDa |
-分子 #1: inositol 1,4,5-triphosphate receptor, type 3
| 分子 | 名称: inositol 1,4,5-triphosphate receptor, type 3 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSFLHIGDI VSLYAEGSVN GFISTLGLVD DRCVVEPAAG DLDNPPKKFR DCLFKVCPMN RYSAQKQYWK AKQTKQDKEK IADVVLLQKL QHAAQMEQKQ NDTENKKVHG DVVKYGSVIQ LLHMKSNKYL TVNKRLPALL EKNAMRVTLD ATGNEGSWLF IQPFWKLRSN ...文字列: MSSFLHIGDI VSLYAEGSVN GFISTLGLVD DRCVVEPAAG DLDNPPKKFR DCLFKVCPMN RYSAQKQYWK AKQTKQDKEK IADVVLLQKL QHAAQMEQKQ NDTENKKVHG DVVKYGSVIQ LLHMKSNKYL TVNKRLPALL EKNAMRVTLD ATGNEGSWLF IQPFWKLRSN GDNVVVGDKV ILNPVNAGQP LHASNYELSD NAGCKEVNSV NCNTSWKINL FMQFRDHLEE VLKGGDVVRL FHAEQEKFLT CDEYKGKLQV FLRTTLRQSA TSATSSNALW EVEVVHHDPC RGGAGHWNGL YRFKHLATGN YLAAEENPSY KGDASDPKAA GMGAQGRTGR RNAGEKIKYC LVAVPHGNDI ASLFELDPTT LQKTDSFVPR NSYVRLRHLC TNTWIQSTNV PIDIEEERPI RLMLGTCPTK EDKEAFAIVS VPVSEIRDLD FANDASSMLA SAVEKLNEGF ISQNDRRFVI QLLEDLVFFV SDVPNNGQNV LDIMVTKPNR ERQKLMREQN ILKQVFGILK APFREKGGEG PLVRLEELSD QKNAPYQHMF RLCYRVLRHS QEDYRKNQEH IAKQFGMMQS QIGYDILAED TITALLHNNR KLLEKHITKT EVETFVSLVR KNREPRFLDY LSDLCVSNHI AIPVTQELIC KCVLDPKNSD ILIRTELRPV KEMAQSHEYL SIEYSEEEVW LTWTDKNNEH HEKSVRQLAQ EARAGNAHDE NVLSYYRYQL KLFARMCLDR QYLAIDEISQ QLGVDLIFLC MADEMLPFDL RASFCHLMLH VHVDRDPQEL VTPVKFARLW TEIPTAITIK DYDSNLNASR DDKKNKFANT MEFVEDYLNN VVSEAVPFAN EEKNKLTFEV VSLAHNLIYF GFYSFSELLR LTRTLLGIID CVQGPPAMLQ AYEDPGGKNV RRSIQGVGHM MSTMVLSRKQ SVFSAPSLSA GASAAEPLDR SKFEENEDIV VMETKLKILE ILQFILNVRL DYRISYLLSV FKKEFVEVFP MQDSGADGTA PAFDSTTANM NLDRIGEQAE AMFGVGKTSS MLEVDDEGGR MFLRVLIHLT MHDYAPLVSG ALQLLFKHFS QRQEAMHTFK QVQLLISAQD VENYKVIKSE LDRLRTMVEK SELWVDKKGS GKGEEVEAGA AKDKKERPTD EEGFLHPPGE KSSENYQIVK GILERLNKMC GVGEQMRKKQ QRLLKNMDAH KVMLDLLQIP YDKGDAKMME ILRYTHQFLQ KFCAGNPGNQ ALLHKHLHLF LTPGLLEAET MQHIFLNNYQ LCSEISEPVL QHFVHLLATH GRHVQYLDFL HTVIKAEGKY VKKCQDMIMT ELTNAGDDVV VFYNDKASLA HLLDMMKAAR DGVEDHSPLM YHISLVDLLA ACAEGKNVYT EIKCTSLLPL EDVVSVVTHE DCITEVKMAY VNFVNHCYVD TEVEMKEIYT SNHIWTLFEN FTLDMARVCS KREKRVADPT LEKYVLSVVL DTINAFFSSP FSENSTSLQT HQTIVVQLLQ STTRLLECPW LQQQHKGSVE ACIRTLAMVA KGRAILLPMD LDAHISSMLS SGASCAAAAQ RNASSYKATT RAFPRVTPTA NQWDYKNIIE KLQDIITALE ERLKPLVQAE LSVLVDVLHW PELLFLEGSE AYQRCESGGF LSKLIQHTKD LMESEEKLCI KVLRTLQQML LKKTKYGDRG NQLRKMLLQN YLQNRKSTSR GDLPDPIGTG LDPDWSAIAA TQCRLDKEGA TKLVCDLITS TKNEKIFQES IGLAIHLLDG GNTEIQKSFH NLMMSDKKSE RFFKVLHDRM KRAQQETKST VAVNMNDLGS QPHEDREPVD PTTKGRVASF SIPGSSSRYS LGPSLRRGHE VSERVQSSEM GTSVLIMQPI LRFLQLLCEN HNRDLQNFLR CQNNKTNYNL VCETLQFLDI MCGSTTGGLG LLGLYINEDN VGLVIQTLET LTEYCQGPCH ENQTCIVTHE SNGIDIITAL ILNDISPLCK YRMDLVLQLK DNASKLLLAL MESRHDSENA ERILISLRPQ ELVDVIKKAY LQEEERENSE VSPREVGHNI YILALQLSRH NKQLQHLLKP VKRIQEEEAE GISSMLSLNN KQLSQMLKSS APAQEEEEDP LAYYENHTSQ IEIVRQDRSM EQIVFPVPGI CQFLTEETKH RLFTTTEQDE QGSKVSDFFD QSSFLHNEME WQRKLRSMPL IYWFSRRMTL WGSISFNLAV FINIIIAFFY PYMEGASTGV LDSPLISLLF WILICFSIAA LFTKRYSIRP LIVALILRSI YYLGIGPTLN ILGALNLTNK IVFVVSFVGN RGTFIRGYKA MVMDMEFLYH VGYILTSVLG LFAHELFYSI LLFDLIYREE TLFNVIKSVT RNGRSILLTA LLALILVYLF SIVGFLFLKD DFILEVDRLP NNHSTASPLG MPHGAAAFVD TCSGDKMDCV SGLSVPEVLE EDRELDSTER ACDTLLMCIV TVMNHGLRNG GGVGDILRKP SKDESLFPAR VVYDLLFFFI VIIIVLNLIF GVIIDTFADL RSEKQKKEEI LKTTCFICGL ERDKFDNKTV SFEEHIKLEH NMWNYLYFIV LVRVKNKTDY TGPESYVAQM IKNKNLDWFP RMRAMSLVSN EGEGEQNEIR ILQDKLNSTM KLVSHLTAQL NELKEQMTEQ RKRRQRLGFV DVQNCISRGE NLYFQSAWSH PQFEKGGGSG GGSGGSAWSH PQFEK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV / 詳細: The grid was blotted for 3 seconds at force 1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 倍率(公称値): 31000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)