+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2048 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the UPF-EJC complex | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of the nonsense-mediated mRNA decay (NMD) complex containing proteins UPF1, UPF2, UPF3 and the Exon Junction Complex (EJC). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NMD / EJC / UPF1 / UPF2 / UPF3 / RNA degradation / GraFix | |||||||||
| 機能・相同性 | WIBG, Mago-binding / UPF3 domain / RNA helicase UPF1, Cys/His rich zinc-binding domain / Btz domain / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Melero R / Buchwald G / Castano R / Raabe M / Gil D / Lazaro M / Urlaub H / Conti E / Llorca O | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2012 ジャーナル: Nat Struct Mol Biol / 年: 2012タイトル: The cryo-EM structure of the UPF-EJC complex shows UPF1 poised toward the RNA 3' end. 著者: Roberto Melero / Gretel Buchwald / Raquel Castaño / Monika Raabe / David Gil / Melisa Lázaro / Henning Urlaub / Elena Conti / Oscar Llorca /  要旨: Nonsense-mediated mRNA decay (NMD) is a eukaryotic surveillance pathway that degrades aberrant mRNAs containing premature termination codons (PTCs). NMD is triggered upon the assembly of the UPF ...Nonsense-mediated mRNA decay (NMD) is a eukaryotic surveillance pathway that degrades aberrant mRNAs containing premature termination codons (PTCs). NMD is triggered upon the assembly of the UPF surveillance complex near a PTC. In humans, UPF assembly is prompted by the exon junction complex (EJC). We investigated the molecular architecture of the human UPF complex bound to the EJC by cryo-EM and using positional restraints from additional EM, MS and biochemical interaction data. The heptameric assembly is built around UPF2, a scaffold protein with a ring structure that closes around the CH domain of UPF1, keeping the helicase region in an accessible and unwinding-competent state. UPF2 also positions UPF3 to interact with the EJC. The geometry is such that this transient complex poises UPF1 to elicit helicase activity toward the 3' end of the mRNP. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2048.map.gz emd_2048.map.gz | 6.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2048-v30.xml emd-2048-v30.xml emd-2048.xml emd-2048.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2048.tif EMD-2048.tif | 1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2048 http://ftp.pdbj.org/pub/emdb/structures/EMD-2048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2048 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2048_validation.pdf.gz emd_2048_validation.pdf.gz | 196.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2048_full_validation.pdf.gz emd_2048_full_validation.pdf.gz | 196.1 KB | 表示 | |
| XML形式データ |  emd_2048_validation.xml.gz emd_2048_validation.xml.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2048 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2048 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2048 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2048 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2048.map.gz / 形式: CCP4 / 大きさ: 9.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2048.map.gz / 形式: CCP4 / 大きさ: 9.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the nonsense-mediated mRNA decay (NMD) complex containing proteins UPF1, UPF2, UPF3 and the Exon Junction Complex (EJC). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
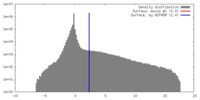
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex containing the NMD proteins UPF1, UPF2 and UPF3 bound to ...
| 全体 | 名称: Complex containing the NMD proteins UPF1, UPF2 and UPF3 bound to the exon junction complex (EJC) |
|---|---|
| 要素 |
|
-超分子 #1000: Complex containing the NMD proteins UPF1, UPF2 and UPF3 bound to ...
| 超分子 | 名称: Complex containing the NMD proteins UPF1, UPF2 and UPF3 bound to the exon junction complex (EJC) タイプ: sample / ID: 1000 / 集合状態: Hetero-octameric / Number unique components: 8 |
|---|---|
| 分子量 | 実験値: 390 KDa / 理論値: 390 KDa |
-分子 #1: Up-frameshift protein 1
| 分子 | 名称: Up-frameshift protein 1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: UPF1 / 詳細: 115-914 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 90 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: RNA helicase UPF1, Cys/His rich zinc-binding domain |
-分子 #2: Up-frameshift protein 2
| 分子 | 名称: Up-frameshift protein 2 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: UPF2 / 詳細: full length / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 148 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: RNA helicase UPF1, Cys/His rich zinc-binding domain |
-分子 #3: Up-frameshift protein 3b
| 分子 | 名称: Up-frameshift protein 3b / タイプ: protein_or_peptide / ID: 3 / Name.synonym: UPF3b / 詳細: full length / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 56 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: UPF3 domain |
-分子 #4: MAGO
| 分子 | 名称: MAGO / タイプ: protein_or_peptide / ID: 4 / Name.synonym: MAGO / 詳細: full length / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 16 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: WIBG, Mago-binding |
-分子 #5: Y14
| 分子 | 名称: Y14 / タイプ: protein_or_peptide / ID: 5 / Name.synonym: Y14 / 詳細: residues 66-154 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 10 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: WIBG, Mago-binding |
-分子 #6: Btz
| 分子 | 名称: Btz / タイプ: protein_or_peptide / ID: 6 / Name.synonym: Btz / 詳細: residues 136-286 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 17 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: Btz domain |
-分子 #7: eIF4AIII
| 分子 | 名称: eIF4AIII / タイプ: protein_or_peptide / ID: 7 / Name.synonym: eIF4AIII / 詳細: full length / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 45 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: Btz domain |
-分子 #8: (U)8 RNA
| 分子 | 名称: (U)8 RNA / タイプ: rna / ID: 8 / Name.synonym: RNA / 詳細: Oligo(U) 8nt / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 3 KDa |
| 配列 | 文字列: UUUUUUUU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.175 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM K-phosphate,150 mM NaCl, 3 mM MgCl2, 20% sucrose, 0.1% glutaraldehyde |
| グリッド | 詳細: Quantifoil grids (R2/2) with thin carbon film on top |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK III 手法: Manual Application (3.5 microliters) Humidifier Off During Process Blot offset: -2 mm Blot Total: 2 Blot Time: 2 s Wait Time: 30 s Drain Time: 1 s |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 温度 | 最低: 83 K / 最高: 100 K / 平均: 91.5 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected calculating the Fourier Transform of the CCD frames. Phase flipping |
| 特殊光学系 | エネルギーフィルター - 名称: FEI エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 10.0 eV |
| 日付 | 2010年10月20日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 平均電子線量: 15 e/Å2 / 詳細: 0.22 nm per pixel, final sampling / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 69494 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.112 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric. 626 cryo-holder (Gatan Inc., Warrendale, PA, USA) 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each CCD Frame using BSOFT |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN, XMIPP, SPIDER, BSOFT 詳細: Initial reference obtained by random conical tilt of negative-stained images 使用した粒子像数: 85000 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: R-factor |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: R-factor |
-原子モデル構築 3
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: R-factor |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)