+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20265 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of voltage-gated sodium channel NavAb N49K/L109A/M116V/G94C/Q150C disulfide crosslinked mutant in the resting state | |||||||||||||||
 マップデータ マップデータ | Cryo-EM map of disulfide crosslinked MBP-NavAb N49K/L109A/M116V/G9C/Q150C in the resting state | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Ion channel / ion transport protein / MEMBRANE PROTEIN / metal transport | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報voltage-gated sodium channel complex / voltage-gated sodium channel activity / detection of maltose stimulus / maltose transport complex / carbohydrate transport / carbohydrate transmembrane transporter activity / maltose binding / maltose transport / maltodextrin transmembrane transport / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing ...voltage-gated sodium channel complex / voltage-gated sodium channel activity / detection of maltose stimulus / maltose transport complex / carbohydrate transport / carbohydrate transmembrane transporter activity / maltose binding / maltose transport / maltodextrin transmembrane transport / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / ATP-binding cassette (ABC) transporter complex / cell chemotaxis / outer membrane-bounded periplasmic space / periplasmic space / DNA damage response / metal ion binding / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Arcobacter butzleri RM4018 (バクテリア) / Arcobacter butzleri RM4018 (バクテリア) /  Arcobacter butzleri (strain RM4018) (バクテリア) Arcobacter butzleri (strain RM4018) (バクテリア) | |||||||||||||||
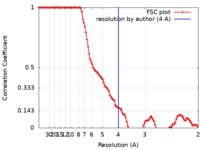
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Wisedchaisri G / Tonggu L | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2019 ジャーナル: Cell / 年: 2019タイトル: Resting-State Structure and Gating Mechanism of a Voltage-Gated Sodium Channel. 著者: Goragot Wisedchaisri / Lige Tonggu / Eedann McCord / Tamer M Gamal El-Din / Liguo Wang / Ning Zheng / William A Catterall /  要旨: Voltage-gated sodium (Na) channels initiate action potentials in nerve, muscle, and other electrically excitable cells. The structural basis of voltage gating is uncertain because the resting state ...Voltage-gated sodium (Na) channels initiate action potentials in nerve, muscle, and other electrically excitable cells. The structural basis of voltage gating is uncertain because the resting state exists only at deeply negative membrane potentials. To stabilize the resting conformation, we inserted voltage-shifting mutations and introduced a disulfide crosslink in the VS of the ancestral bacterial sodium channel NaAb. Here, we present a cryo-EM structure of the resting state and a complete voltage-dependent gating mechanism. The S4 segment of the VS is drawn intracellularly, with three gating charges passing through the transmembrane electric field. This movement forms an elbow connecting S4 to the S4-S5 linker, tightens the collar around the S6 activation gate, and prevents its opening. Our structure supports the classical "sliding helix" mechanism of voltage sensing and provides a complete gating mechanism for voltage sensor function, pore opening, and activation-gate closure based on high-resolution structures of a single sodium channel protein. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20265.map.gz emd_20265.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20265-v30.xml emd-20265-v30.xml emd-20265.xml emd-20265.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20265_fsc.xml emd_20265_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20265.png emd_20265.png | 162.1 KB | ||
| Filedesc metadata |  emd-20265.cif.gz emd-20265.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20265 http://ftp.pdbj.org/pub/emdb/structures/EMD-20265 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20265 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20265 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20265_validation.pdf.gz emd_20265_validation.pdf.gz | 351.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20265_full_validation.pdf.gz emd_20265_full_validation.pdf.gz | 351.3 KB | 表示 | |
| XML形式データ |  emd_20265_validation.xml.gz emd_20265_validation.xml.gz | 12.1 KB | 表示 | |
| CIF形式データ |  emd_20265_validation.cif.gz emd_20265_validation.cif.gz | 16 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20265 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20265 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20265 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20265 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20265.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20265.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of disulfide crosslinked MBP-NavAb N49K/L109A/M116V/G9C/Q150C in the resting state | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.056 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fusion of maltose-binding protein and voltage-gated sodium channe...
| 全体 | 名称: Fusion of maltose-binding protein and voltage-gated sodium channel NavAb in the resting state |
|---|---|
| 要素 |
|
-超分子 #1: Fusion of maltose-binding protein and voltage-gated sodium channe...
| 超分子 | 名称: Fusion of maltose-binding protein and voltage-gated sodium channel NavAb in the resting state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Arcobacter butzleri RM4018 (バクテリア) Arcobacter butzleri RM4018 (バクテリア) |
| 分子量 | 理論値: 300 KDa |
-分子 #1: Fusion of Maltose-binding protein and voltage-gated sodium channe...
| 分子 | 名称: Fusion of Maltose-binding protein and voltage-gated sodium channel NavAb タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Arcobacter butzleri (strain RM4018) (バクテリア) Arcobacter butzleri (strain RM4018) (バクテリア)株: RM4018 |
| 分子量 | 理論値: 74.072797 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MKIKTGARIL ALSALTTMMF SASALAKIEE GKLVIWINGD KGYNGLAEVG KKFEKDTGIK VTVEHPDKLE EKFPQVAATG DGPDIIFWA HDRFGGYAQS GLLAEITPDK AFQDKLYPFT WDAVRYNGKL IAYPIAVEAL SLIYNKDLLP NPPKTWEEIP A LDKELKAK ...文字列: MKIKTGARIL ALSALTTMMF SASALAKIEE GKLVIWINGD KGYNGLAEVG KKFEKDTGIK VTVEHPDKLE EKFPQVAATG DGPDIIFWA HDRFGGYAQS GLLAEITPDK AFQDKLYPFT WDAVRYNGKL IAYPIAVEAL SLIYNKDLLP NPPKTWEEIP A LDKELKAK GKSALMFNLQ EPYFTWPLIA ADGGYAFKYE NGKYDIKDVG VDNAGAKAGL TFLVDLIKNK HMNADTDYSI AE AAFNKGE TAMTINGPWA WSNIDTSKVN YGVTVLPTFK GQPSKPFVGV LSAGINAASP NKELAKEFLE NYLLTDEGLE AVN KDKPLG AVALKSYEEE LAKDPRIAAT MENAQKGEIM PNIPQMSAFW YAVRTAVINA ASGRQTVDEA LKDAQTNAMY LAIT NIVES SFFTKFIIYL IVLNGITMGL ETSKTFMQSF GVYTTLFKQI VITIFTIEII LRIYVHRISF FKDPWSLFDF FVVAI SLVP TSSCFEILRV LRVLRLFRAV TAVPQVRKIV SALISVIPGM LSVIALMTLF FYIFAIMATC LFGERFPEWF GTLGES FYT LFQVMTLESW SMGIVRPLME VYPYAWVFFI PFIFVVTFVM INLVVAIIVD AMAILNQKEE QHIIDEVQSH EDNINNE II KLREEIVELK ELIKTSLKN UniProtKB: Maltodextrin-binding protein, Ion transport protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.04 kPa | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 40 % / チャンバー内温度: 298 K / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 90.8 K / 最高: 103.5 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-43 / 撮影したグリッド数: 9 / 実像数: 5000 / 平均露光時間: 8.6 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 1001-1221 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-6p6w: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)