+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Fab4251-DS-SOSIP complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV / VIRAL PROTEIN | |||||||||
| 生物種 | HIV whole-genome vector AA1305#18 (その他) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
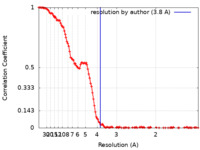
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Nortier P / Perez L | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Res Sq / 年: 2024 ジャーナル: Res Sq / 年: 2024タイトル: RAIN: a Machine Learning-based identification for HIV-1 bNAbs. 著者: Laurent Perez / Mathilde Foglierini /  要旨: Broadly neutralizing antibodies (bNAbs) are promising candidates for the treatment and prevention of HIV-1 infection. Despite their critical importance, automatic detection of HIV-1 bNAbs from immune ...Broadly neutralizing antibodies (bNAbs) are promising candidates for the treatment and prevention of HIV-1 infection. Despite their critical importance, automatic detection of HIV-1 bNAbs from immune repertoire is still lacking. Here, we developed a straightforward computational method for apid utomatic dentification of bAbs ) based on Machine Learning methods. In contrast to other approaches using one-hot encoding amino acid sequences or structural alignment for prediction, RAIN uses a combination of selected sequence-based features for accurate prediction of HIV-1 bNAbs. We demonstrate the performance of our approach on non-biased, experimentally obtained sequenced BCR repertoires from HIV-1 immune donors. RAIN processing leads to the successful identification of novel HIV-1 bNAbs targeting the CD4-binding site of the envelope glycoprotein. In addition, we validate the identified bNAbs using neutralization assay and we solve the structure of one of them in complex with the soluble native-like heterotrimeric envelope glycoprotein by single-particle cryo-electron microscopy (cryo-EM). Overall, we propose a method to facilitate and accelerate HIV-1 bNAbs discovery from non-selected immune repertoires. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19665.map.gz emd_19665.map.gz | 154.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19665-v30.xml emd-19665-v30.xml emd-19665.xml emd-19665.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19665_fsc.xml emd_19665_fsc.xml | 16.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19665.png emd_19665.png | 28.9 KB | ||
| Filedesc metadata |  emd-19665.cif.gz emd-19665.cif.gz | 6.9 KB | ||
| その他 |  emd_19665_half_map_1.map.gz emd_19665_half_map_1.map.gz emd_19665_half_map_2.map.gz emd_19665_half_map_2.map.gz | 151.8 MB 151.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19665 http://ftp.pdbj.org/pub/emdb/structures/EMD-19665 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19665 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19665 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19665_validation.pdf.gz emd_19665_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19665_full_validation.pdf.gz emd_19665_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_19665_validation.xml.gz emd_19665_validation.xml.gz | 18.1 KB | 表示 | |
| CIF形式データ |  emd_19665_validation.cif.gz emd_19665_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19665 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19665 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19665 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19665 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19665.map.gz / 形式: CCP4 / 大きさ: 198.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19665.map.gz / 形式: CCP4 / 大きさ: 198.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.73277 Å | ||||||||||||||||||||||||||||||||||||
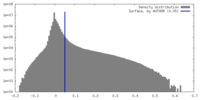
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_19665_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
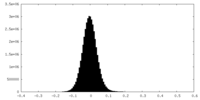
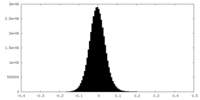
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19665_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
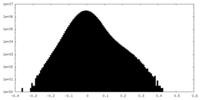
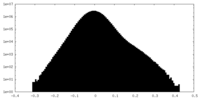
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Envelope glycoprotein Human immunodeficiency virus 1 with Fab
| 全体 | 名称: Envelope glycoprotein Human immunodeficiency virus 1 with Fab |
|---|---|
| 要素 |
|
-超分子 #1: Envelope glycoprotein Human immunodeficiency virus 1 with Fab
| 超分子 | 名称: Envelope glycoprotein Human immunodeficiency virus 1 with Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種: HIV whole-genome vector AA1305#18 (その他) |
| 分子量 | 理論値: 400 kDa/nm |
-分子 #1: variable heavy chain
| 分子 | 名称: variable heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.951986 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVHVMQSGDQ VKKPGASVQV SCTTSGSSFI EDSLHWIQQV PGQEPEWLGW VNPRHGAVNY SWKIRDRITM TRDISKSTMY VQMRGLQSD DTAMYYCAKS RRGANWALYW GWGTRITVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG ...文字列: QVHVMQSGDQ VKKPGASVQV SCTTSGSSFI EDSLHWIQQV PGQEPEWLGW VNPRHGAVNY SWKIRDRITM TRDISKSTMY VQMRGLQSD DTAMYYCAKS RRGANWALYW GWGTRITVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKKVE PK |
-分子 #2: variable light chain
| 分子 | 名称: variable light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.257809 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQLTQSPSY LAASVGDRVT ITCRESNDLN WYLQRPGKPP KLLISGASKL ERGVPSRFRG SSSGSLTISG LQPEDIGTYY CQVFEFFGG GTRVDIKRTV AAPSVFIFPP SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD S TYSLSSTL ...文字列: DIQLTQSPSY LAASVGDRVT ITCRESNDLN WYLQRPGKPP KLLISGASKL ERGVPSRFRG SSSGSLTISG LQPEDIGTYY CQVFEFFGG GTRVDIKRTV AAPSVFIFPP SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD S TYSLSSTL TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC |
-分子 #3: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 3 詳細: Envelope glycoprotein gp120|Human immunodeficiency virus 1 コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: HIV whole-genome vector AA1305#18 (その他) |
| 分子量 | 理論値: 49.558484 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKDAET TLFCASDHNV WATHACVPTD PNPQEIHLEN VTEEFNMWKN NMVEQMHTDI ISLWDQSLKP CVKLTPLCV TLQCTNVTNN ITDDMRGELK NCSFNMTTEL RDKKQKVYSL FYRLDVVQIK EYRLINCNTS ACTQACPKVS F EPIPIHYC ...文字列: AENLWVTVYY GVPVWKDAET TLFCASDHNV WATHACVPTD PNPQEIHLEN VTEEFNMWKN NMVEQMHTDI ISLWDQSLKP CVKLTPLCV TLQCTNVTNN ITDDMRGELK NCSFNMTTEL RDKKQKVYSL FYRLDVVQIK EYRLINCNTS ACTQACPKVS F EPIPIHYC APAGFAILKC KDKKFNGTGP CPSVSTVQCT HGIKPVVSTQ LLLNGSLAEE EVMIRSENIT NNAKNILVQF NT PVQINCT RPNNNTRKSI RIGPGQAFYA TGDIIGDIRQ AHCNVSKATW NETLGKVVKQ LRKHFGNNTI IRFANSSGGD LEV TTHSFN CGGEFFYCNT SGLFNSTWIS NSNDSITLPC RIKQIINMWQ RIGQCMYAPP IQGVIRCVSN ITGLILTRDG GSTN STTET FRPGGGDMRD NWRSELYKYK VVKIEPLGVA PTRCKR |
-分子 #4: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: HIV whole-genome vector AA1305#18 (その他) |
| 分子量 | 理論値: 14.561529 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSTVWGI KQLQARVLAV ERYLRDQQLL GIWGCSGKLI CCTNVPWNSS WSNRNLSEI WDNMTWLQWD KEISNYTQII YGLLEESQNQ QEKNEQDLLA L |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 17 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 6 |
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 4 / 実像数: 15163 / 平均電子線量: 39.89 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-8s2e: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)