+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human RNF213: focused refinement of ATPase domain | |||||||||
 マップデータ マップデータ | Unsharpened map of RNF213 ATPase domain after focused refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | E3 ubiquitin ligase / ANTIMICROBIAL PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Naydenova K / Randow F | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO Rep / 年: 2024 ジャーナル: EMBO Rep / 年: 2024タイトル: Recognition of phylogenetically diverse pathogens through enzymatically amplified recruitment of RNF213. 著者: Ana Crespillo-Casado / Prathyush Pothukuchi / Katerina Naydenova / Matthew C J Yip / Janet M Young / Jerome Boulanger / Vimisha Dharamdasani / Ceara Harper / Pierre-Mehdi Hammoudi / Elsje G ...著者: Ana Crespillo-Casado / Prathyush Pothukuchi / Katerina Naydenova / Matthew C J Yip / Janet M Young / Jerome Boulanger / Vimisha Dharamdasani / Ceara Harper / Pierre-Mehdi Hammoudi / Elsje G Otten / Keith Boyle / Mayuri Gogoi / Harmit S Malik / Felix Randow /   要旨: Innate immunity senses microbial ligands known as pathogen-associated molecular patterns (PAMPs). Except for nucleic acids, PAMPs are exceedingly taxa-specific, thus enabling pattern recognition ...Innate immunity senses microbial ligands known as pathogen-associated molecular patterns (PAMPs). Except for nucleic acids, PAMPs are exceedingly taxa-specific, thus enabling pattern recognition receptors to detect cognate pathogens while ignoring others. How the E3 ubiquitin ligase RNF213 can respond to phylogenetically distant pathogens, including Gram-negative Salmonella, Gram-positive Listeria, and eukaryotic Toxoplasma, remains unknown. Here we report that the evolutionary history of RNF213 is indicative of repeated adaptation to diverse pathogen target structures, especially in and around its newly identified CBM20 carbohydrate-binding domain, which we have resolved by cryo-EM. We find that RNF213 forms coats on phylogenetically distant pathogens. ATP hydrolysis by RNF213's dynein-like domain is essential for coat formation on all three pathogens studied as is RZ finger-mediated E3 ligase activity for bacteria. Coat formation is not diffusion-limited but instead relies on rate-limiting initiation events and subsequent cooperative incorporation of further RNF213 molecules. We conclude that RNF213 responds to evolutionarily distant pathogens through enzymatically amplified cooperative recruitment. #1:  ジャーナル: Biorxiv / 年: 2024 ジャーナル: Biorxiv / 年: 2024タイトル: Recognition of phylogenetically diverse pathogens through enzymatically amplified recruitment of RNF213 著者: Casado AC / Pothukuchi P / Naydenova K / Yip MCJ / Young JM / Boulanger J / Dharamdasani V / Harper C / Hammoudi PM / Otten EG / Boyle K / Gogoi M / Malik HS / Randow F | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19655.map.gz emd_19655.map.gz | 475.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19655-v30.xml emd-19655-v30.xml emd-19655.xml emd-19655.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19655_fsc.xml emd_19655_fsc.xml | 18.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19655.png emd_19655.png | 54.8 KB | ||
| マスクデータ |  emd_19655_msk_1.map emd_19655_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19655.cif.gz emd-19655.cif.gz | 4.9 KB | ||
| その他 |  emd_19655_half_map_1.map.gz emd_19655_half_map_1.map.gz emd_19655_half_map_2.map.gz emd_19655_half_map_2.map.gz | 413.9 MB 413.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19655 http://ftp.pdbj.org/pub/emdb/structures/EMD-19655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19655 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19655_validation.pdf.gz emd_19655_validation.pdf.gz | 994.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19655_full_validation.pdf.gz emd_19655_full_validation.pdf.gz | 994 KB | 表示 | |
| XML形式データ |  emd_19655_validation.xml.gz emd_19655_validation.xml.gz | 25.9 KB | 表示 | |
| CIF形式データ |  emd_19655_validation.cif.gz emd_19655_validation.cif.gz | 34.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19655 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19655 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19655.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19655.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map of RNF213 ATPase domain after focused refinement | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.921 Å | ||||||||||||||||||||||||||||||||||||
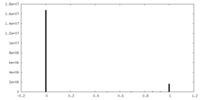
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19655_msk_1.map emd_19655_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
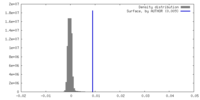
| 密度ヒストグラム |
-ハーフマップ: Half-map (2) of RNF213 ATPase domain focused refinement
| ファイル | emd_19655_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map (2) of RNF213 ATPase domain focused refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
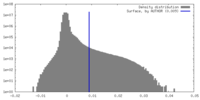
| 密度ヒストグラム |
-ハーフマップ: Half-map (1) of RNF213 ATPase domain focused refinement
| ファイル | emd_19655_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map (1) of RNF213 ATPase domain focused refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
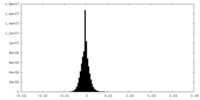
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human RNF213
| 全体 | 名称: Human RNF213 |
|---|---|
| 要素 |
|
-超分子 #1: Human RNF213
| 超分子 | 名称: Human RNF213 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: E3 ubiquitin ligase RNF213, human, N1045D natural variant |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 590 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 平均露光時間: 4.86 sec. / 平均電子線量: 29.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)