+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Procapsid of bacteriophage JBD30 computed in I4 symmetry | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bacteriophage / virion / procapsid / VIRUS | |||||||||
| 機能・相同性 | Bacteriophage Mu, GpT / Mu-like prophage major head subunit gpT / Bacteriophage Mu GpT domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) | |||||||||
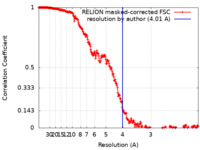
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.01 Å | |||||||||
 データ登録者 データ登録者 | Valentova L / Fuzik T / Plevka P | |||||||||
| 資金援助 | European Union,  チェコ, 2件 チェコ, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2024 ジャーナル: EMBO J / 年: 2024タイトル: Structure and replication of Pseudomonas aeruginosa phage JBD30. 著者: Lucie Valentová / Tibor Füzik / Jiří Nováček / Zuzana Hlavenková / Jakub Pospíšil / Pavel Plevka /  要旨: Bacteriophages are the most abundant biological entities on Earth, but our understanding of many aspects of their lifecycles is still incomplete. Here, we have structurally analysed the infection ...Bacteriophages are the most abundant biological entities on Earth, but our understanding of many aspects of their lifecycles is still incomplete. Here, we have structurally analysed the infection cycle of the siphophage Casadabanvirus JBD30. Using its baseplate, JBD30 attaches to Pseudomonas aeruginosa via the bacterial type IV pilus, whose subsequent retraction brings the phage to the bacterial cell surface. Cryo-electron microscopy structures of the baseplate-pilus complex show that the tripod of baseplate receptor-binding proteins attaches to the outer bacterial membrane. The tripod and baseplate then open to release three copies of the tape-measure protein, an event that is followed by DNA ejection. JBD30 major capsid proteins assemble into procapsids, which expand by 7% in diameter upon filling with phage dsDNA. The DNA-filled heads are finally joined with 180-nm-long tails, which bend easily because flexible loops mediate contacts between the successive discs of major tail proteins. It is likely that the structural features and replication mechanisms described here are conserved among siphophages that utilize the type IV pili for initial cell attachment. #1:  ジャーナル: Embo J. / 年: 2024 ジャーナル: Embo J. / 年: 2024タイトル: Structure and replication of Pseudomonas aeruginosa phage JBD30 著者: Valentova L / Plevka P / Fuzik T / Novacek J / Pospisil J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19285.map.gz emd_19285.map.gz | 1.3 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19285-v30.xml emd-19285-v30.xml emd-19285.xml emd-19285.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19285_fsc.xml emd_19285_fsc.xml | 25.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19285.png emd_19285.png | 257 KB | ||
| マスクデータ |  emd_19285_msk_1.map emd_19285_msk_1.map | 1.4 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19285.cif.gz emd-19285.cif.gz | 6.2 KB | ||
| その他 |  emd_19285_half_map_1.map.gz emd_19285_half_map_1.map.gz emd_19285_half_map_2.map.gz emd_19285_half_map_2.map.gz | 1.1 GB 1.1 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19285 http://ftp.pdbj.org/pub/emdb/structures/EMD-19285 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19285 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19285 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19285_validation.pdf.gz emd_19285_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19285_full_validation.pdf.gz emd_19285_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_19285_validation.xml.gz emd_19285_validation.xml.gz | 33.4 KB | 表示 | |
| CIF形式データ |  emd_19285_validation.cif.gz emd_19285_validation.cif.gz | 45.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19285 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19285 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19285 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19285 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8rkxMC  8rk3C  8rk4C  8rk5C  8rk6C  8rk7C  8rk8C  8rk9C  8rkaC  8rkbC  8rkcC  8rknC  8rkoC  8rqeC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19285.map.gz / 形式: CCP4 / 大きさ: 1.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19285.map.gz / 形式: CCP4 / 大きさ: 1.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.057 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19285_msk_1.map emd_19285_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19285_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19285_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pseudomonas phage JBD30
| 全体 | 名称:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Pseudomonas phage JBD30
| 超分子 | 名称: Pseudomonas phage JBD30 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Phage JBD30 was propagated in P. aeruginosa strain BAA-28 and purified using CsCl gradient. NCBI-ID: 1223260 / 生物種: Pseudomonas phage JBD30 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 13.971 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: JBD30 procapsid / 直径: 570.0 Å / T番号(三角分割数): 7 |
-分子 #1: Bacteriophage Mu GpT domain-containing protein
| 分子 | 名称: Bacteriophage Mu GpT domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
| 分子量 | 理論値: 33.492707 KDa |
| 配列 | 文字列: IITPALISAL KTSFQKHFQD ALATAPSTYL QVATVIPSTT ASNTYGWLGQ FPKLREWIGQ RVIKDMAAQG YQITNKLFES TVGVKRTDI EDDNLGVYGP LMQEMGRAAG AHPDELVFAL LKAGNANLCY DGQNFFDTDH PVYPNVDGTG TATTVSNLFA P AADPGAAW ...文字列: IITPALISAL KTSFQKHFQD ALATAPSTYL QVATVIPSTT ASNTYGWLGQ FPKLREWIGQ RVIKDMAAQG YQITNKLFES TVGVKRTDI EDDNLGVYGP LMQEMGRAAG AHPDELVFAL LKAGNANLCY DGQNFFDTDH PVYPNVDGTG TATTVSNLFA P AADPGAAW YLLDTSRSLK PLIYQERMKP SFTSMTKEDD EQVFMADEYR YGVRSRCNVG FGFWQLAAMS TEELNQVNFE KV YDAMRNQ KADGGRPLDI RPNLLVVPTT LRSKAKEVVG VQRLANGADN PNFELVQVLD TAWLN UniProtKB: Bacteriophage Mu GpT domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 10 mM MgSO4, 10 mM NaCl, 50 mM Tris pH 8 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: OTHER / 詳細: Gatan Solarus II | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: blotting force 0, blotting time 2 s, waiting time 15 s. | ||||||||||||
| 詳細 | phage titer 10^11 PFU |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 13769 / 平均露光時間: 7.5 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | de novo model building |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-8rkx: |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)