+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Thinner is not always better: Optimising cryo lamellae for subtomogram averaging | |||||||||
 マップデータ マップデータ | Ribosome from Dictyostelium discoideum, from in-situ data using cryo-ET and STA. "high-quality" particles, 23k particles. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | In-situ Dictyostelium discoideum ribosome / RIBOSOME | |||||||||
| 生物種 |  | |||||||||
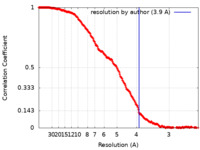
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Tuijtel MW / Cruz-Leon S / Kreysing JP / Welsch S / Hummer G / Beck M / Turonova B | |||||||||
| 資金援助 |  米国, 米国,  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: Thinner is not always better: Optimizing cryo-lamellae for subtomogram averaging. 著者: Maarten W Tuijtel / Sergio Cruz-León / Jan Philipp Kreysing / Sonja Welsch / Gerhard Hummer / Martin Beck / Beata Turoňová /  要旨: Cryo-electron tomography (cryo-ET) is a powerful method to elucidate subcellular architecture and to structurally analyze biomolecules in situ by subtomogram averaging, yet data quality critically ...Cryo-electron tomography (cryo-ET) is a powerful method to elucidate subcellular architecture and to structurally analyze biomolecules in situ by subtomogram averaging, yet data quality critically depends on specimen thickness. Cells that are too thick for transmission imaging can be thinned into lamellae by cryo-focused ion beam (cryo-FIB) milling. Despite being a crucial parameter directly affecting attainable resolution, optimal lamella thickness has not been systematically investigated nor the extent of structural damage caused by gallium ions used for FIB milling. We thus systematically determined how resolution is affected by these parameters. We find that ion-induced damage does not affect regions more than 30 nanometers from either lamella surface and that up to ~180-nanometer lamella thickness does not negatively affect resolution. This shows that there is no need to generate very thin lamellae and lamella thickness can be chosen such that it captures cellular features of interest, thereby opening cryo-ET also for studies of large complexes. #1:  ジャーナル: BiorXiv ジャーナル: BiorXivタイトル: Thinner is not always better: Optimising cryo lamellae for subtomogram averaging 著者: Tuijtel MW / Cruz-Leon S / Kreysing JP / Welsch S / Hummer G / Beck M / Turonova B | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19136.map.gz emd_19136.map.gz | 81 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19136-v30.xml emd-19136-v30.xml emd-19136.xml emd-19136.xml | 35.7 KB 35.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19136_fsc.xml emd_19136_fsc.xml | 20 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19136.png emd_19136.png | 94.8 KB | ||
| Filedesc metadata |  emd-19136.cif.gz emd-19136.cif.gz | 5 KB | ||
| その他 |  emd_19136_additional_1.map.gz emd_19136_additional_1.map.gz emd_19136_additional_2.map.gz emd_19136_additional_2.map.gz emd_19136_additional_3.map.gz emd_19136_additional_3.map.gz emd_19136_additional_4.map.gz emd_19136_additional_4.map.gz emd_19136_additional_5.map.gz emd_19136_additional_5.map.gz emd_19136_additional_6.map.gz emd_19136_additional_6.map.gz emd_19136_additional_7.map.gz emd_19136_additional_7.map.gz emd_19136_additional_8.map.gz emd_19136_additional_8.map.gz emd_19136_additional_9.map.gz emd_19136_additional_9.map.gz emd_19136_half_map_1.map.gz emd_19136_half_map_1.map.gz emd_19136_half_map_2.map.gz emd_19136_half_map_2.map.gz | 81 MB 81 MB 81 MB 80.9 MB 80.9 MB 80.9 MB 81.2 MB 81.2 MB 80.5 MB 81 MB 81 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19136 http://ftp.pdbj.org/pub/emdb/structures/EMD-19136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19136 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19136.map.gz / 形式: CCP4 / 大きさ: 87.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19136.map.gz / 形式: CCP4 / 大きさ: 87.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Ribosome from Dictyostelium discoideum, from in-situ data using cryo-ET and STA. "high-quality" particles, 23k particles. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.223 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+追加マップ: Ribosome from Dictyostelium discoideum, from in-situ data using...
+ハーフマップ: halfmap 1 "high-quality" particles, 23k particles.
+ハーフマップ: halfmap 2 "high-quality" particles, 23k particles.
- 試料の構成要素
試料の構成要素
-全体 : Ribosome
| 全体 | 名称: Ribosome |
|---|---|
| 要素 |
|
-超分子 #1: Ribosome
| 超分子 | 名称: Ribosome / タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Ribosome |
|---|---|
| 由来(天然) | 生物種:  細胞中の位置: cytoplasm |
| 分子量 | 理論値: 4 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 20 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 2 / 平均露光時間: 0.24 sec. / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 2.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X