[English] 日本語
 Yorodumi
Yorodumi- EMDB-18849: Influence of lipid bilayer on structure of acetylcholine receptor -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Influence of lipid bilayer on structure of acetylcholine receptor | |||||||||
 Map data Map data | Acetylcholine receptors in tubular vesicle; (-17,5) helical family | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Ion channel / MEMBRANE PROTEIN | |||||||||
| Biological species |  | |||||||||
| Method | helical reconstruction / cryo EM / Resolution: 8.2 Å | |||||||||
 Authors Authors | Unwin N | |||||||||
| Funding support |  United Kingdom, 1 items United Kingdom, 1 items
| |||||||||
 Citation Citation |  Journal: Proc Natl Acad Sci U S A / Year: 2024 Journal: Proc Natl Acad Sci U S A / Year: 2024Title: Influence of lipid bilayer on the structure of the muscle-type nicotinic acetylcholine receptor. Authors: Nigel Unwin /  Abstract: The muscle-type nicotinic acetylcholine receptor is a transmitter-gated ion channel residing in the plasma membrane of electrocytes and striated muscle cells. It is present predominantly at synaptic ...The muscle-type nicotinic acetylcholine receptor is a transmitter-gated ion channel residing in the plasma membrane of electrocytes and striated muscle cells. It is present predominantly at synaptic junctions, where it effects rapid depolarization of the postsynaptic membrane in response to acetylcholine released into the synaptic cleft. Previously, cryo-EM of intact membrane from revealed that the lipid bilayer surrounding the junctional receptor has a uniquely asymmetric and ordered structure, due to a high concentration of cholesterol. It is now shown that this special lipid environment influences the transmembrane (TM) folding of the protein. All five submembrane MX helices of the membrane-intact junctional receptor align parallel to the surface of the cholesterol-ordered lipids in the inner leaflet of the bilayer; also, the TM helices in the outer leaflet are splayed apart. However in the structure obtained from the same protein after extraction and incorporation in nanodiscs, the MX helices do not align to a planar surface, and the TM helices arrange compactly in the outer leaflet. Realignment of the MX helices of the nanodisc-solved structure to a planar surface converts their adjoining TM helices into an obligatory splayed configuration, characteristic of the junctional receptor. Thus, the form of the receptor sustained by the special lipid environment of the synaptic junction is the one that mediates fast synaptic transmission; whereas, the nanodisc-embedded protein may be like the extrajunctional form, existing in a disordered lipid environment. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_18849.map.gz emd_18849.map.gz | 502.1 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-18849-v30.xml emd-18849-v30.xml emd-18849.xml emd-18849.xml | 18.4 KB 18.4 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_18849.png emd_18849.png | 138.8 KB | ||
| Filedesc metadata |  emd-18849.cif.gz emd-18849.cif.gz | 4.9 KB | ||
| Others |  emd_18849_half_map_1.map.gz emd_18849_half_map_1.map.gz emd_18849_half_map_2.map.gz emd_18849_half_map_2.map.gz | 960.9 MB 962.7 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18849 http://ftp.pdbj.org/pub/emdb/structures/EMD-18849 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18849 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18849 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_18849.map.gz / Format: CCP4 / Size: 1.9 GB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_18849.map.gz / Format: CCP4 / Size: 1.9 GB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Acetylcholine receptors in tubular vesicle; (-17,5) helical family | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: Acetylcholine receptors in tubular vesicle; (-17,5) helical family
| File | emd_18849_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Acetylcholine receptors in tubular vesicle; (-17,5) helical family | ||||||||||||
| Projections & Slices |
| ||||||||||||
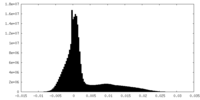
| Density Histograms |
-Half map: Acetylcholine receptors in tubular vesicle; (-17,5) helical family
| File | emd_18849_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Acetylcholine receptors in tubular vesicle; (-17,5) helical family | ||||||||||||
| Projections & Slices |
| ||||||||||||
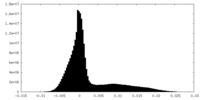
| Density Histograms |
- Sample components
Sample components
-Entire : Muscle-type nicotinic acetylcholine receptor in intact synaptic m...
| Entire | Name: Muscle-type nicotinic acetylcholine receptor in intact synaptic membrane |
|---|---|
| Components |
|
-Supramolecule #1: Muscle-type nicotinic acetylcholine receptor in intact synaptic m...
| Supramolecule | Name: Muscle-type nicotinic acetylcholine receptor in intact synaptic membrane type: organelle_or_cellular_component / ID: 1 / Parent: 0 / Macromolecule list: #1-#4 Details: Postsynaptic membranes were isolated from fresh electric organ and incubated in low salt buffer to form ordered arrays of acetylcholine receptors in tubular vesicles |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 290 KDa |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | helical reconstruction |
| Aggregation state | helical array |
- Sample preparation
Sample preparation
| Buffer | pH: 7 Component:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 95 % / Chamber temperature: 283 K / Instrument: HOMEMADE PLUNGER | |||||||||
| Details | Specimen comprises tubular vesicles which are imaged in thin ice over holes in the support film |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Temperature | Min: 70.0 K / Max: 70.0 K |
| Details | All images taken manually: by searching at low magnification for long straight and narrow tubes, then recording in integrating mode |
| Image recording | Film or detector model: FEI FALCON III (4k x 4k) / Detector mode: INTEGRATING / Number grids imaged: 200 / Number real images: 4045 / Average exposure time: 2.0 sec. / Average electron dose: 40.0 e/Å2 Details: Images were collected in integrating mode, 2 seconds exposure |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Calibrated defocus max: 2.8000000000000003 µm / Calibrated defocus min: 1.2 µm / Calibrated magnification: 104478 / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.8000000000000003 µm / Nominal defocus min: 1.2 µm / Nominal magnification: 59000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Initial model |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Details | Refinement parameters were chosen to minimise changes to the original secondary structure | ||||||||||||||||||
| Refinement | Space: REAL / Protocol: FLEXIBLE FIT / Overall B value: 350 |
 Movie
Movie Controller
Controller






































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)