+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of C. thermophilum RNA exosome core | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | nuclease / RNA degradation / RNA metabolism / RNA binding / RNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報CUT catabolic process / cytoplasmic exosome (RNase complex) / U1 snRNA 3'-end processing / U5 snRNA 3'-end processing / TRAMP-dependent tRNA surveillance pathway / U4 snRNA 3'-end processing / nuclear polyadenylation-dependent rRNA catabolic process / poly(A)-dependent snoRNA 3'-end processing / nuclear exosome (RNase complex) / exonucleolytic trimming to generate mature 3'-end of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) ...CUT catabolic process / cytoplasmic exosome (RNase complex) / U1 snRNA 3'-end processing / U5 snRNA 3'-end processing / TRAMP-dependent tRNA surveillance pathway / U4 snRNA 3'-end processing / nuclear polyadenylation-dependent rRNA catabolic process / poly(A)-dependent snoRNA 3'-end processing / nuclear exosome (RNase complex) / exonucleolytic trimming to generate mature 3'-end of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / nuclear mRNA surveillance / rRNA catabolic process / mRNA 3'-UTR AU-rich region binding / RNA processing / rRNA processing / nucleolus / RNA binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) | |||||||||
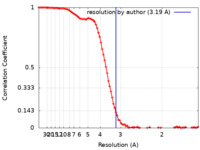
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.19 Å | |||||||||
 データ登録者 データ登録者 | Lazzaretti D / Liebau J / Pilsl M / Sprangers R | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: 4D structural biology-quantitative dynamics in the eukaryotic RNA exosome complex. 著者: Jobst Liebau / Daniela Lazzaretti / Torben Fürtges / Anna Bichler / Michael Pilsl / Till Rudack / Remco Sprangers /  要旨: Molecular machines play pivotal roles in all biological processes. Most structural methods, however, are unable to directly probe molecular motions. Here, we demonstrate that dedicated NMR ...Molecular machines play pivotal roles in all biological processes. Most structural methods, however, are unable to directly probe molecular motions. Here, we demonstrate that dedicated NMR experiments can provide quantitative insights into functionally important dynamic regions in very large asymmetric protein complexes. We establish this for the 410 kDa eukaryotic RNA exosome complex that contains ten distinct protein chains. Methyl-group and fluorine NMR experiments reveal site-specific interactions among subunits and with an RNA substrate. Furthermore, we extract quantitative insights into conformational changes within the complex in response to substrate and subunit binding for regions that are invisible in static cryo-EM and crystal structures. In particular, we identify a flexible plug region that can block an aberrant route for RNA towards the active site. Based on molecular dynamics simulations and NMR data, we provide a model that shows how the flexible plug is structured in the open and closed conformations. Our work thus demonstrates that a combination of state-of-the-art structural biology methods can provide quantitative insights into large molecular machines that go significantly beyond the well-resolved and static images of biomolecular complexes, thereby adding the time domain to structural biology. #1:  ジャーナル: Biorxiv / 年: 2024 ジャーナル: Biorxiv / 年: 2024タイトル: Beyond static structures: quantitative dynamics in the eukaryotic RNA exosome complex 著者: Liebau J / Lazzaretti D / Bichler A / Pilsl M / Sprangers R | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18825.map.gz emd_18825.map.gz | 49.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18825-v30.xml emd-18825-v30.xml emd-18825.xml emd-18825.xml | 35.6 KB 35.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18825_fsc.xml emd_18825_fsc.xml | 11.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18825.png emd_18825.png | 92.5 KB | ||
| Filedesc metadata |  emd-18825.cif.gz emd-18825.cif.gz | 9.3 KB | ||
| その他 |  emd_18825_additional_1.map.gz emd_18825_additional_1.map.gz emd_18825_half_map_1.map.gz emd_18825_half_map_1.map.gz emd_18825_half_map_2.map.gz emd_18825_half_map_2.map.gz | 56.6 MB 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18825 http://ftp.pdbj.org/pub/emdb/structures/EMD-18825 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18825 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18825 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18825_validation.pdf.gz emd_18825_validation.pdf.gz | 921.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18825_full_validation.pdf.gz emd_18825_full_validation.pdf.gz | 921.5 KB | 表示 | |
| XML形式データ |  emd_18825_validation.xml.gz emd_18825_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_18825_validation.cif.gz emd_18825_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18825 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18825 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18825 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18825 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8r1oMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18825.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18825.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.772 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Post-processed map (deepEMhancer)
| ファイル | emd_18825_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map (deepEMhancer) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18825_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18825_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : RNA exosome core (Exo9)
+超分子 #1: RNA exosome core (Exo9)
+分子 #1: Rrp45
+分子 #2: Exoribonuclease phosphorolytic domain-containing protein
+分子 #3: Exoribonuclease-like protein
+分子 #4: Exoribonuclease phosphorolytic domain-containing protein
+分子 #5: Exoribonuclease phosphorolytic domain-containing protein
+分子 #6: Exosome complex component MTR3
+分子 #7: Ribosomal RNA-processing protein 40
+分子 #8: Putative exosome complex protein
+分子 #9: Putative exosome 3'->5 protein
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.53 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20 mM Na-Phosphate buffer pH 7.5, 300 mM NaCl, 1 mM DTT | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 200 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa 詳細: Grids were glow discharged twice at 15 mA, 0.39 mBar, for 100 seconds each time | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Sample volume 3 ul Wait time 5 s, Blot time 5 s, Delay time 0 s Force 12. | |||||||||||||||
| 詳細 | Sample frozen directly after S200 size exclusion column |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 200 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 6579 / 平均露光時間: 6.5 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)